TCGA数据处理流程中各种组织机构介绍
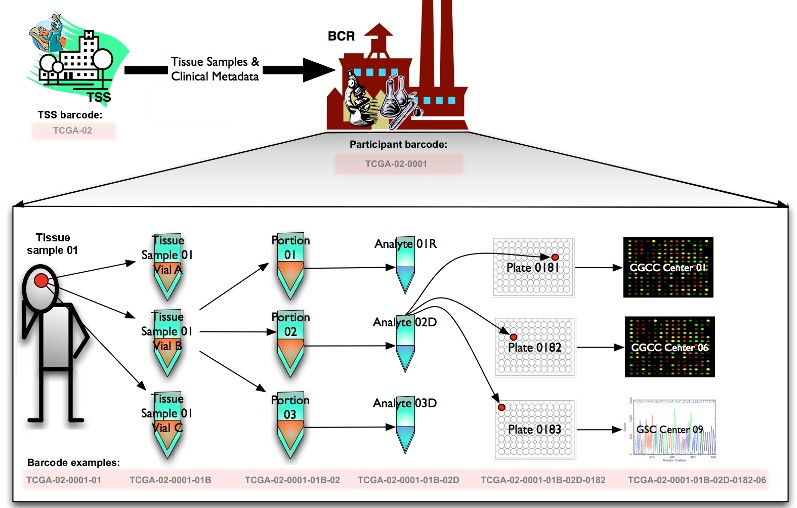
图中总结了TCGA的样品到数据处理流程:
1.组织样本及其临床数据是由Tissue Source Sites(TSS)组织来源点收集的,然后送交给Biospecimen Core Resources(BCRs)生物标本核心资源。
2.BCRs提交临床数据和元数据到Data Coordinating Center(DCC)数据整理中心,并把分析物送交给Genome Characterization Center(GCCs)基因组鉴定中心和Sequencing Center(GSCs)测序中心,在这里生成突变信号并把信号提交到DCC。
3.GSCs同样也提交跟踪文件、序列和比对图到Cancer Genomics Hub(CGHub)癌症基因组中心。
4.被提交到DCC和CGHub的数据可供研究团体和Genome Data Analysis Centers(GDACs)基因组数据分析中西使用。
5.分析渠道和GDACs产出的数据结果通过DCC对研究团体提供服务。
下表提供了对来自TCGA不同中心和小组的快速总览,要想对特定的小组或中心获得更多的信息,请单击相应的标签:
中心/小组 | 描述 |
TSS | 组织来源点,收集样本(组织,细胞,血液)和临床元数据,然后把这些数据和样本送到BCR。每一个组织来源点都有一个ID来识别。 |
BCR | 生物标本核心资源,是TCGA的样本中心,在这里样本及其参与者的临床信息被小心地分类,处理,质量检验和存储。分析物在BCR被等分并且分配等分条形码,之后送到其它中心。 BCR缩写对照表:https://gdc.cancer.gov/resources-tcga-users/tcga-code-tables/bcr-batch-codes |
GCC | 基因组鉴定中心,在这里用高通量技术来分析癌症基因组的改变。鉴定出来的基因组的改变被GSCs用来进一步的研究。GCCs把在数据档案中的鉴定实验的实验结果文件转移到DCC。 |
GSC | 基因组测序中心,在这里利用高通量方法去识别各种癌症的基因组序列中的改变。GSCs对分析物(由BCRs提供)进行测序并且分析假定的体细胞和生殖细胞的突变。测序结果被送到癌症基因组中心,突变结果被送到被送到DCC。 |
DCC | 数据整理中心,是提供TCGA数据的核心。DCC对数据格式进行标准化并且验证提交的数据。 DCC接受并且验证从BCRs,GCCs和GSCs获得的数据,之后研究团体才能通过TCGA门户网站的应用程序对数据进行使用。 |
GDAC | 基因组数据分析中心,这里给研究团体提供了新颖的信息学工具和TCGA数据的分析结果,DCC不接受通过自动验证和展开系统的任何GDAC的数据提交。GDAC目前通过受控访问临时上传分析数据到DCC。 |
CGHub | 癌症基因组中心,一个安全的数据库,用来存储,分类和访问TCGA及其相关项目的的癌症基因组序列,比对和突变信息。在SAIC-Frederick分包合同之下,CGHub由加州大学圣克鲁斯分校(UCSC)管理。GSCs上传跟踪文件,短信号序列和BAM文件到CGHub。 |
Project Team | 协调TCGA的项目团队,由NCI和NHGRI中的成员组成。 |
延伸阅读
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、biolinux搭建生物信息分析环境、linux命令处理生物大数据、perl入门到精通、perl语言高级、R语言画图、R语言快速入门与提高、python语言入门到精通
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、 GEO芯片数据不同平台标准化 、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代fastq测序数据解读、
9.全部课程可点击:组学大讲堂视频课程
- 发表于 2019-10-23 17:51
- 阅读 ( 6886 )
- 分类:TCGA
你可能感兴趣的文章
相关问题
- 请教一下TCGA的蛋白数据怎么理解,它是有正负值的,这个正负值代表什么意思,为什么蛋白表达会有负值?我只想给它分成高表达,低表达和表达缺失组应该怎么处理呀? 0 回答
- TCGA下载 docker报错 0 回答
- TCGA差异分析如何根据临床信息分组? 1 回答
- TCGA用R数据下载错误 1 回答
- 老师,想请教一下TCGA基因表达数据的问题,我从xena.ucsc网页上下载了基因表达数据TCGA-CESC.htseq_counts.tsv;然后发现该数据中只有Ensembl格式的基因ID ,没有SYMBOL格式的。所以接下来进行基因ID格式转换,却发现同一个SYMBOL ID对应的多个Ensembl格式的ID,想问下老师,这种情况该怎么处理?同一个SYMBOL ID所对应的多个Ensembl格式ID的基因表达数据应该留下哪一个? 1 回答
- TCGA的RNA-seq count annotation过滤,有些不知道是否需要删除 0 回答
