BSA分析之MutMap分析原理详解!
BSA分析原理
BSA(分离体分组混合分析法或混合分组分析法,又称 集团分离分析法,Bulked Segregant Analysis)分析法首次由Michlmore等提出并成功地在莴苣中筛选出与目的基因相连锁的标记。该方法首先从一对具有目标基因的表型差异的亲本所产生的任何一种分离群体中,根据目标基因的表型分别选取一定数量的植株,构成 2个亚群或集团。将每群的 DNA等量混合,形成两个相对性状 的“基因池”(gene pool),然后用合适的分子标记对两个基因池进行分析,在两群间表现多态性的分子标记遗传上与目标性状基因座位相连锁。在获得了与目标基因相连锁的分子标记以后,可以利用某一作图群体进行分析以便进一步检测所得分子标记与目标性状基因的连锁程度,以及其在某已知分子图谱中或染色体上的位置,这样才能完成真正意义上的对基因的标记定位。由于建池时使用了特定的分离群体,并且在分组时仅对目标性状进行选择,这样可以保证其他性状的遗传背景基本相同,两个基因池之间理论上就应主要在目标基因区段存在差异,因此两基因池又被称为近等基因池,这就排除了环境及人为因素的影响,使研究结果更为准确可靠。BSA法克服了很多作物难以得到近等基因系的限制,并且比近等基因系法省时省力,是一种非常实用的基因标记定位的方法,应用非常广泛。与BSA相结合用于基因定位的分子标记有多种,常用的分子标记有 RFLP(限制性片段长度多态性,Restriction Fragment Length Polymorphism),RAPD(随机扩增多态性 DNA,Random Amplified Polymorphism DNA),AFLP (扩增片段长度多态性,Amplified Fragment Length Polymorphism),SSR(简单重复序列,Simple Sequence Repeats)SSR及SNP等。
MutMap分析原理
在传统的图位克隆中,我们一般先利用BSA原理进行粗定位,寻找和突变表型连锁的marker,再在附近设计新的marker,利用作图群体进行精细定位,一步步缩小和突变表型连锁的染色体区段,直到鉴定出突变基因。MutMap的原理和图位克隆本质上是一样的,只不过由于高通量测序技术的出现,我们常规使用的marker换成了SNP,把通过PCR和酶切进行多态性鉴定,换成了用重测序的方法直接对SNP的多态性进行分析。说到底,MutMap及类似的方法都是利用了BSA的原理。下面结合图片讲解其分析原理:
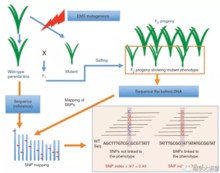
MutMap适合对EMS诱变的隐性突变基因进行分析。通过EMS诱变和自交得到纯合突变体后,将突变体和其亲本回交得到F1,F1自交得到的F2后代会出现表型的分离,得到野生型表型群体和突变表型群体(耐盐表型)。对这两个群体的DNA分别进行等量混合,得到野生型DNA混池和突变体DNA混池。将两个混池分别进行DNA测序,得到野生型和突变型混池测序变异SNP信息;突变为隐性,根据遗传学定律,在F2群体中,与耐盐表型不相关的突变会随机的分布在F2群体个体中,因此,与目标性状不相干的SNP会以:野生型类型:突变体类型=1:1的比例进行分离,而导致突变体表型的SNP,受到了人为选择,在突变体混池中所有的个体都是是纯合的。为了方便分析,作者定义了一个参数SNP-index,突变体类型的SNP所占的比例(也就是上图中右下角图示)。那么在突变位点,SNP-index为1,越往两侧,SNP-index越小,并最终接近于0.5。对SNP-index进行滑窗作图后,就会出现一个峰,该处就是连锁区域。在附近进行候选基因的筛选和排查,可以比较容易找到突变基因。
如何确定突变碱基
作者利用重测序的方法,对野生型品种(Hitomebore)进行重测序,将read比对到参考基因组日本晴(Nipponbare )上,将Hitomebore和Nipponbare 之间的差异进行替换,得到Hitomebore的参考基因组,如果我们的突变型测序结果比对到Hitomebore参考基因组上,与参考基因组不同的碱基即为突变碱基。以下是作者原文:reference sequence was constructed by replacing nucleotides in Nipponbare with those of Hitomebore at the 124,968 SNP positions identified between the two cultivars by alignment of 12.25 Gb of Illumina Hitomebore short reads to the Nipponbare reference genome
MutMap适用条件:
1. 一般为隐性突变,最好是EMS诱导的点突变;2. 必须测野生型基因组;3. 必须有回交过程:纯合之后再与野生型回交,再自交得到F2;参考文献:https://www.nature.com/articles/nbt.3188
好了,今天MutMap先介绍到这里,下一期将给大家介绍MutMap+,敬请期待!
基因组重测序数据分析视频课程:
- 发表于 2020-06-08 13:32
- 阅读 ( 8958 )
- 分类:重测序
你可能感兴趣的文章
- BSA分析中MutMap 分析:95% 99%阈值置信区间是如何计算的 4593 浏览
- BSA分析中QTL-seq 分析:95% 99%阈值置信区间是如何计算的 5456 浏览
- BSA分析算法中的ED算法和SNP-index有什么区别? 19275 浏览
- 混池(BSA)定位必看的10个知识点! 26664 浏览
- MutMap+和mutmap分析差异(BSA) 14141 浏览
相关问题
- 请问如何合并VCF文件中同一样本的生物学重复信息 1 回答
- 老师您好,我用mutmap的方法分析了f2群体,有三个峰,有个一个峰在99阈值之上,但是做基因分分型时 要么是突变体基因型要么是野生型基因型,而且区间前半段设计不出引物 0 回答
- 基因变异的VCF文件除了染色体部分外的其他变异数据如何过滤? 1 回答
- 老师,我想问一下BSA分析实操课程中qtlplot这个命令是一个脚本吗 qtlplot -v qtlseq.RS-blast.clean.vcf.gz -r HitomeboreWT --bulk1ID S-bulk --bulk2ID R-bulk --N-bulk1 20 --N-bulk2 20 -m 0.3 --out qtlseq_SR-blast 1 回答
- 老师好,请问课程bsa分析中,mutmap能同时输入两个子代混池吗? 1 回答
- mutmap方法为什么必须要有回交过程? 1 回答

