PlantPAN数据库
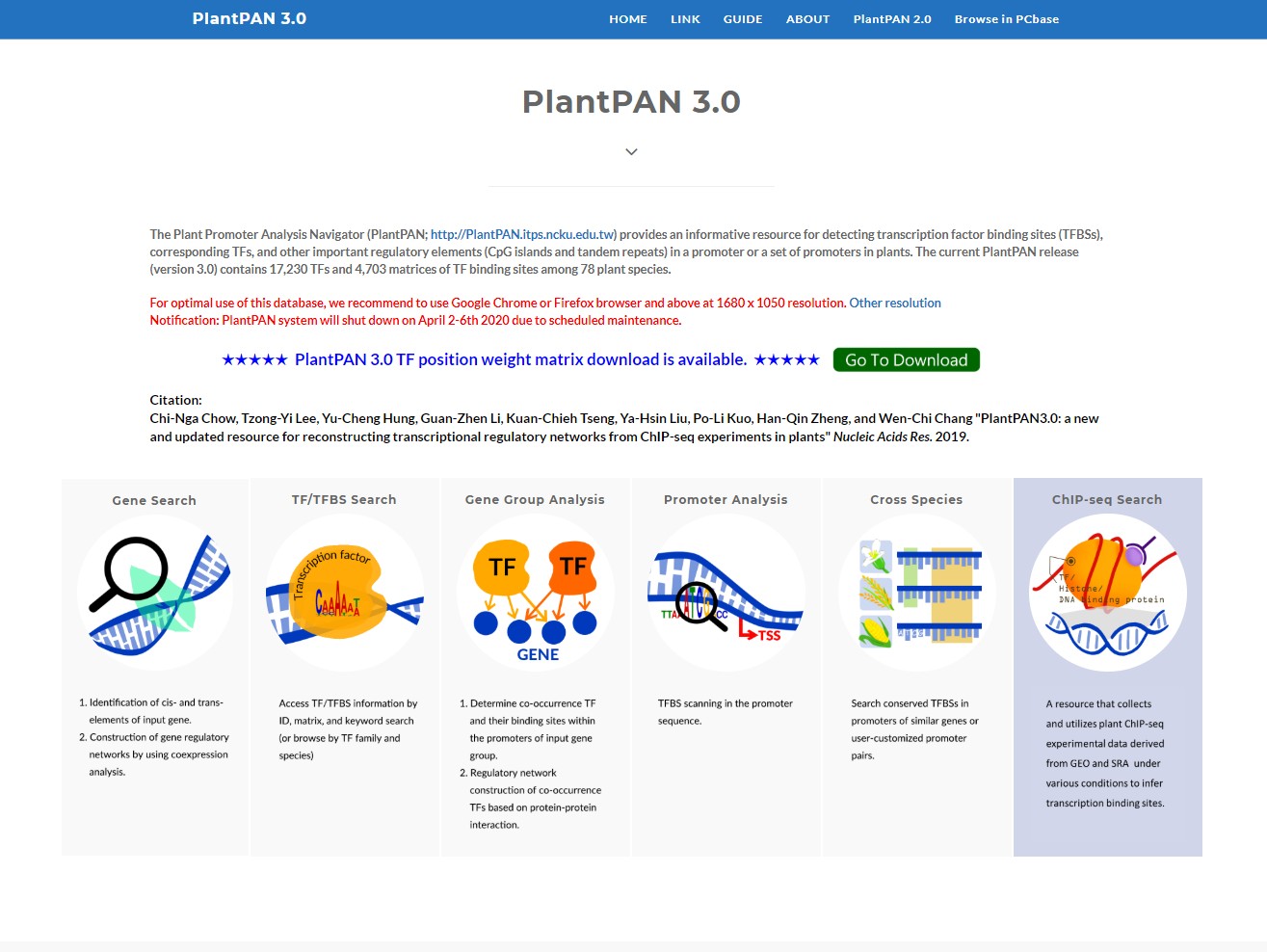
植物启动子分析浏览器(PlantPAN; http://plantpan.itps.ncku.edu.tw)是针对植物基因预测调节原件和重建转录调节网络的有效资源。
PlantPAN3.0 的首页,包含六个模块,分别是基因搜索、转录因子/转录因子结合位点搜索、基因集分析、启动子分析、跨物种和 ChIP-seq 搜索。
启动子分析
点击Promoter analysis,进入的页面为单个基因启动子序列的分析页面,还可选择多个基因的启动子序列分析以及在PCBase中分析
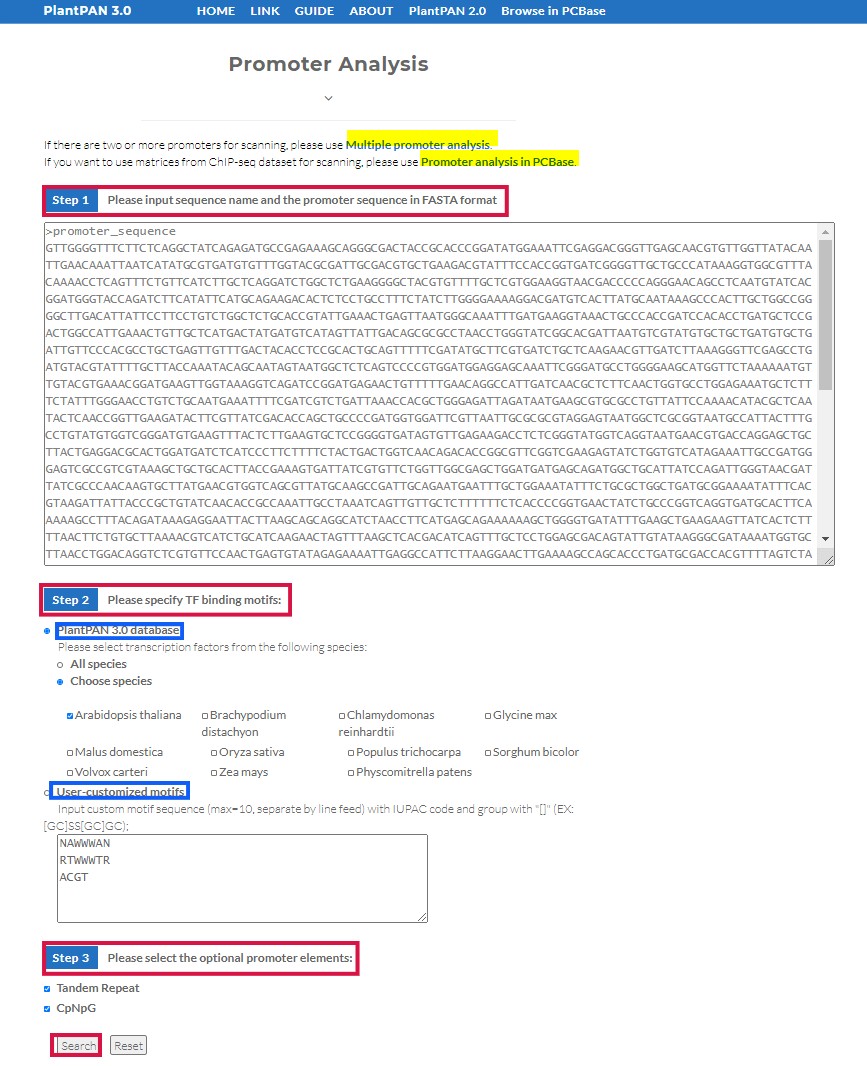
步骤一:输入单个基因的启动子序列
步骤二:有两个选择:1)选择在PlantPAN中使用所有物种的启动子序列或者是自己勾选部分物种启动子序列;2)选择自己感兴趣的motif序列
步骤三:选择启动子其他元件
最后点击search即可
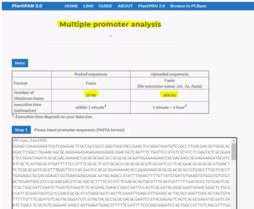
多个基因的启动子序列分析
1. 选择输入或上传要分析的FASTA文件。对于输入文件的大小以及上传文件的大小都有限制,分别为20kb和600kb。等待分析结果的时间与文件大小有关,文件越大,时间越长,最长等待时间为1小时。2. 选项同单个基因启动子分析3. 输入邮箱,分析完成后会结果直接发送到邮箱里
分析结果页面
1. 文件会用显示的符号及高亮标注Tandem Repeat 和 CPGisland(显示如下),可点击Download 下载分析结果

2. 分析结果详细如下:分为匹配搜索结果,CPG岛,串联重复,以及可视化选项。在匹配搜索结果中,可点击显示的转录因子矩阵文件ID,就会在上面文本中显示出来,ID 后有标注 CPG岛则说明这个位点与它重合了,点击ID,会出现更详细的信息。
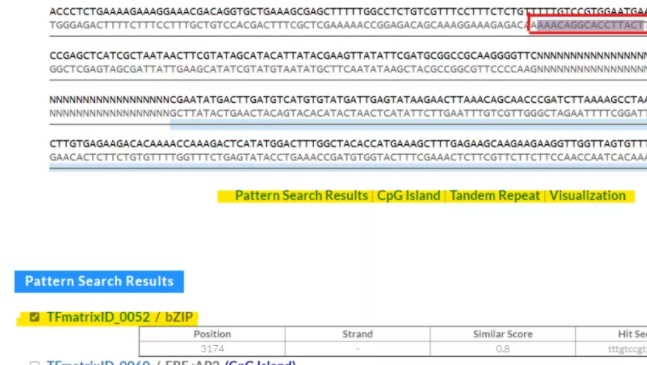 3. Pattern Search Result 的内容会比较长,继续下拉会看到以下内容。点击最下方可视化下面的图片,进行可视化展示
3. Pattern Search Result 的内容会比较长,继续下拉会看到以下内容。点击最下方可视化下面的图片,进行可视化展示
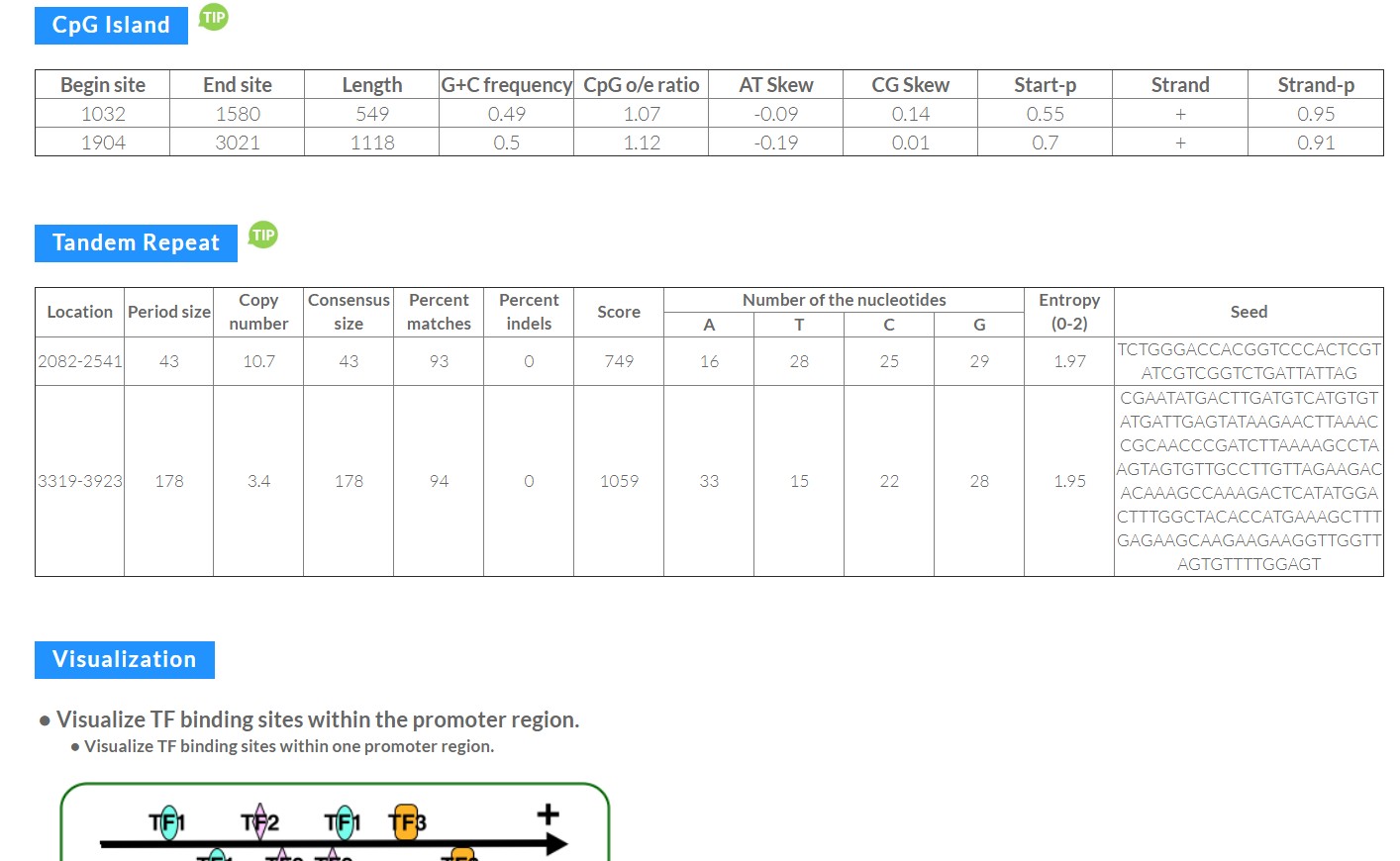
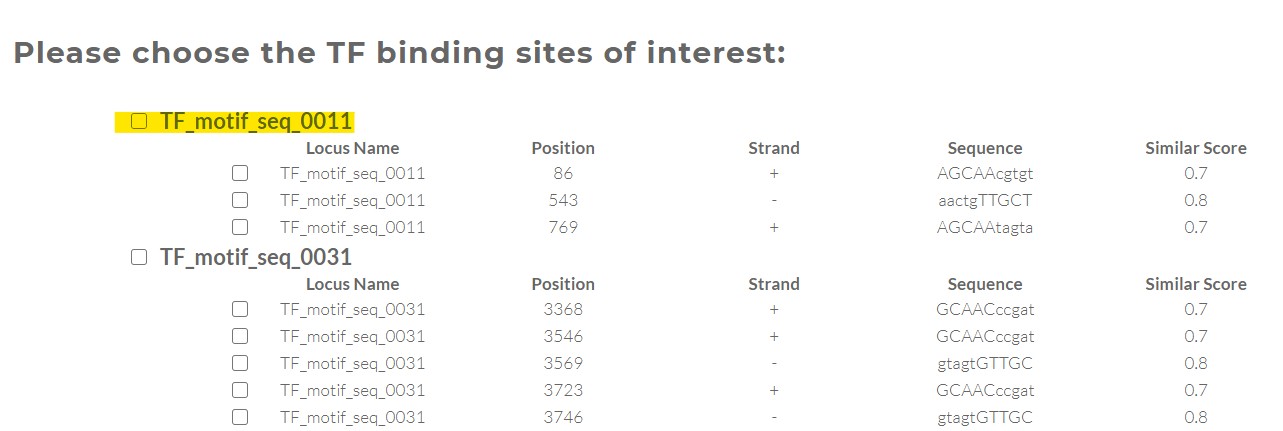
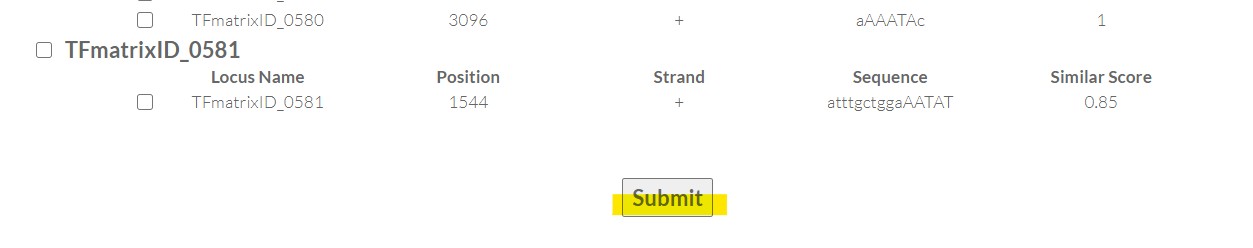
点击图片后如下,勾选感兴趣的选项,下拉到底部,点击提交
会进入如下界面:会用不同符号显示勾选的转录因子位点,点击Download可下载图片。
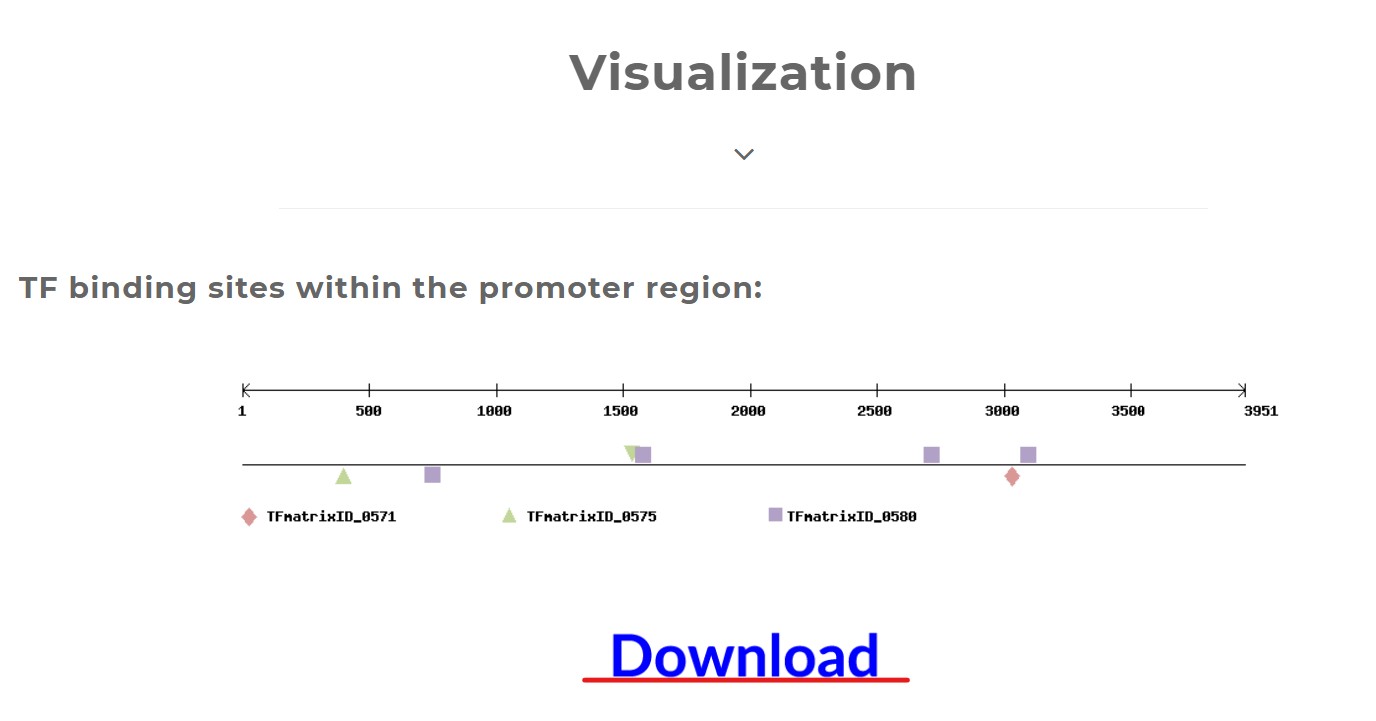
目前只有在输入文本可下载可视化图片,对于多个基因的启动子序列分析的上传文件无法在网站上可视化,可以得到结果文件后在GSDS网站或使用TBtool进行可视化。
PlantPAN3.0网址: http://PlantPAN.itps.ncku.edu.tw/
此外,我们在网易云课堂上有各种教学视频,有兴趣可以了解一下:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘
6. 更多学习内容:linux、perl、R语言画图,更多免费课程请点击以下链接:
http://m.study.163.com/provider/400000000234009/index.htm?share=1&shareId=1433306807
- 发表于 2020-07-13 18:10
- 阅读 ( 5895 )
- 分类:其他
你可能感兴趣的文章
- Broad GDAC Firehose—TCGA数据分析中心 5880 浏览
- HPA:人类蛋白图谱数据库 6184 浏览
- 常见的植物数据库(更新-Ing) 8625 浏览
- 转录组测序分析数据库介绍 18441 浏览