启动子序列提取-EPD真核生物启动子数据库!
基因结构有很多概念,经常会把我们绕晕,之前小编给大家整理过mRNA、CDS、ORF等概念知识(感兴趣的点击查看:CDS、cDNA、ORF等等傻傻分不清);今天我们再补充一些概念性知识及着重说下启动子以及启动子序列查找方法。
基础概念
1. 启动子(Promoter):启动子是RNA 聚合酶识别、结合和开始转录的一段DNA 序列,它含有RNA 聚合酶特异性结合和转录起始所需的保守序列,多数位于结构基因转录起始点的上游,启动子本身不被转录。所以一般所说的启动子是DNA序列上的结构,在mRNA、cDNA中它是不存在;但是也有一些例外,如tRNA启动子就位于转录起始点的下游,这些DNA序列是可以被转录的,只能说启动子一般位于转录起始位点的上游。
2. 转录组起始点(TSS):是指与新生RNA链第一个核苷酸相对应的DNA链上的碱基,通常为一个嘌呤(A 或G),即5’UTR的上游第一个碱基;注意转录起始点和起始密码子的区别。
3. 起始密码子和终止密码子:mRNA的开放阅读框架中,每3个相邻的核苷酸编码一种氨基酸,这种存在于mRNA开放阅读框架区的三联体形式的核苷酸序列称为密码子(codon);由A、U、C、G四种核苷酸可组成64个密码子,其中有61个密码子可编码氨基酸。AUG既编码甲硫氨酸,又作为多肽链合成的起始信号,作为起始信号的密码子称为起始密码子;而终止翻译的密码子称为终止密码子,包含3个:UAG、UAA、UGA。
4. UTR区:UTR(Untranslated Region),即非翻译区;在分子遗传学中,是指任意一个位于mRNA链编码序列两端的片段;如果其位于5′端,则称为5′非翻译区(5'-untranslated region,5'-UTR)(或"前导序列,leader"),反之若位于3′端,则称为3′非翻译区(3'-untranslated region,3'-UTR)(或"尾随序列,trailer")。尽管它们被称为"非翻译区",并且不是构成该基因的蛋白质编码区,但在5′非翻译区内的上游可读框可以被翻译成多肽。
5. 5'帽子(cap):真核生物mRNA的5'端有特殊的帽子(cap)结构,它由甲基化鸟苷酸经焦磷酸与mRNA的5'末端核苷酸相连,形成5',5'-三磷酸连接(5',5'-triphosphate linkage);这种结构有抗5'-核酸外切酶的降解作用;在蛋白质合成过程中,它有助于核糖体对mRNA的识别和结合,使翻译得以正确起始。
6. PolyA尾巴:真核生物mRNA尾部特有的150-200个腺苷酸残基,保护mRNA,免受核酸外切酶攻击,并且对转录终结、将mRNA从细胞核输出及进行翻译都十分重要;PolyA尾巴是mRNA转录后修饰加上去的,DNA基因序列中是不存在的,经mRNA反转录出的cDNA是有PolyA结构的。
7. CDS与ORF:这是一个经常被人混淆的两个概念;CDS是Coding sequence的缩写,是指编码一段蛋白产物的序列,是与蛋白质密码子一一对应的序列,注意其与mRNA序列的差异;ORF是open reading frame的缩写,翻译成开放阅读框,是指从一个起始密码子开始到一个终止密码子结束的一段序列,但并不是所有ORF都能表达出蛋白产物,但CDS必定是一个ORF,但也可能包括多个ORF,相反,每个ORF不一定都是CDS。
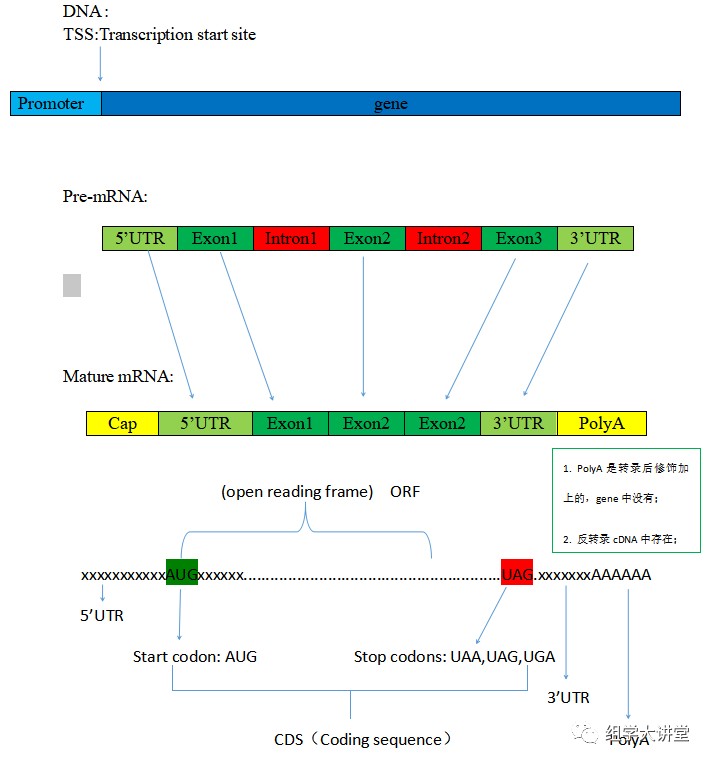 DNA/mRNA结构示意图
DNA/mRNA结构示意图
启动子序列的查找
在实际生信分析中,一般取转录组起始位置前1500-2000bp作为启动子区域序列,拥有生信技能的人提取及批量提取某些基因启动子序列简直就是小菜一碟;但是对于生信小白来说就难上青天了;不过还有一些在线数据库是可供我们查找提取启动子序列之用,常见的比如NCBI,ensembl,UCSC 等等;但小编觉得那些都不怎么好用,上周我们其实给大家分享过一个非常好用的植物启动子分析数据库PlantPAN3.0,简单好用,功能非常强大,感兴趣点此链接查看:植物启动子分析网站PlantPAN3.0-不可或缺的基因表达调控数据库!;今天我再跟大家分享一个小巧好用的真核生物启动子数据库:The eukaryotic promoter database(EPD)。
EPD数据库操作演示
EPD 是一个非冗余的真核启动子数据库,其转录起始位点已经通过实验验证。该数据库包含来自十余种物种的启动子序列信息(如下图),界面友好,操作简单,适合小白操作学习。
 1. EPD界面非常简单,如下图所示可在是检索框输入基因ID或者基因名(gene symbol),再选择物种,点击search即可,我们以拟南芥基AT1G66550为例。
1. EPD界面非常简单,如下图所示可在是检索框输入基因ID或者基因名(gene symbol),再选择物种,点击search即可,我们以拟南芥基AT1G66550为例。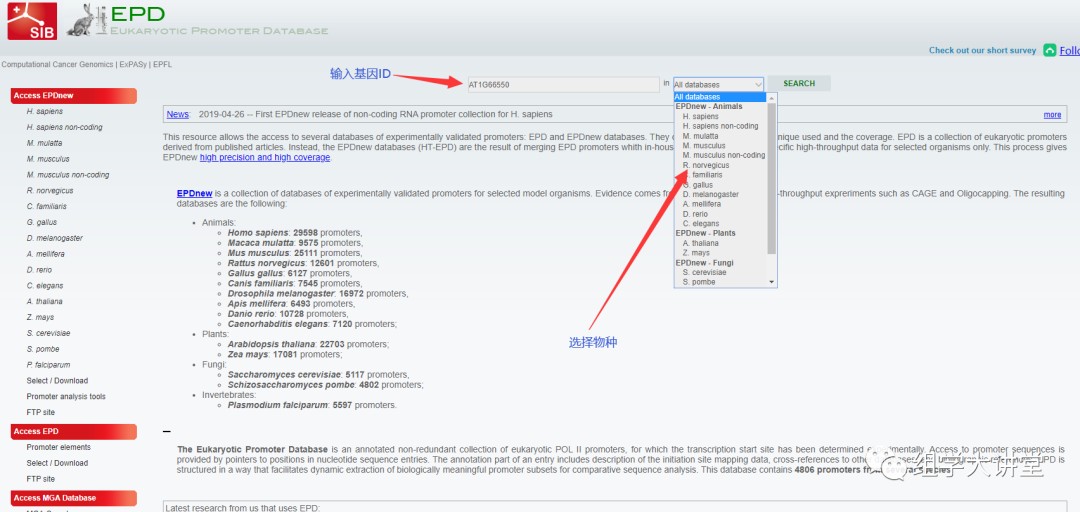
2. 有时候会检索出多个基因,选择自己感兴趣的即可,示例数据仅一个检索结果,直接给出了启动子序列信息页面,如下图,可在Sequence Retrieval Tool 下设置启动子序列提取范围,一般转录组起始点前1500-2000bp,设置好点击右侧的GET SEQL即可;还可以在Search Motif Tool项下预测TATA-box等顺式作用元件。
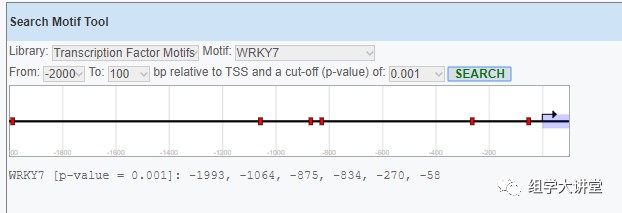
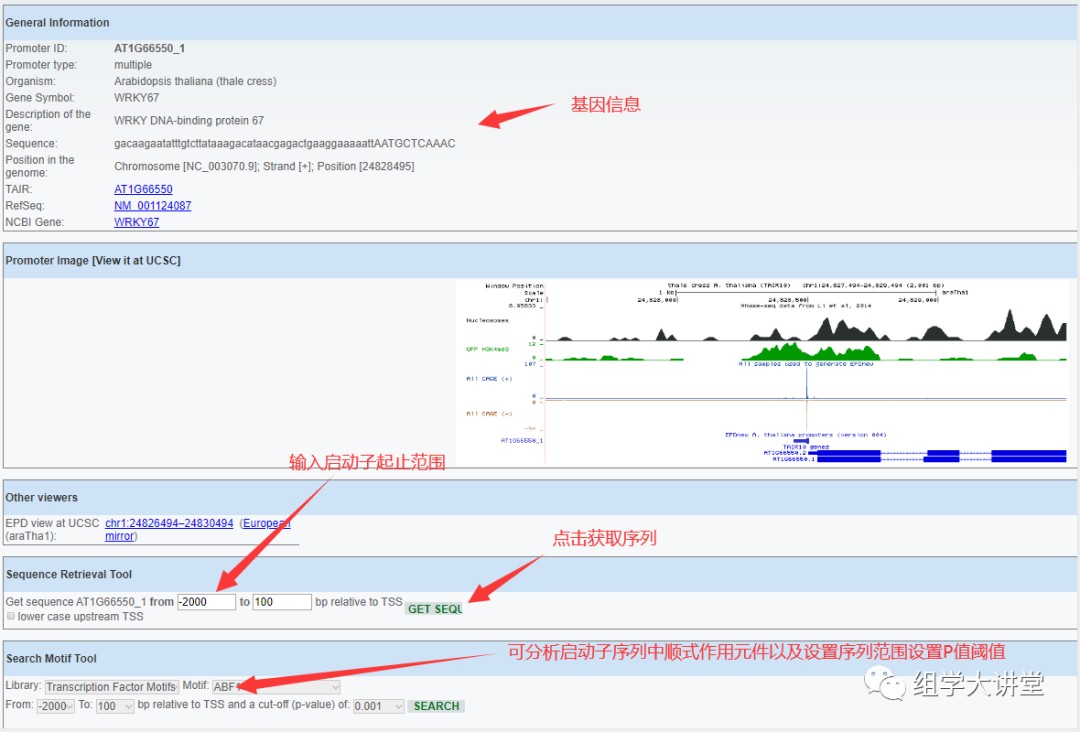 3. 下图是提取好的启动子序列及预测的顺式作用元件,不过小编觉得顺式作用元件绘图很是难看,我们完全可以用提取的序列用其他软件去绘图,顺式作用元件预测及绘图操作教程见链接:充电课-顺式作用元件分析(基础知识及预测)和收藏贴-顺式作用元件分布图绘制。
3. 下图是提取好的启动子序列及预测的顺式作用元件,不过小编觉得顺式作用元件绘图很是难看,我们完全可以用提取的序列用其他软件去绘图,顺式作用元件预测及绘图操作教程见链接:充电课-顺式作用元件分析(基础知识及预测)和收藏贴-顺式作用元件分布图绘制。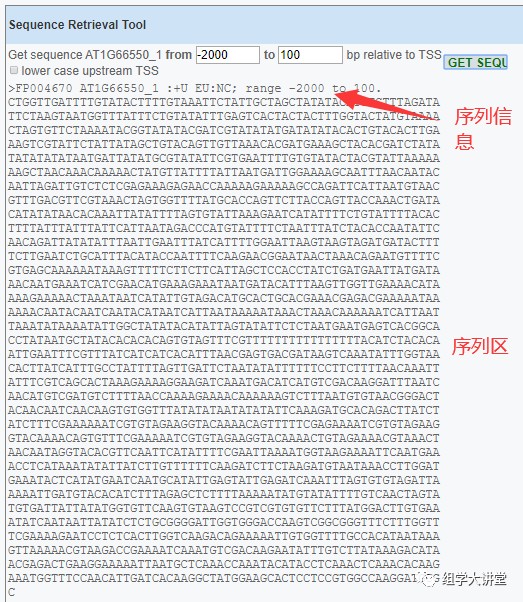
 好了,这样启动子序列就提取好了,你get到了吗?最后奉上EPD数据库网址:https://epd.epfl.ch//index.php
好了,这样启动子序列就提取好了,你get到了吗?最后奉上EPD数据库网址:https://epd.epfl.ch//index.php1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?多学点数据处理技能:学习链接:转录组标准分析后的数据挖掘
6. 学生物的必学生信技能:linux系统入门
7. 学生物的必学生信技能:Perl语言入门到精通
8. 学生物的必学生信技能:perl语言高级编程
9. 更多学习内容:linux、perl、R语言画图,更多免费课程请扫描下方二维码:
- 发表于 2020-09-07 09:16
- 阅读 ( 15747 )
- 分类:软件工具

