微生物群落功能注释-FAPROTAX数据库介绍及使用
扩增子分析视频课程推荐:https://bdtcd.xetslk.com/s/qZRiF
微生物多样性分析中,常常需要对微生物群落功能进行注释,FAPROTAX数据库方便简单效果好,一行代码即可生成功能注释表,今天就给大家介绍该数据库内容及使用方法。
FAPROTAX数据库简介
1. FAPROTAX是Louca等人根据已发表的文献手动构建的数据库,它把原核微生物的分类和代谢等功能对应起来,目前收集自4600多个原核微生物的80多个功能分组7600多条功能注释信息,并且还在不断更新。
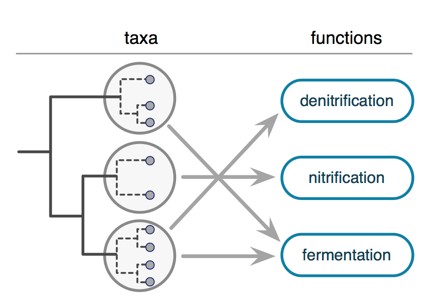
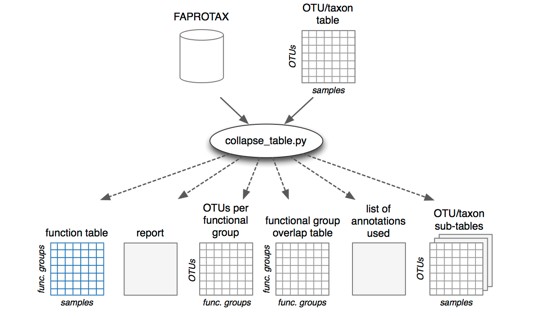
2. 作者编写了一个python脚本,把样本的OUT表转换成下图所示的六个表。分别是:样本的功能表、过程报告、每个功能分组的OTUs、每个功能分组重叠的表、分析过程用到的注释信息、输入样本OTU表的子表(仅列出与特定功能相关的OTU)。
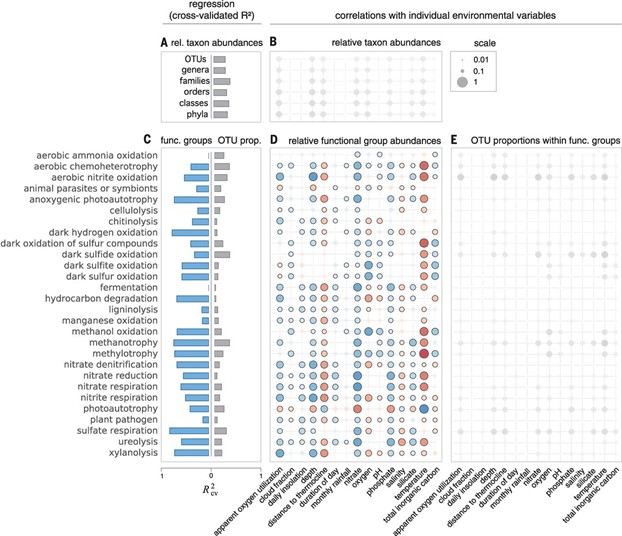
3. FAPROTAX会更适用于海洋湖泊生物化学过程(硫、氮、氢和碳循环)中的功能注释,因为作者发表的文章中就是用FAPROTAX来分析海洋微生物的分类和功能,所以这方面的功能更全一些,但很多已发表的文章也用它将进行土壤微生物的功能注释,效果也很好。下面是作者基于FAPROTAX数据库生成的功能表进行绘制的图片。
Docker使用
1. 准备文件:样本的OTU表(biom格式)、作者写的脚本和FAPROTAX数据库。获取链接:http://www.loucalab.com/archive/FAPROTAX/lib/php/index.php?section=Download
(1)下载

(2)解压后(中间两个文件)
2. 启动docker及下载扩增子镜像:
docker search omicsclass #搜索omicsclass提供的docker镜像docker pull omicsclass/ampliseq-q1 #下载扩增子镜像docker images #检查是否下载完成
3. 在D盘下建立FAPROTAX文件夹,把准备好的三个文件放进去。
4. 运行以下代码进入虚拟机后,查看文件。
docker run --rm -v D:/FAPROTAX:/work -it omicsclass/ampliseq-q1:latest #进入虚拟机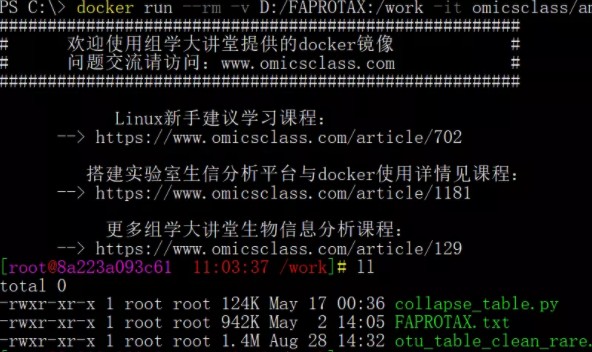
5. 运行以下代码,生成功能表和过程报告。
python collapse_table.py -i otu_table_clean_rare.biom --collapse_by_metadata 'taxonomy' -v --force -o faprotax1.txt -r faprotax_report.txt
功能表faprotax1,基于这个表就可以进行样本和注释功能的图片绘制(最好先进行筛选)。
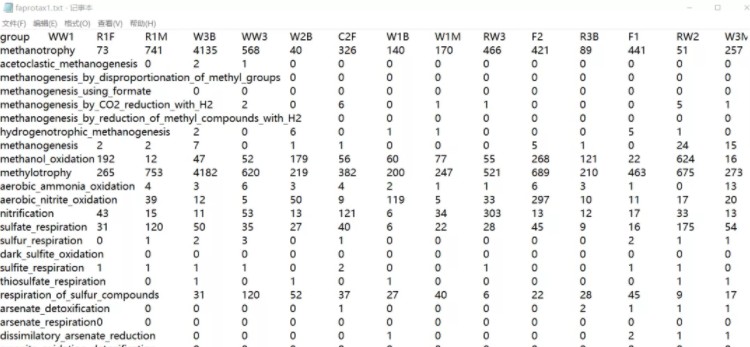
过程报告faprotax_report,可以看到具体信息,挑选感兴趣功能的进行筛选。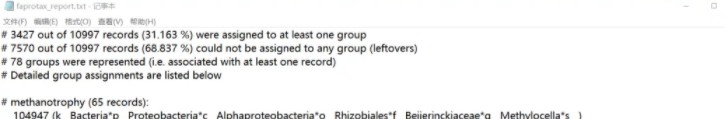
参考文章:DOI: 10.1126/science.aaf4507
更多FAPROTAX数据库使用说明:http://www.loucalab.com/archive/FAPROTAX/lib/php/index.php?section=Instructions
此外,我们在网易云课堂上有各种教学视频,有兴趣可以了解一下:
2.OTU网络图
6. 更多学习内容:linux、perl语言画图,更多免费课程请点击以下链接:
http://m.study.163.com/provider/400000000234009/index.htm?share=1&shareId=1433306807
- 发表于 2020-09-17 14:20
- 阅读 ( 15359 )
- 分类:宏基因组
