QIIME1格式转换
faprotaxs数据库注释得到的过程报告如何转化成绘物种分类功能热图所需的输入文件?
faprotaxs数据库注释得到的过程报告如何转化成绘物种分类功能热图所需的输入文件?
1.对过程报告整理:
grep 'OTU' -B 1 faprotax_report.txt | grep -v -P '^--$' > faprotax_report.clean
perl otu.pl -iclean faprotax_report.clean -o1 func_otu.txt -o2 func_nums.txt -o3 mat.txt -o4 otu_func.txt
得到功能有无矩阵mat.txt(格式同otu_table后跟物种注释)
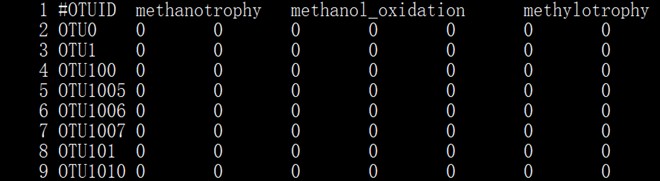
2.转换成biom格式文件:(docker上下载QIIME1镜像)
biom convert -i mat.txt -o otu_table.from_txt.biom --table-type="OTU table" --process-obs-metadata taxonomy --to-hdf5
3.在运用QIIME1脚本进行分类:
不同水平下相对丰度文件: summarize_taxa.py -i otu_table.from_txt.biom -o taxa_summary/relative_abundance --level 2,3,4,5,6,7
不同水平下绝对丰度文件: summarize_taxa.py -i otu_table.from_txt.biom -o taxa_summary/absolute_abundance -a --level 2,3,4,5,6,7
4.绘图:
- 输入文件:L3.txt R脚本
- Rscript faprofax1.R -i L3.txt -o draw -n class
- 结果:
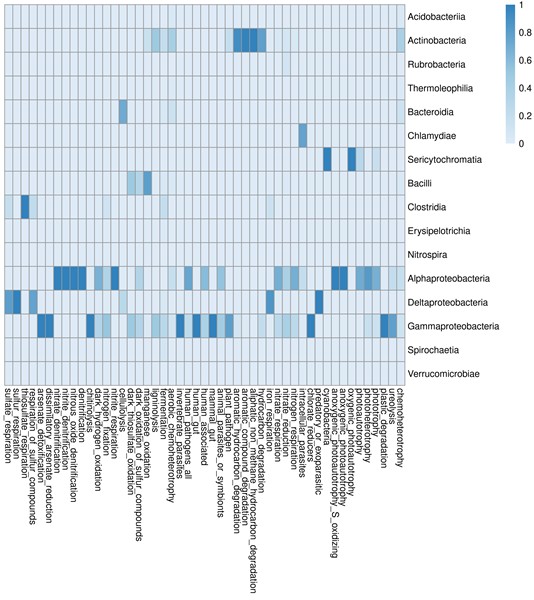
- 发表于 2020-10-14 15:19
- 阅读 ( 2956 )
- 分类:宏基因组
你可能感兴趣的文章
0 条评论
请先 登录 后评论
