Bugbase表型预测
一、BugBase数据库介绍
1、Bugbase用于预测人体或环境样本中原核微生物的表型,主要包含以下几个方面:革兰氏阴性、革兰氏阳性、耐氧性、生物膜合成、致病潜力、移动元件含量、氧化胁迫耐受等。文章于17年发表在预印本杂志bioRxiv上,没有正式发表。
 2、了解原核微生物表型作用:在人体中,通过了解微生物表型,可以选择不同治疗方法,比如知道胃肠道中革兰氏阴性菌和革兰氏阳性菌的比例,可以采用更具针对性的抗生素治疗;确定粪便样品的厌氧菌和病原体含量可以帮助预测肠胃道的健康等。
2、了解原核微生物表型作用:在人体中,通过了解微生物表型,可以选择不同治疗方法,比如知道胃肠道中革兰氏阴性菌和革兰氏阳性菌的比例,可以采用更具针对性的抗生素治疗;确定粪便样品的厌氧菌和病原体含量可以帮助预测肠胃道的健康等。
3、BugBase工作流程图:输入两个文件,运行脚本,得到四种文件。

二、使用方式
1、在线网站.https://bugbase.cs.umn.edu/
点击parse data,进入上传数据页面。输入的文件有两个,网站对文件的格式有着明确要求。
首先是OTU_table:必须是BIOM1.0JSON格式(文件格式转化参考:https://www.omicsclass.com/article/1314);16S必须是GreenGenes为van靠数据库;宏基因组以IMG为参考;文件大小不超过15M;
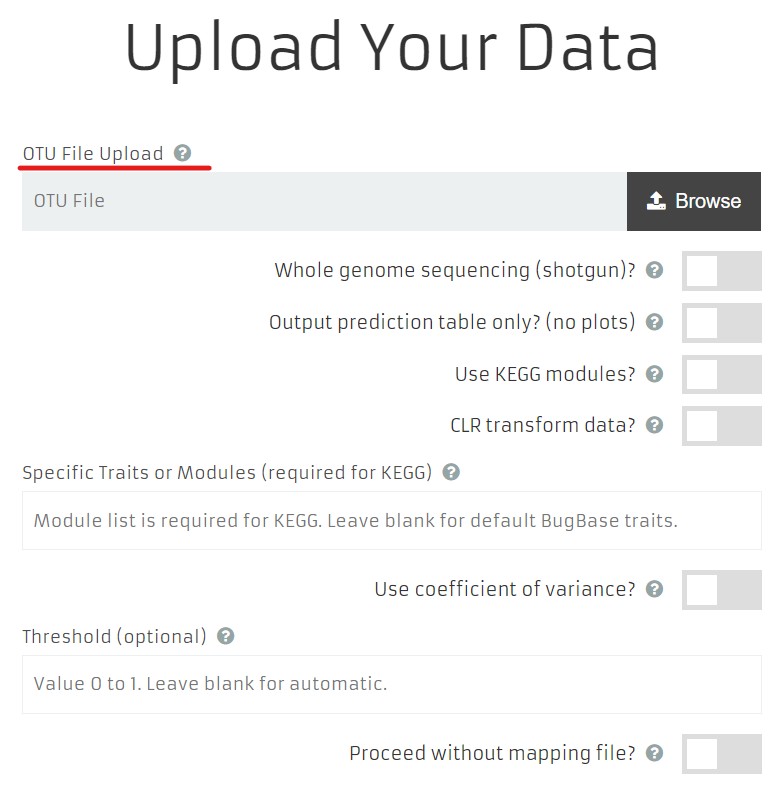
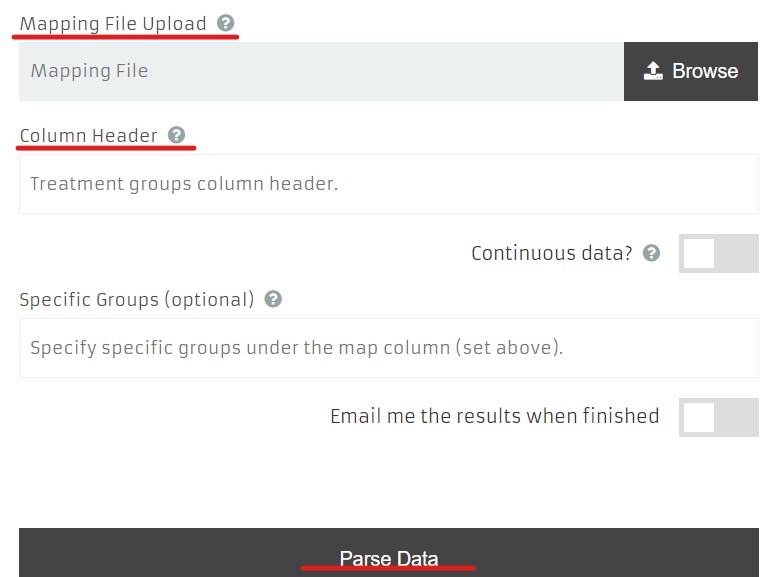 其次是样本信息文件:制表符分隔;第一行必须以#SampleID起始;第一行全为列标题;第一列必须为SampleID;只允许使用字母、数字、下划线和连字符;不允许包含空格,逗号、引号、括号。示例:
其次是样本信息文件:制表符分隔;第一行必须以#SampleID起始;第一行全为列标题;第一列必须为SampleID;只允许使用字母、数字、下划线和连字符;不允许包含空格,逗号、引号、括号。示例:
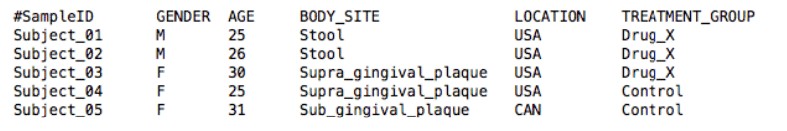 最后要指定分组名称(如示例中的 BODY_SITE),提交数据即可。
最后要指定分组名称(如示例中的 BODY_SITE),提交数据即可。
2、本地使用
下载QIIME1镜像:docker pull omicsclass/ampliseq-q1:v1.2(镜像里安装好运行所需的所有R包)
在本地文件夹 D:\project\bugbase 中放入号准备的文件:

进入镜像:docker run --rm -v D:\project\bugbase:/work -it omicsclass/ampliseq-q1:v1.2
运行下行代码即可出结果。
run.bugbase.r -i table.from_txt_json.biom -m fastmap.txt -c loc -o bugbase_loc
参数解释(部分):
-i otu_table biom1.0格式文件
-m 样品信息表
-c 指定样品分组
-o 输出文件名称
-t 指定分类水平 1-7,默认门水平
-p 表型,特殊表型预测
-x 只输出预测表,不出图片
-T 阈值 0-1,可指定过滤阈值
三、结果
运行成功会有如下显示:分别是标准化的OTUtable、阈值图片,每种表型预测的具体OTU、每种表型预测相对丰度图、统计表。
参考文章
doi:https://doi.org/10.1101/133462
此外,我们在网易云课堂上有各种教学视频,有兴趣可以了解一下:https://study.omicsclass.com/index
- 发表于 2020-11-03 17:19
- 阅读 ( 12177 )
- 分类:宏基因组
