机器学习文章套路-预测土壤枯萎病-ISME IF=9
前面我们介绍了采用机器学习的算法利用肠道微生物菌群特征预测疾病的文章(肠道微生物诊断慢性肾病),这次我们再介绍一下其在农业土壤微生物中的应用并发表高水平文章的例子:
- The ISME Journal, [IF 9.49] 2020-07-17
- DOI:https://doi.org/10.1038/s41396-020-0720-5
实验背景
镰刀菌枯萎病作为土传病害的典型代表,在世界范围内的多种作物频繁爆发。在过去的几十年里,相关学者进行了大量案例研究发现,尽管枯萎病爆发与土壤中病原菌丰度呈正相关关系,但并不存在可以确定是否发病的阈值。土壤是一个化学组成复杂、物理相系交错、生物及功能神秘的体系,对镰刀菌的存活及致病能力影响难以揭示,但普遍认为土壤抑病功能很难从个别微生物及功能上说明问题。因此,从分子生态学角度出发,以整合分析的方法来解析发病土壤微生物群落的普遍性特征,有望能为更好地防控土传枯萎病提供解决方案。
数据收集
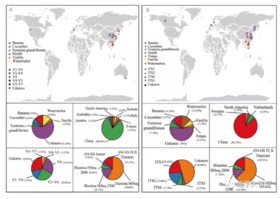
作者收集了世界上公开发表的大部分有关镰刀菌枯萎病相关的土壤微生物测序研究(1500多个土壤样本,包括真菌,细菌等),并展示了这些研究主要的分布,发现大部分研究取样点都在中国。
机器学习应用
作者想通过机器学习的方法,找到发病土壤与未发病土壤微生物群落结构差异来预测枯萎病的发生,把火热的机器学习用到生物领域也是这篇文章发高分的关键。
发现差异
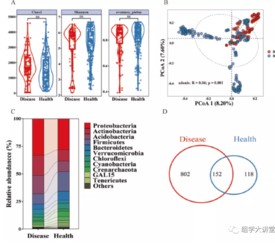
1.健康土壤与发病土壤多样性差异(细菌)
作者分析发现健康土壤和发病土壤alpha多样性没有显著差异(A),但是beta多样性分析存在差异(B):通过Bray-Curtis举例对群落进行PCoA排序,通过多元置换方差分析进行差异检测后发现发病细菌群落和健康土壤群落之间显著不同(p = 0.001;permutational multivariate analysis of variance (PERMANOVA) by Adonis);另外物种组成上存在差异(C,D)。
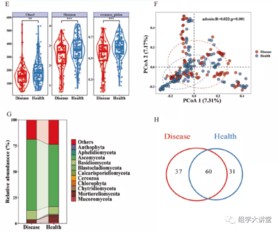
2.健康土壤与发病土壤多样性差异(真菌)
相较于土壤中的细菌,土壤中的真菌差异更加明显:alpha多样性与beta多样性都存在差异:
机器学习区分健康与发病土壤
作者将收集来的数据随机分成两组:其中80%的数据用于预测模型训练构建,剩余20%的数据用于模型验证。机器学习使用到的方法包括:随机森林(RF)、支持向量机(SVM)和逻辑回归(LR)。作者分别对发病土壤和健康土壤的微生物群落进行学习并构建分类模型,根据准确度,作者选择了随机森林(一种用于分类和回归分析的稳定机器学习算法)创建了分类模型(A-D)。作者用不同分类水平的物种丰度表构建预测分类模型,发现在OTU水平构建的分类模型最准确(E,F)。

独立验证实验
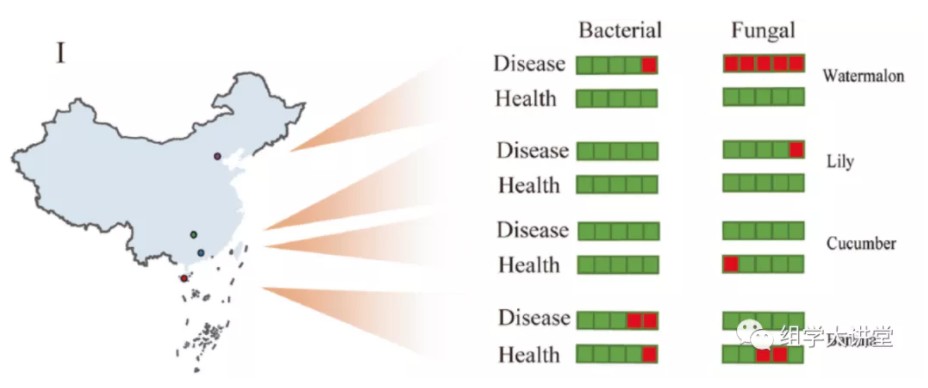
另外,为判断预测模型准确性,增加独立验证实验:作者于2019年4月下旬分别从海南、广东、北京和江苏采集了包含香蕉、黄瓜、西瓜和百合在内的4种不同作物的田间土壤样品,采样时选择至少连作5年且遭受镰刀菌枯萎病感染的地块,同时选择了没有发生枯萎病的新开垦农田进行健康土壤收集,为了避免地理因素引起的差异,在病害地附近选择健康地。使用细菌模型对样本分类预测,发现所有样本的平均准确率为90.00%,其中对患病样本的准确率为87.50%,对健康样本的准确率为92.50%(I)。使用真菌模型进行分类预测,表明所有样本的平均准确率为80.00%,其中对病土样本的准确率为97.50%,对健康土样本的准确率为62.50%(I)。这一结果说明通过随机森林建立的模型得到的分类器对枯萎病发病和健康土壤的微生物群落特征进行了良好的区分。
总结
机器学习的文章,至少需要三组数据,训练数据集,验证数据集,用于构建和验证模型的准确性。要想发高分文章必须还要有独立验证数据集,以验证预测模型的普适性。这种思路已经成为发文套路。机器学习相关分析代码:(肠道微生物诊断慢性肾病)
Yuan, J., Wen, T., Zhang, H. et al. Predicting disease occurrence with high accuracy based on soil macroecological patterns of Fusarium wilt. ISME J 14, 2936–2950 (2020). https://doi.org/10.1038/s41396-020-0720-5
课程推荐:微生物扩增子分析课程实操 微生物16S/ITS/18S分析原理及结果解读
延伸阅读:
1. 微生物测序原理2. 肠道“君”与人类二三事(科普)3. 什么是OTU4. alpha多样性5. GraPhlAn树状图6. OTU网络图MENA7. beta多样性解读
8. 一文读懂微生物多样性中的各种排序分析方法异同点9. 微生物组间差异分析之LEfSe分析10. 用测序研究微生物多样性,您选对区了吗?11.微生物多样性分析docker镜像使用12.生成OTU方法如何选择,去噪还是聚类?13.微生物多样性组间差异分析—STAMP使用!
14.16S rDNA你知道多少?
- 发表于 2020-12-18 20:06
- 阅读 ( 3559 )
- 分类:宏基因组
你可能感兴趣的文章
- 微生物与环境因子,代谢物,功能等相关性分析linkET 1939 浏览
- 微生物多样性扩增子(16S/18S/ITS)课程更新v2.0 1801 浏览
- PICRUSt2扩增子功能预测与差异比较分析 4184 浏览
- 微生物多样性分类柱状图按照分组进行排序脚本升级;tax_bar_plot.r 2128 浏览
- qiime2 18S 分类器构建 3622 浏览
- 扩增子16S/ITS/18S微生物多样性课程更新-机器学习随机森林分析 3356 浏览
