使用方法:
$Rscript DEG_limma.R -h
usage: DEG_limma.R [-h] -e filepath -m filepath -t treatname --control CONTROL
--case CASE [-f fdr] [-c fc] [-s size] [-a alpha]
[-X x.lab] [-Y y.lab] [-T title] [-H height] [-W width]
[-o path] [-p prefix]
limma analysis: https://www.omicsclass.com/article/1519
optional arguments:
-h, --help show this help message and exit
-e filepath, --expr filepath
input read count file[required]
-m filepath, --metadata filepath
input metadata file[required]
-t treatname, --treatname treatname
treat colname in group file[required]
--control CONTROL set control group name[required]
--case CASE set case group name[required]
-f fdr, --fdr fdr set fdr threshold[default 0.05]
-c fc, --fc fc set fold change threshold[default 2]
-s size, --size size point size[optional,default:0.7]
-a alpha, --alpha alpha
point transparency[0-1][optional,default:1]
-X x.lab, --x.lab x.lab
the label for x axis[optional,default:log2FC]
-Y y.lab, --y.lab y.lab
the label for y axis[optional,default:-log10(FDR)]
-T title, --title title
the label for main title[optional,default:Volcano]
-H height, --height height
the height of pic inches[default:5]
-W width, --width width
the width of pic inches[default:5]
-o path, --outdir path
output file directory[default:/share/work/fangs]
-p prefix, --prefix prefix
out file name prefix[default:Volcano]
参数说明:
-e 输入基因表达矩阵文件,必须为count表达文件:
| TCGA-LN-A9FP-01A-31R-A38D-31
| TCGA-LN-A4MQ-01A-11R-A28J-31
| TCGA-IG-A3YB-01A-11R-A36D-31
| TCGA-IC-A6RF-01A-13R-A336-31
| TCGA-VR-A8EX-01A-11R-A36D-31
| TCGA-L5-A88S-01A-11R-A36D-31
| TCGA-IG-A6QS-01A-12R-A336-31
|
| 1447 | 3247 | 1319 | 2943 | 3462 | 1109 | 1475 |
| 18 | 0 | 1 | 2 | 0 | 2 | 0 |
| 2084 | 2807 | 2798 | 2888 | 2683 | 1839 | 4146 |
| 1164 | 2339 | 1329 | 1271 | 941 | 622 | 899 |
| 553 | 1686 | 273 | 516 | 582 | 355 | 639 |
| 958 | 136 | 770 | 546 | 164 | 570 | 410 |
| 27342 | 2164 | 2073 | 1757 | 2170 | 9953 | 2956 |
-m metadata文件路径,样本的分组信息,第一列必须和表达文件的样本名称对应:
| | | | | |
| TCGA-LN-A9FP-01A-31R-A38D-31 |
| | | | | |
| TCGA-LN-A4MQ-01A-11R-A28J-31 |
| | | | | |
| TCGA-IG-A3YB-01A-11R-A36D-31 |
| | | | | |
| TCGA-IC-A6RF-01A-13R-A336-31 |
| | | | | |
| TCGA-VR-A8EX-01A-11R-A36D-31 |
| | | | | |
| TCGA-L5-A88S-01A-11R-A36D-31 |
| | | | | |
| TCGA-IG-A6QS-01A-12R-A336-31 |
| | | | | |
-t subtype.hclust --case S1 --control S2
指定metadata 分组列名,分组里面的比较组名字,如果分组名字有空格,应该用引号引起来:"Stage IA"
--fdr 0.05 --fc 2
设置差异基因的筛选条件:显著性和差异倍数(默认fdr=0.05,fold change=2)
使用举例:
$$Rscript DEG_limma.R -e TCGA-ESCA_gene_expression_Counts.tsv \
> --fdr 0.05 --fc 2 -m metadata.group.tsv -t subtype.hclust \
> --case S1 --control S2 -p S1_vs_S2.limma
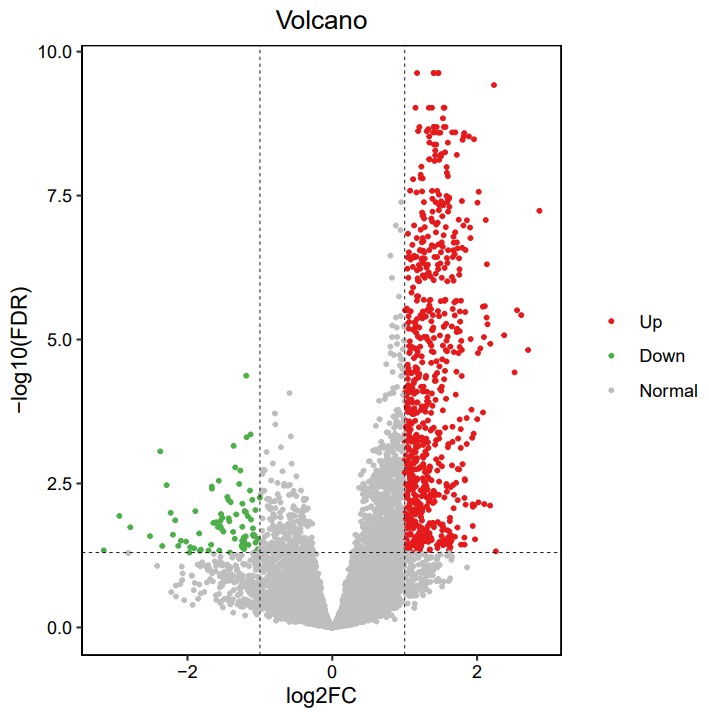
结果展示:
火山图: