蛋白质定量—MaxQuant软件的使用
简介
MaxQuant是基于质谱(Ms)的蛋白质组学数据分析最常用的平台之一,目前认可度相对较高,可针对多种质谱数据。拥有自己的肽段搜索引擎- Andromeda。对于碰撞诱导离解(CID)、高能碰撞离解(HCD)和电子转移离解(ETD)所产生的串联光谱可以很容易地用MaxQuant进行分析。
使用
1.打开maxquant界面
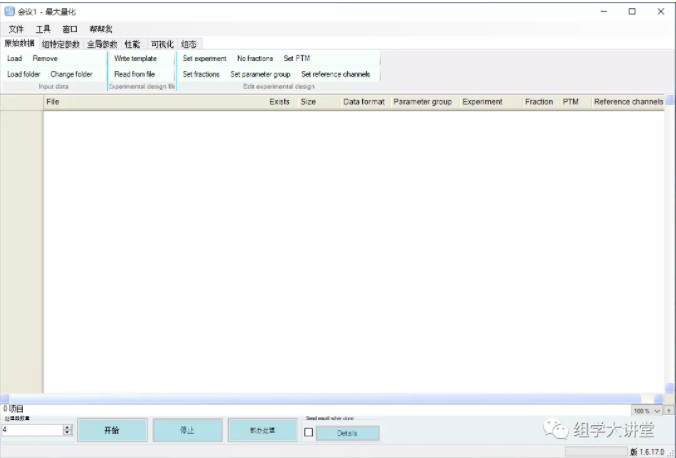
6大参数配置模块,大部分默认就可以,较重要模块介绍如下:
原始数据:导入原始数据和设计实验,包括组分和样本,Group(指不同的实验,如label/SILAC同时进行即为2个Group0和Group1,一般跑一个就可以);
组特定参数:Group参数的设置,一般只是Group0;
性能:显示正在运行的具体步骤和动态;
可视化:数据可视化模块, 能在labelfree定量中呈现三维图形。视情况可不设置;
组态:用于配置修饰、酶,以及数据库ID规则设置等,一般无额外需求,无需设置。
2.导入原始数据“Load”:将.raw文件直接导入软件,根据样本选择“No fractions”或者”Set fractions”。
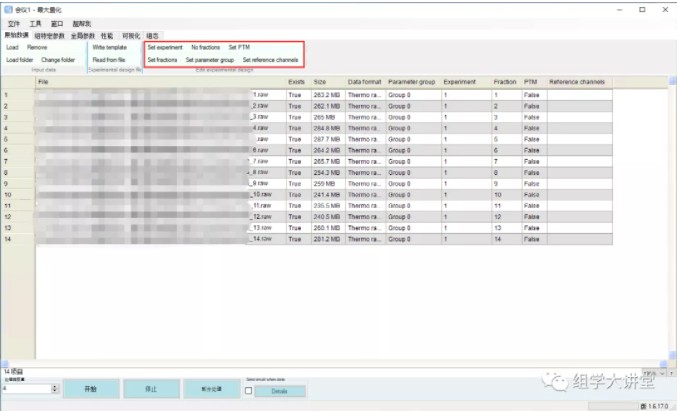
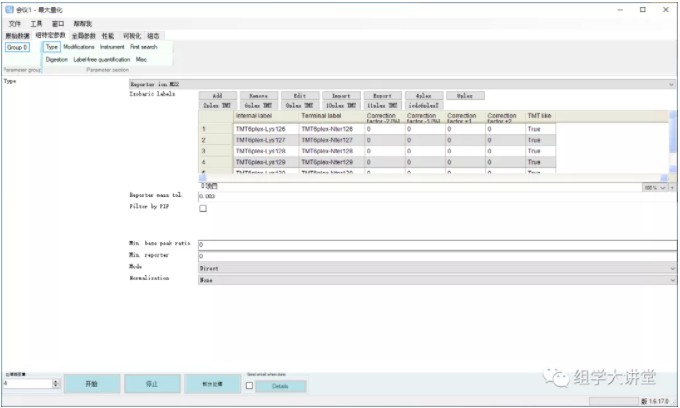
3.设置组特定参数Type 中设置使用的标记技术。
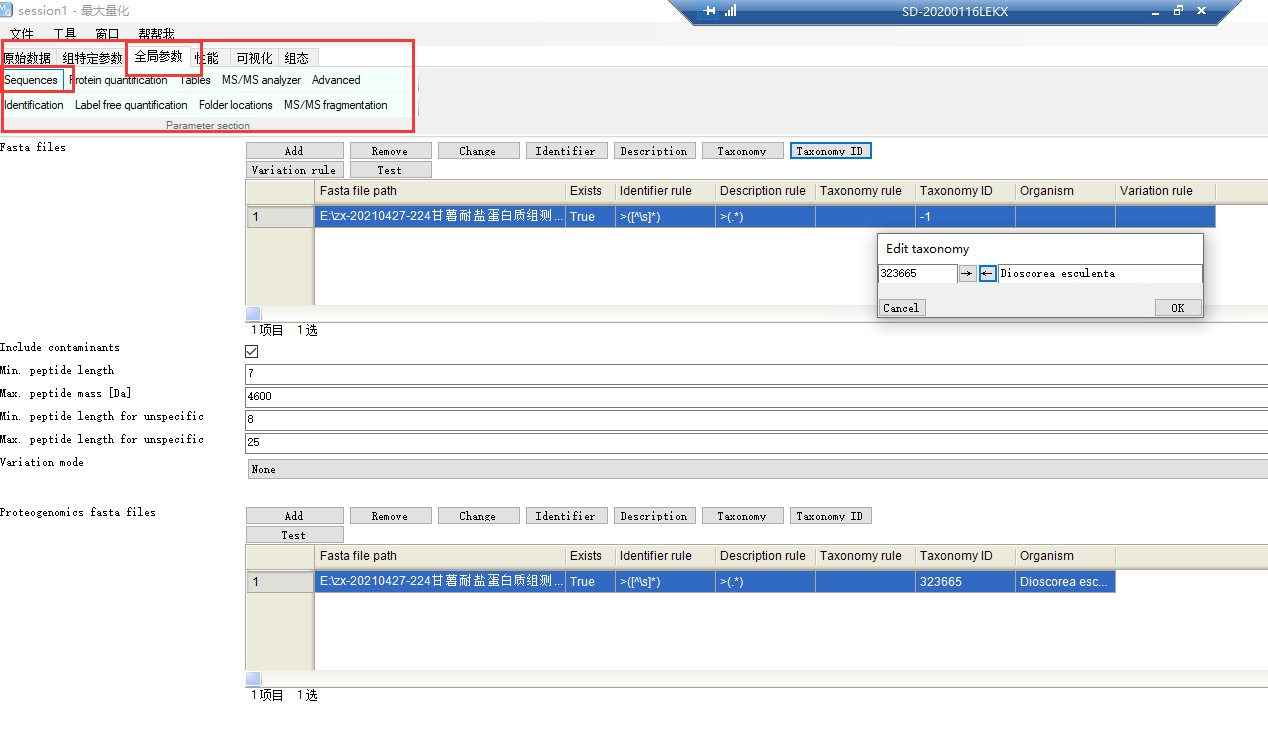
4.设置全局参数
Sequences 中添加物种蛋白质序列文件。设置Taxonomy ID,输入物种拉丁文名称即可得到。
5.点击开始,启动运行。
好了今天先介绍到这里,你也动手去试试吧!
此外,我们在网易云课堂上有各种教学视频,有兴趣可以了解一下:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘
6. 更多学习内容:linux、perl、R语言画图,更多免费课程请点击以下链接:
- 发表于 2021-08-04 14:08
- 阅读 ( 7081 )
- 分类:蛋白质组学
你可能感兴趣的文章
- 蛋白质组学数据定量搜库 | MaxQuant软件的使用 6195 浏览
- Windows里MaxQuant下载与安装 3728 浏览
- Linux里安装Maxquant 2184 浏览
- Maxquant报错 2973 浏览
- 蛋白质定量—MaxQuant软件分析多样品定量 6358 浏览
- 蛋白定量-Maxquant软件输出结果说明 7817 浏览
