蛋白定量-Maxquant软件输出结果说明
之前小编在蛋白质定量—MaxQuant软件的使用这篇文章中分享了Maxquant软件的使用方法。分析完成后会产生很多的结果文件,第一次使用的同学可能会很困惑,这里再给大家介绍一下如何查看其结果文件。
结果解读
1.如果参数中路径没有额外设置,那么在原始数据同级目录下会得到搜库结果: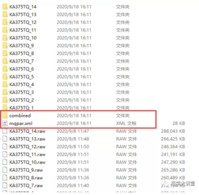
其中,mqpar.xml是所有参数配置文件,若二次搜库,可直接导入MaxQuant中,无需重复设置。所以,一般建议保留下来,这样也可对结果的设置进行追溯。
2.combined文件夹包含了搜库结果,如果是DIA建库,导入的即是这个文件夹。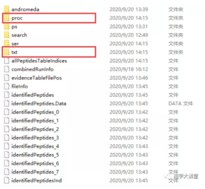
其中proc文件夹下的#runningTimes.txt文件,记录了每一步运行的时间。
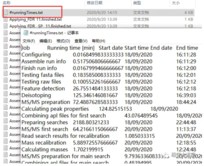
3.蛋白质鉴定和定量的全部结果都位于txt文件夹下。
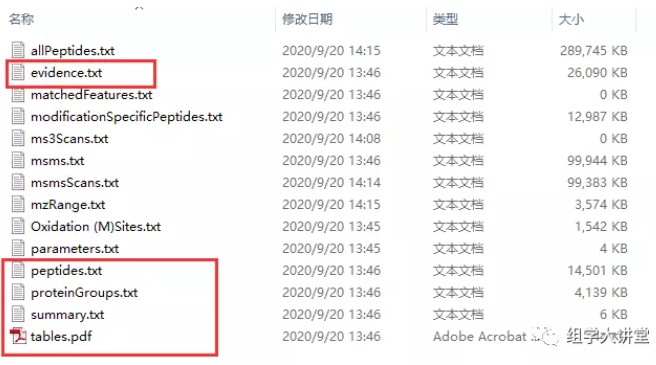
其中msms.txt、peptides.txt、proteinGroups.txt分别为谱图、肽段和蛋白结果文件。另外summary.txt文件记录了所有原始数据文件的总体信息;evidence.txt文件记录了已鉴定肽段的所有信息。每个文件的列数(表头)都很多,就不一一列举了,需详细了解的同学可自行查阅结果中tables.pdf文件,所有文件都有详细解释。
好了今天先介绍到这里,你也动手去试试吧!
此外,我们在网易云课堂上有各种教学视频,有兴趣可以了解一下:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘
6. 更多学习内容:linux、perl、R语言画图,更多免费课程请点击以下链接:
- 发表于 2021-08-04 14:27
- 阅读 ( 7818 )
- 分类:蛋白质组学
你可能感兴趣的文章
- 蛋白质组学数据定量搜库 | MaxQuant软件的使用 6196 浏览
- Windows里MaxQuant下载与安装 3728 浏览
- Linux里安装Maxquant 2185 浏览
- Maxquant报错 2974 浏览
- 蛋白质定量—MaxQuant软件分析多样品定量 6360 浏览
