群体进化选择消除分析
选择消除
在自然选择下,一种新的正向选择或有利性状会在群体中增加频率(普遍率),这是适应性进化的主要驱动力。一个性状要经历正向选择,必须有两个显著特征。首先,这种特性必须是有益的;换句话说,它必须增加有机体生存和繁殖的可能性。第二,这种特征必须是可遗传的,这样才能遗传给生物体的后代。如果一个特征导致更多的后代拥有相同的特征,那么这个特征比随机产生的特征更可能在群体中变得普遍。在分子水平上,当一种特定的DNA变异由于对携带它的生物体的影响而变得更加普遍时,选择就发生了。
简单的说就是基因组某区域由于受到了选择而消除多态性,即遗传多样性降低,在群体中出现高频的等位基因和低频的等位基因。图1显示了在选择前后沿一条染色体的多态性,包括受选择基因。祖先的等位基因(Ancestral alleles)显示为灰色,衍生的等位基因(Non-ancestral alleles)显示为蓝色。当一个新的正向选择基因(红色)上升到高频率时,染色体上邻近的连锁等位基因也随之上升到高频率,形成了选择消除(Selective sweep)
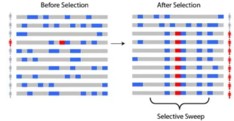
选择消除分析
选择消除分析有助于我们对物种自然历史的理解。在驯化物种中,通常可以利用对品种或品种间表型多样性的理解,结合选择消除分析,来更好地理解控制表型变异的基本生物学意义。例如,在狗(所有陆生脊椎动物中体型多样性最大的物种)中,研究人员试图在最小和最大的品种之间进行选择消除分析。发现与大型犬种相比,小型犬种几乎只拥有一种单一基因。该基因编码胰岛素样生长因子1(IGF1),已有研究表明它同样影响着小鼠和人类的体型。类似的研究设计已应用于几种不同的驯化物种,如牛、猪、羊、兔子和水稻。
1. 基于群体分化(FST 法)
群体的固定系数 F 反映了群体等位基因杂合性水平。固定系数 F 是 F 统计量(FST)的一个特例:FST 分析扫描全基因组范围内的 SNP 位点,计算每个 SNP 的 FST 值,其取值范围为0~1,0代表群体间所有位点都没有出现分化,1代表群体间已经完全分化,故 FST 值越大,群体分化程度越高,受选择程度越高。
2. 基于群体多样性分析(π)
核苷酸多样性 π 值反映了群体基因组碱基多样性水平。π 值用来分析碱基多态性,多态性越低,受选择程度越高。
3. 基于群体中性进化分析
Tajima 检验将计算并标准化目标序列 alignment 中每条序列的分离位点数目以及每条序列的核苷酸多样性的值,反映了基因序列在进化过程中是否遵循中性进化模型。Tajima's D 分析表示中性进化,越偏离0,受选择程度越高。
4. 基于群体基因连锁不平衡分析(XP-EHH 法)
群体间扩展单倍型纯合度法(XP-EHH)是在原有 EHH 方法上提出的一种新的基于连锁不平衡计算两个群体间近期发生选择信号的方法。XP-EHH 值用来检测在一个群体中为纯合而在另一个群体中为多态的处于选择固定阶段的位点,值越大,受选择程度越高。
5. 基于群体等位基因频率谱分析(XP-CLR 法)
跨群体混合似然比法(XP-CLR)是利用群体间的等位基因频率的差异,对覆盖扩展连锁区域末端等位基因变异频率进行扫描,高 XP-CLR 的临近窗口被聚集到同一种特征下,每一种特征代表一个单一选择扫描效应从而有效定位受正选择的位点。
6. 基于个体间SNP差异分析(RSD)
相对 SNP 密度(Relative SNP Density,RSD)分析只针对纯合 SNP 位点,用于筛选个体特异的受选择区域,主要应用于驯化物种品系间的选择分析。
7. 基于纯合区域分析(ROH)
一般认为野生型个体中 ROH 的数量和总长度少于驯化型个体,基于此统计不同群体共有纯合区域/不同个体连续纯合区域间的差异进行分析。
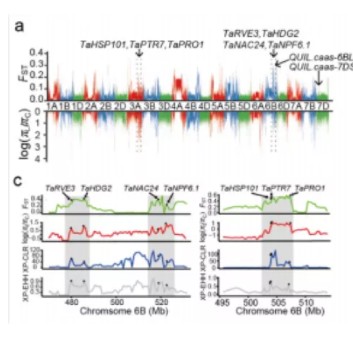
经典选择消除多种方法结合分析图例
(图片引自 Chen HF, et al. 2019
- 发表于 2021-09-09 17:00
- 阅读 ( 9875 )
- 分类:遗传进化
你可能感兴趣的文章
- 2023年秋季线下/直播培训火热报名中! 4181 浏览
- 群体遗传进化与GWAS分析动植物文献 4861 浏览
- 群体进化和GWAS文章没那么难发! 3855 浏览
- 群体进化和GWAS文章没那么难发! 5109 浏览
