Uniprot蛋白数据库介绍及使用详解!
Uniprot数据库是资源最广、信息最丰富的蛋白质数据库,是查询蛋白功能的首选数据库。Uniprot数据库由Swiss-Prot、TrEMBL和PIR-PSD三大子数据库构成,数据主要来自于各物种基因组测序完成后得到的全基因蛋白质序列,并包含了很多来自文献中的蛋白及其功能信息。尤其是swiss-prot 子数据库,库中蛋白质信息都是手工核对过的 ,非冗余, 有详细注释信息的蛋白数据。作为一名科研工作者,Uniprot数据库的使用技能应该是必备的技能之一。
Uniprot数据库简介
Uniprot 数据库包含蛋白质序列,功能信息,研究论文索引等信息,整合了包括EBI( European Bioinformatics Institute)、SIB(the Swiss Institute of Bioinformatics)、PIR(Protein Information Resource)三大数据库的资源。
EBI( European Bioinformatics Institute):
欧洲生物信息学研究所(EMBL-EBI)是欧洲生命科学旗舰实验室EMBL的一部分。位于英国剑桥欣克斯顿的惠康基因组校内,是世界上基因组学领域最强的存在之一。
SIB(the Swiss Institute of Bioinformatics):
瑞士日内瓦的SIB维护着ExPASy(专家蛋白质分析系统)服务器,这里包含有蛋白质组学工具和数据库的主要资源。
PIR(Protein Information Resource):
PIR是美国国家生物医学研究基金会(NBRF)于1984年创立,旨在协助研究人员识别和解释蛋白质序列信息。
Uniprot数据库主要子数据库组成:
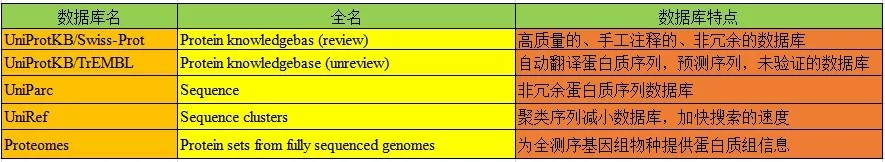 以上子数据库间的关系如下:uniprot会收集EMBL,GenBank,DDBJ等公共数据库中的蛋白质序列及功能信息等原始数据,处理后存入UniParc的非冗余蛋白质序列数据库;UniPrc作为数据仓库,再分别给UniProtKB,Proteomes,UNIRef提供可靠的数据集,其中在UniProtKB数据库中Swiss-Prot是由TrEMBL经过手动注释后得到的高质量非冗余数据库,也是我们最常用的蛋白质数据库之一。
以上子数据库间的关系如下:uniprot会收集EMBL,GenBank,DDBJ等公共数据库中的蛋白质序列及功能信息等原始数据,处理后存入UniParc的非冗余蛋白质序列数据库;UniPrc作为数据仓库,再分别给UniProtKB,Proteomes,UNIRef提供可靠的数据集,其中在UniProtKB数据库中Swiss-Prot是由TrEMBL经过手动注释后得到的高质量非冗余数据库,也是我们最常用的蛋白质数据库之一。
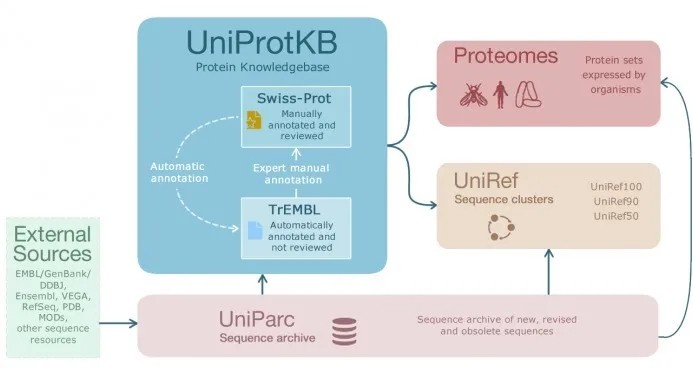
Uniprot数据库官方链接:https://www.uniprot.org/
Uniprot数据的使用
1. 单个蛋白质信息查询
下图是Uniprot官方网站首页,在UniprotKB栏输入蛋白ID或Accession number,然后点击search,就可以查询蛋白功能。
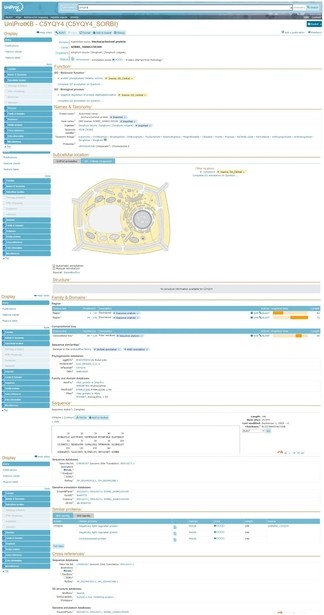
 我们以高粱基因SORBI_3008G150300为例,搜索其在Uniprot数据库中的信息,如下图,页面默认显示Entry模式,页面显示内容包括:蛋白名称、物种来源、GO功能注释、亚细胞定位、组织特异性表达情况、互作蛋白、Domain、序列信息、同源蛋白以及其他数据链接等信息。
我们以高粱基因SORBI_3008G150300为例,搜索其在Uniprot数据库中的信息,如下图,页面默认显示Entry模式,页面显示内容包括:蛋白名称、物种来源、GO功能注释、亚细胞定位、组织特异性表达情况、互作蛋白、Domain、序列信息、同源蛋白以及其他数据链接等信息。
 点击Display下Publications模式,数据库会展示处该蛋白涉及的收录的文章。
点击Display下Publications模式,数据库会展示处该蛋白涉及的收录的文章。
 点击Display下Feature viewer模式,数据库会图形展示结构域与序列信息。
点击Display下Feature viewer模式,数据库会图形展示结构域与序列信息。
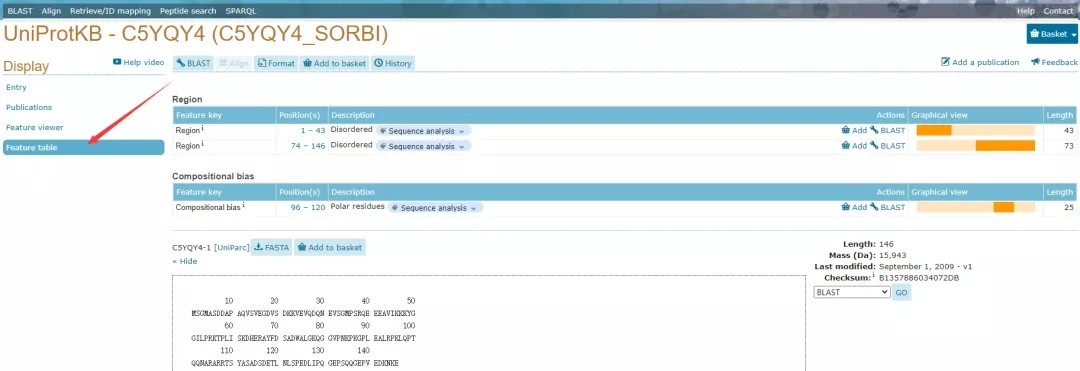
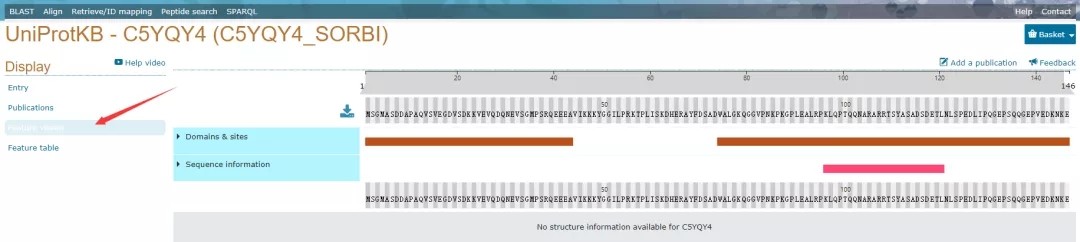 点击Display下Feature table模式,数据库会展示结构域位置、分子量、序列长度等基本信息。
点击Display下Feature table模式,数据库会展示结构域位置、分子量、序列长度等基本信息。
2. 批量蛋白质信息查询
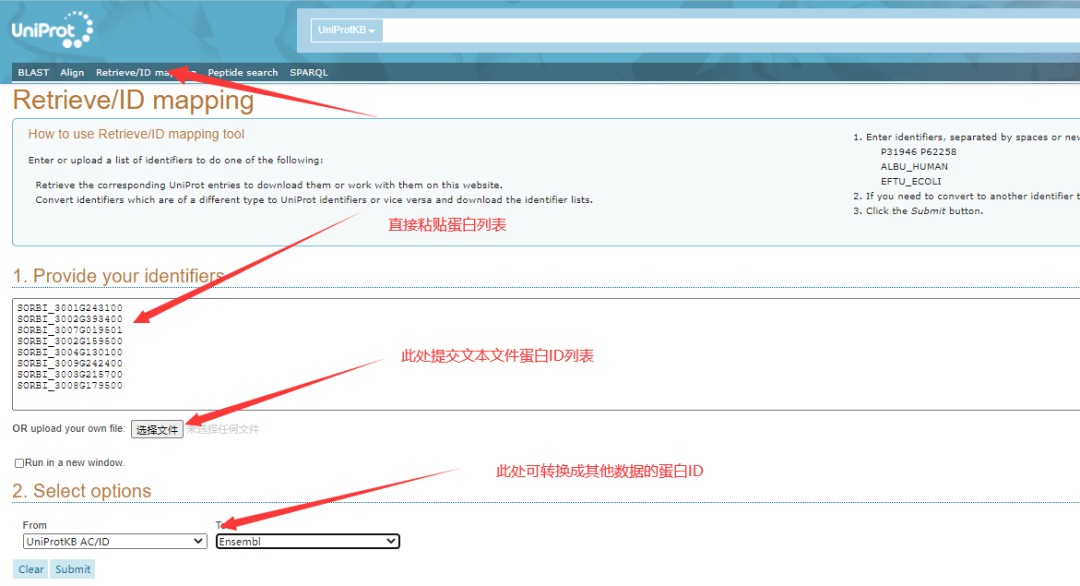
假如需要查询的蛋白较多,则可以通过点击首行任务栏Retrieve/ID mapping,如下图,查询蛋白列表可直接粘贴在下图1. Provide your identifiers文本框中,也可以将蛋白ID单列粘贴于TXT文本中提交到网站。另外该页面2. Select options 还可提供ID转换功能,支持多种数据库间的ID转换。
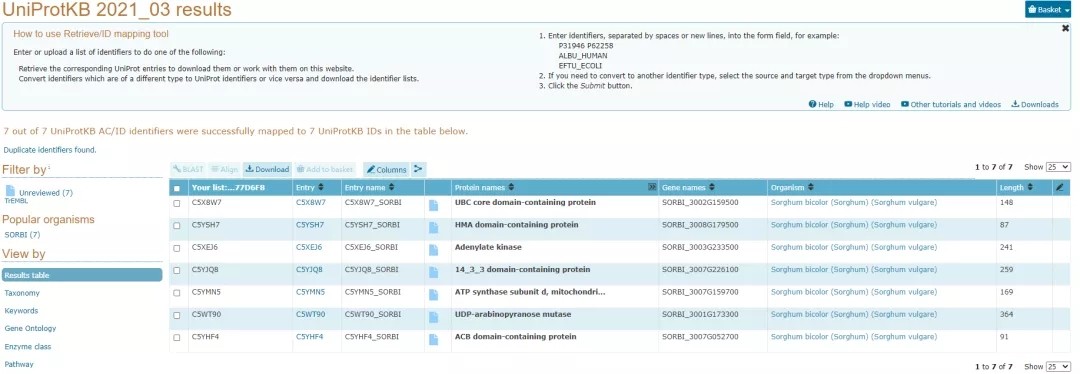
提交好蛋白列表后,点击Submit,网站便会自动分析,结果展现形式如下:
展示信息包括:蛋白对应的基因名、蛋白描述、序列长度等信息。
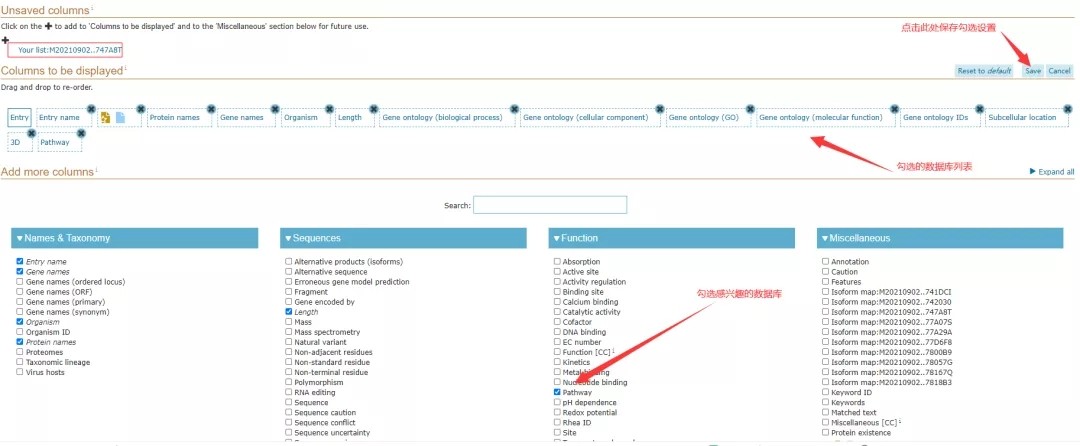
 点击Column按钮,可以选择需要展示的数据库信息,如GO、pathway、亚细胞定位等注释信息,如下图,选择完毕后点击save保存设置,系统会自动跳转至信息展示页面。
点击Column按钮,可以选择需要展示的数据库信息,如GO、pathway、亚细胞定位等注释信息,如下图,选择完毕后点击save保存设置,系统会自动跳转至信息展示页面。
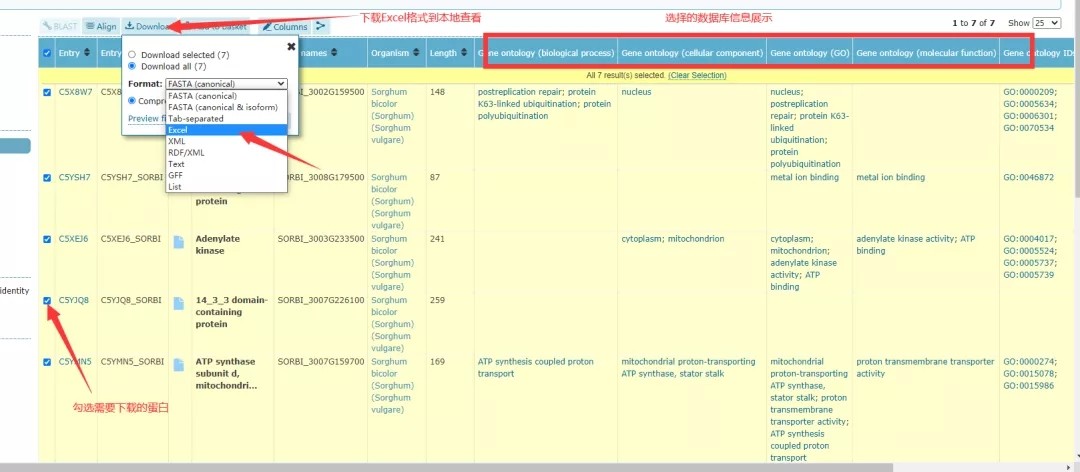
 最终结果展示如下图,勾选感兴趣的蛋白,即可将本次注释结果下载到本地查看,并且支持包括Excel格式在内的多种文本格式。
最终结果展示如下图,勾选感兴趣的蛋白,即可将本次注释结果下载到本地查看,并且支持包括Excel格式在内的多种文本格式。
 好了,今天Uniprot数据库的使用就介绍到这里,希望对您的科研有所帮助!
好了,今天Uniprot数据库的使用就介绍到这里,希望对您的科研有所帮助!
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,又能发文章,又能学生信技能,一举两得的SCI好思路;基因家族分析课程已更新分析内容,最新版课程学习链接:基因家族分析实操课程
2. 转录组数据结果理解不深入?图表看不懂?这就是你转录组数据不会挖掘、文章不会撰写的原因,让我带你一起深入了解转录组数据结果,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?多学点数据处理技能:学习链接:转录组标准分析后的数据挖掘
5.微生物多样性分析很简单,但是分析内容项目并不少,理解起来有困难?看我深入浅出讲给你听,学习链接: 微生物16S/ITS/18S分析原理及结果解读
6. 学生物的必学生信技能:Docker安装与使用、linux系统入门、Perl语言入门到精通、Python生信入门到精通、perl语言高级编程
7. 生信绘图、科研绘图技能:Cytoscape与网络图绘制、微生物OTU网络图绘制(Cytoscape)、R语言基础与绘图、R语言绘图基础(ggplot2)
8.生物信息实战技能,0基础也可以学会,内含脚本及demo数据:RNAseq有参转录组自主分析、基因组重测序自主分析、微生物多样性自主分析
9. 更多学习内容:linux、perl、R语言画图,更多免费课程请扫描下方二维码进入组学大讲堂网校学习:
- 发表于 2022-01-19 22:26
- 阅读 ( 36260 )
- 分类:软件工具

