ascp 高速下载NCBI各种数据库中的数据(SRA NR NT 分类数据库,)
ascp快速下载NCBI各种数据库种的数据
NR NT 数据库:
#wget -c https://ftp.ncbi.nih.gov/pub/taxonomy/taxdump.tar.gz
#wget -c https://ftp.ncbi.nlm.nih.gov/genbank/livelists/gi2acc_mapping/gi2acc_lmdb.db.gz
#wget -c https://ftp.ncbi.nlm.nih.gov/genbank/livelists/gi2acc_mapping/gi2accession.py
#wget -c https://ftp.ncbi.nih.gov/blast/db/FASTA/nr.gz
#wget -c https://ftp.ncbi.nih.gov/pub/taxonomy/accession2taxid/prot.accession2taxid.gz
#快速下载方法
/share/work/biosoft/aspera/latest/cli/bin/ascp -v -k 1 -T -l 400m -i ~/asperaweb_id_dsa.openssh anonftp@ftp.ncbi.nlm.nih.gov:/genbank/livelists/gi2acc_mapping/gi2acc_lmdb.db.gz ./
/share/work/biosoft/aspera/latest/cli/bin/ascp -v -k 1 -T -l 400m -i ~/asperaweb_id_dsa.openssh anonftp@ftp.ncbi.nlm.nih.gov:/pub/taxonomy/accession2taxid/prot.accession2taxid.gz ./
/share/work/biosoft/aspera/latest/cli/bin/ascp -v -k 1 -T -l 400m -i ~/asperaweb_id_dsa.openssh anonftp@ftp.ncbi.nlm.nih.gov:/blast/db/FASTA/nr.gz ./
sra数据库可以利用ascp ENA网站搜索下载:
ENA主页ENA
搜索SRR号,SRR9169172
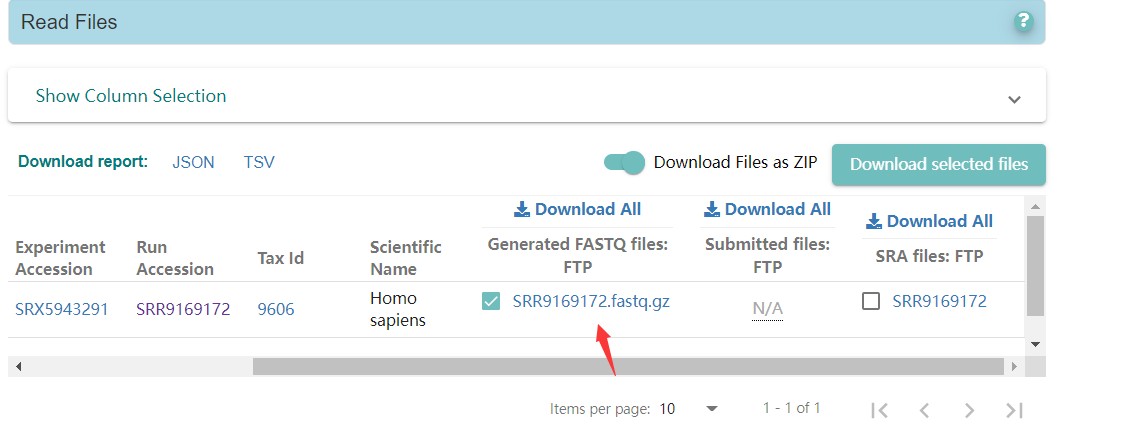
命令:
/share/work/biosoft/aspera/latest/cli/bin/ascp -QT -l 300m -P33001 -i ~/asperaweb_id_dsa.openssh era-fasp@fasp.sra.ebi.ac.uk:/vol1/fastq/SRR916/002/SRR9169172/SRR9169172.fastq.gz ./
参数说明:
-v verbose mode 唠叨模式,能让你实时知道程序在干啥,方便查错。
-T 取消加密,否则有时候数据下载不了
-i 提供私钥文件的地址,免密从SRA和ENA下载,不能少,地址一般是:/share/work/biosoft/aspera/latest/cli/etc/asperaweb_id_dsa.openssh
-l 设置最大传输速度,一般200m到500m,如果不设置,反而速度会比较低,可能有个较低的默认值
-k 断点续传,一般设置为值1
-Q 用于自适应流量控制,磁盘限制所需
-P 用于SSH身份验证的TCP端口,一般是33001
-T 取消加密,否则有时候数据下载不了
-i 提供私钥文件的地址,免密从SRA和ENA下载,不能少,地址一般是:/share/work/biosoft/aspera/latest/cli/etc/asperaweb_id_dsa.openssh
-l 设置最大传输速度,一般200m到500m,如果不设置,反而速度会比较低,可能有个较低的默认值
-k 断点续传,一般设置为值1
-Q 用于自适应流量控制,磁盘限制所需
-P 用于SSH身份验证的TCP端口,一般是33001
- 发表于 2022-01-24 13:27
- 阅读 ( 5549 )
- 分类:其他
你可能感兴趣的文章
- 探索基因大数据的利器——SRA Explorer 2543 浏览
- NCBI 基因组文件下载——datasets 2468 浏览
- 使用NCBI——Batch Entrez批量下载蛋白序列 12058 浏览
- NCBI的SRA Lite和SRA Normalized数据有什么区别 7742 浏览
- NCBI下载SRR并转换为fastq文件 12721 浏览
- NCBI下载物种叶绿体或线粒体基因组fasta,gff3,gtf,gb文件等 10728 浏览
相关问题
- SRA下载 1 回答
- SRA数据下载 2 回答
- 请问老师怎么将NCBI里的gbff文件转化为gff格式的文件,因为里面找不到gff文件 0 回答
- 利用命令行进行sra数据下载有如下报错 1 回答
- NCBI序列上传 1 回答
- sra文件转换为fastq文件 1 回答
0 条评论
请先 登录 后评论
