Monocle2|单细胞测序的拟时序分析
Monocle2|单细胞测序的拟时序分析
什么是拟时序分析?拟时序(pseudotime)分析,又称细胞轨迹(cell trajectory)分析。通过拟时序分析可以推断出发育过程中细胞的分化轨迹或细胞亚型的演化过程。我们可以理解为在一堆细胞中包含各种各样不同的发育状态的细胞,有的发育早,有的发育晚,有的分化了,有的未分化,有的处于中间态。利用算法基于基因表达推断每个细胞的相对分化时间,从而确定分化轨迹。Monocle是拟时序分析常用的R包。
文章案例
例如2021年5月发表在NC的一篇单细胞测序文章《Single-cell profiling of tumor heterogeneity and the microenvironment in advanced non-small cell lung cancer》(IF=14.92)就多次采用拟时序分析,探讨不同细胞亚型之间的分化关系。
 拟时序分析
拟时序分析为了方便大家能高效快捷的对单细胞数据进行拟时序分析,我们给大家提供一个R脚本,这个脚本只需要准备好相应的输入文件,再进行简单的命令行操作即可进行拟时序分析并绘制可直接用于文章发表的图片。
Rscript single_cell/cell_trajectory.r -i data/pbmc_ctrl.rds --downsample 100 -o trajectory_result/pbmc_ctrl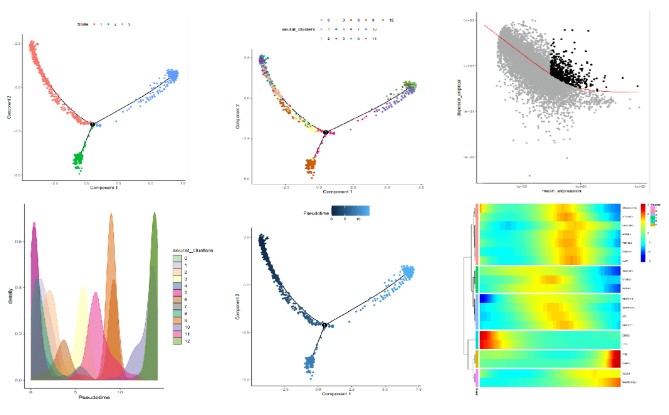 输入文件准备
输入文件准备这个脚本所必需的输入文件只有一个,即经聚类注释后的seurat对象,为rds文件。
更多脚本参数设置及说明
可以通过-h参数获得以下帮助信息:
usage: single_cell/cell_trajectory.r [-h] -i rds_data [--assay assay]
[--Customization]
[--marker.genes marker.genes]
[--gene.list filepath]
[--downsample downsample] [-g group]
[-d density.group] [-n heatmap.gene]
[-c heatmap.clusters] [-H height]
[-W width] [-o path] [-p prefix]
single cell trajectory analysis : http://cole-trapnell-lab.github.io/monocle-
release/docs/#constructing-single-cell-trajectories
optional arguments:
-h, --help show this help message and exit
-i rds_data, --rds_data rds_data
input rds_data file path[required]
--assay assay Name of assay to use, defaults to the active assay
[default RNA]
--Customization If set, use Customization [optional, default: False]
--marker.genes marker.genes
Select the method to choose genes that define a cell's
progress. Available methods are:monocle,seurat,deg
[default monocle]
--gene.list filepath input the file of genes that define a cell's progress
[default NULL]
--downsample downsample
subset cells numbers for analysis [default None]
-g group, --group group
the group that coloring the cells by [default
seurat_clusters]
-d density.group, --density.group density.group
the density group that coloring density map [default
seurat_clusters]
-n heatmap.gene, --heatmap.gene heatmap.gene
the number of marker genes used to draw heatmap
[default 20]
-c heatmap.clusters, --heatmap.clusters heatmap.clusters
Number of clusters for the heatmap of branch genes
[default 6]
-H height, --height height
the height of pic inches [default 7]
-W width, --width width
the width of pic inches [default 6]
-o path, --outdir path
output file directory [default
/share/nas5/zhangll/self/single_cell]
-p prefix, --prefix prefix
out file name prefix [default demo]
必需参数
-i 经聚类注释后的seurat对象,为rds文件
其他参数
--assay 指定seurat对象中的assay,默认为RNA
--Customization 自定义特征基因,默认为False;为Ture时需添加--gene.list参数
--marker.genes 选择获取特征基因的方式,默认为monocle选择的高变 基因,可选方式包括monocle、seurat、deg
--gene.list 指定特征基因
--downsample 是否减少样本量,默认为False
-g 拟时序散点图的着色分组,默认按照seurat_clusters着色
-d 细胞密度图的着色分组,默认按照seurat_clusters着色
-n 指定用于绘制热图的特征基因个数,默认为20
-c 热图基因聚类个数,默认为6
-H 输出图片尺寸(高),默认为7英寸
-W 输出图片尺寸(高),默认为7英寸
-o 输出路径,默认为当前路径
-p 输出文件名,默认为demo
脚本获取方法
为了方便感兴趣的学员小伙伴想复现这个分析内容,我把数据和代码一并打包,免费供大家使用学习。只需要如下几步,即可获得下载链接。
1、关注生信博士公众号;
2、转发本文章至朋友圈,并截图;
3、在公众号菜单栏回复朋友圈截图和cell_trajectory即可获得下载链接。
如果没有数据分析的基础,不知道从何下手,可以学习一下我们的《医学数据分析环境搭建》视频课程,扫描下方二维码了解课程详情。

- 发表于 2022-06-10 13:30
- 阅读 ( 9475 )
- 分类:转录组
你可能感兴趣的文章
- SCP 安装成功的单细胞转录组 docker镜像使用 single-cell-v5 144 浏览
- 单细胞转录组数据标准化: 878 浏览
- 植物单细胞转录组文章 1371 浏览
- 单细胞分析绘制marker基因展示图时如何设置细胞排布顺序 1858 浏览
- monocle3分析结果解读 8867 浏览
相关问题
- 提供感兴趣基因进行分支分析时,出现Error in simple_vs_index(x, rlang::eval_tidy(args[[1]]), na_ok) : Unknown vertex selected报错 1 回答
- 轨迹分析出现killed报错 1 回答
- 老师,图1是我用图2的代码实现的,现在我想仿照提取细胞亚类的方式,使用图3的代码单独提取TIP这个maker基因,不提取整个cluster3,这个代码可以实现吗,或者是否有其他代码可以使用 1 回答
- 老师,拟时序基础初步分析时,运行以下代码时Rscript $scripts/cell_trajectory_monocle3.r --rds ../04.cell_type_ann_leaf/leaf.added.celltype.qs \ --use.seurat.umap --groupby mycelltype -o monocle3.1 --cpu 6,出现图中报错,是因为聚类分群的时候resolution值太低了吗 2 回答
- 拟时分析筛选出的差异基因数量太少,有没有可以调整的参数去增加筛选数量 1 回答
- 单细胞测序使用subset脚本提出子集,再进行拟时分析报错plot_genes_by_group 1 回答
0 条评论
请先 登录 后评论

