区分cds utr.exon
英文版的基因结构图:
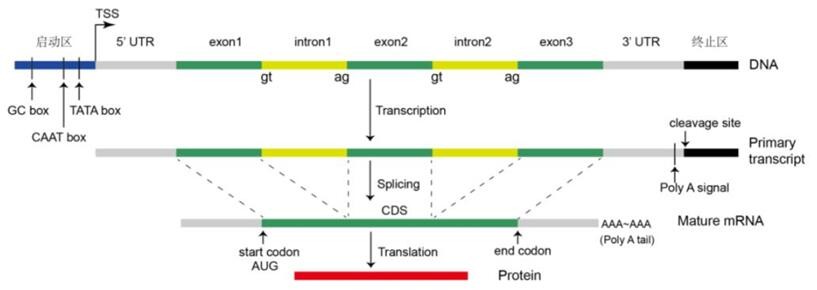
中文版的基因结构图:
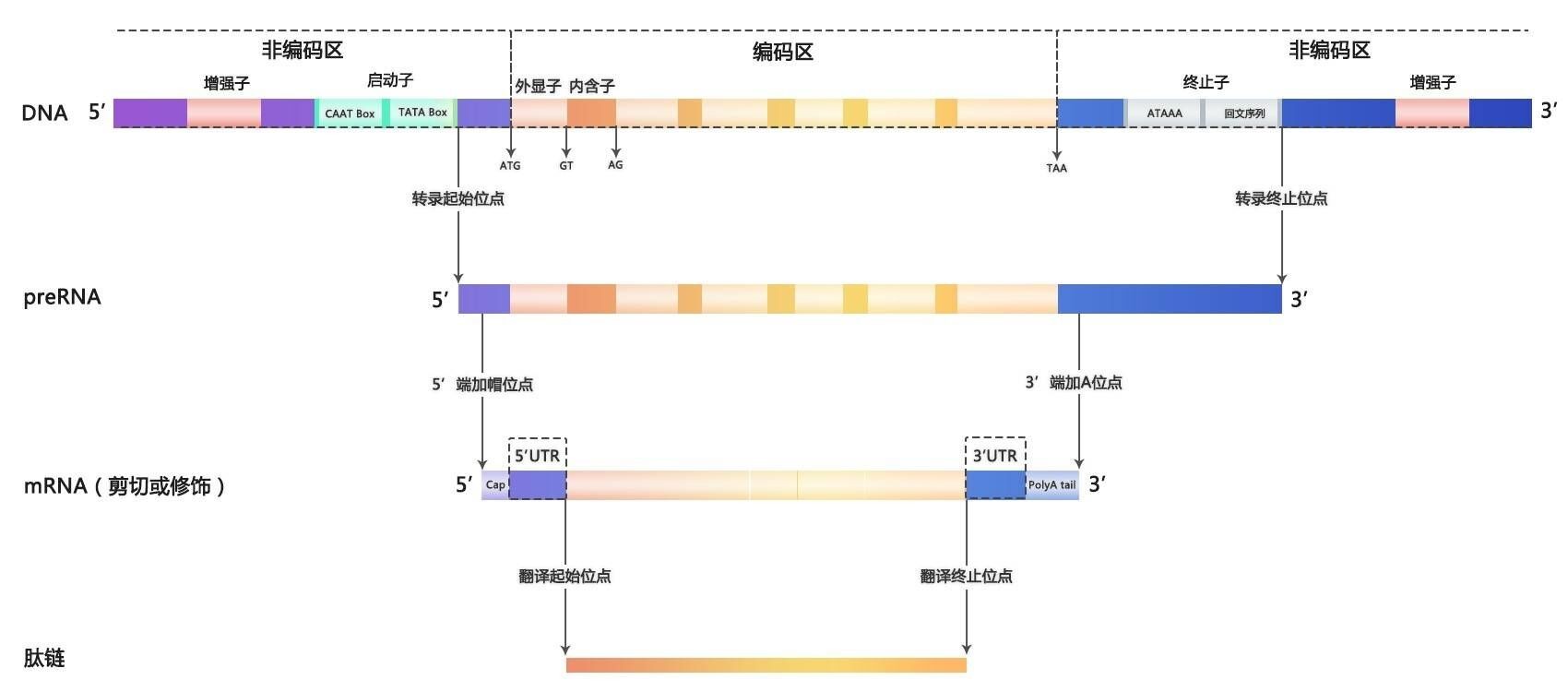
先来认识几个概念:
3'UTR: 3‘端非翻译区,也就是mRNA两端的非编码片段
5'UTR: 5‘端非翻译区,也就是mRNA两端的非编码片段
UTR代表了RNA EXON的非蛋白质编码部分
ORF:开放读取框,指任意一段序列,只要起于ATG止于终止子,都可以叫做ORF。ORF是一种预测,而不是一种已知的翻译区
CDS: Coding DNA Sequence,是指mRNA序列中编码蛋白质的那部分序列。属于ORF(open reading frame),既然编码蛋白,那肯定以ATG开始--终止密码子结束。
CDS,start_codon(启动子),stop_codon(终止子),UTR等概念都是针对可以编码蛋白质的转录本(mRNA)而言的。当然也存在不编码蛋白的转录本(mRNA),那么他只有exon了,没有CDS,start_codon,stop_codon。另外exon与intron是互斥的,exon包含UTR和CDS。
基于这些信息我们可以得到以下结论 (可以自行统计基因注释文件:gencode.v18.annotation.gtf):
1). 对start_codon和stop_codon而言,只涉及3个碱基。以ENST00000583352.1 为例 (GENCODE hg19),其中涉及以下两行信息(基因组位置后部分信息已省去)
chr17 HAVANA start_codon 46051392 46051394
chr17 HAVANA stop_codon 46053014 46053016
2). 对于任意一个编码蛋白转录本而言,可以没有5'UTR,但是肯定有3’UTR,3'UTR最短也就和stop_codon完全相同,即3个碱基。 以ENST00000583352.1 为例 (GENCODE hg19),其中涉及以下两行信息(基因组位置后部分信息已省去)
chr17 HAVANA stop_codon 46053014 46053016
chr17 HAVANA UTR 46053014 46053016
结论:可以看出此转录本stop_codon和UTR基因组位置相同。
3). 对于编码蛋白转录本而言,第一个外显子可以部分或者整体全是在5’UTR内部。此外,前面多个外显子也可以都在5’UTR中。以ENST00000342066.3为例(GENCODE hg19),其中涉及以下信息(基因组位置后部分信息已省去)
chr1 HAVANA exon 861118 861180
chr1 HAVANA exon 861302 861393
chr1 HAVANA CDS 861322 861393
chr1 HAVANA start_codon 861322 861324
...
chr1 HAVANA UTR 861118 861180
chr1 HAVANA UTR 861302 861321
chr1 HAVANA UTR 879531 879955
结论:可以看出此转录本 第一个外显子在5'UTR内部,第二个外显子含有部分5'UTR。start_codon是CDS的一部分
4). 部分编码蛋白转录本最后一个外显子可以部分或者整体全是在3’UTR内部。此外,后面多个外显子也可以都在3’UTR中。以ENST00000435070.3为例(GENCODE hg19),其中涉及以下信息(基因组位置后部分信息已省去)
chr12 HAVANA CDS 69656153 69656336
chr12 HAVANA stop_codon 69656337 69656339
chr12 HAVANA exon 69663292 69668138
chr12 HAVANA UTR 69633317 69633426
chr12 HAVANA UTR 69656337 69656342
chr12 HAVANA UTR 69663292 69668138
结论:可以看出此转录本最后一个外显子在3'UTR内部,倒数第二个外显子含有部分3'UTR。stop_codon是3’UTR的一部分。
5. 可以是2),3)和4)的组合。
- 发表于 2022-09-26 11:58
- 阅读 ( 4874 )
- 分类:基础知识
