组学大讲堂QIIME2(v1.5)镜像更新说明
QIIME2是Rob Knight实验室和Greg Caporaso实验室(QIIME1文章的第一作者,现在拥有独立实验室)共同开发的一款跨时代革命性的全新扩增子微生物组分析流程。QIIME2不是QIIME1的一个简单升级,它是一次采用python3完全的重写,不仅支持聚类方法生成OTUtable,还支持最新的降噪方法生成feature table。QIIME2是一个综合的分析工具,它将是未来扩增子分析的绝对分析标准。
我们组学大讲堂提供的扩增子分析docker镜像(v1.4)使用的qiime2 版本为:Qiime2.2020.8版本。目前qiime2的最新版本为2022.8,相较于2020.8版本有很多大的更新,更新信息如下: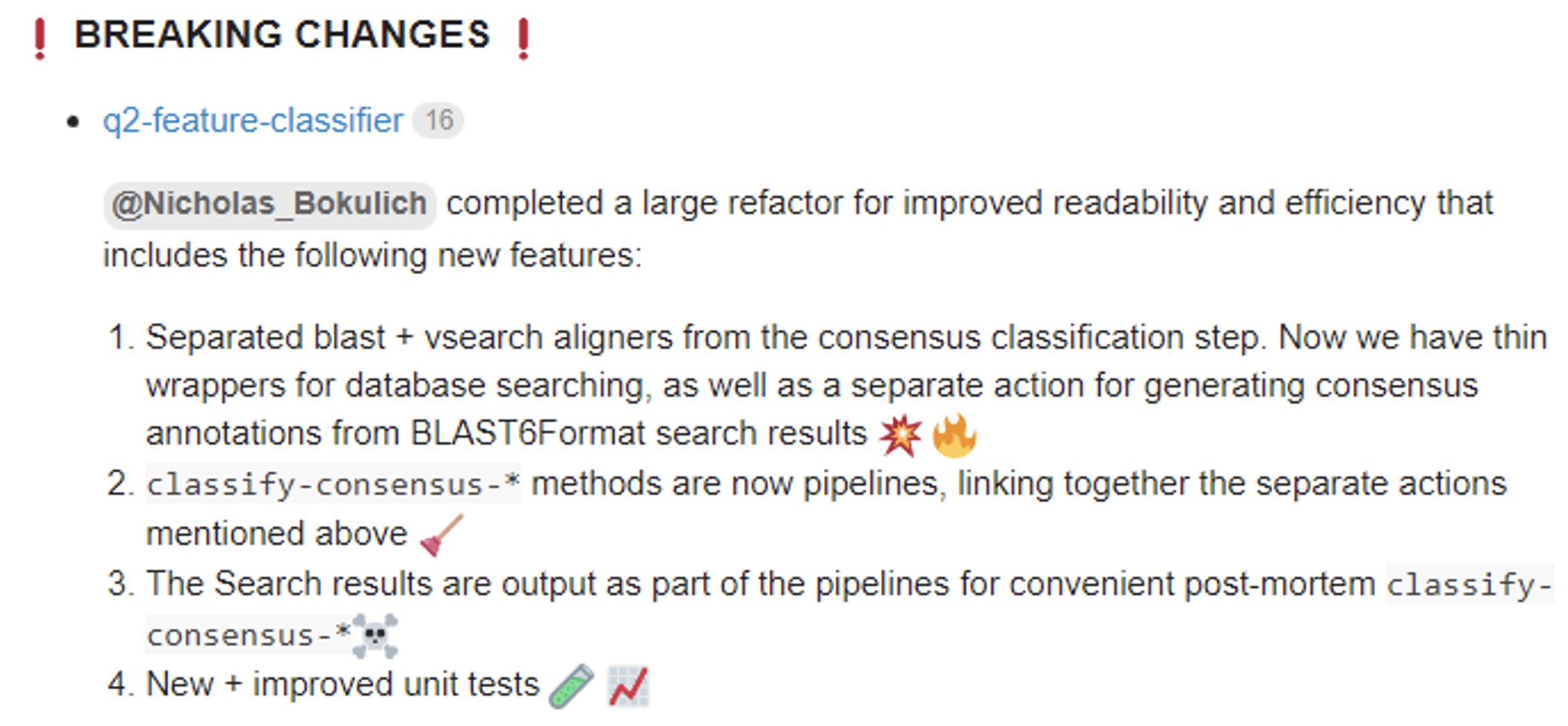
扩增子docker镜像更新说明
为使大家使用上最新的qiime2软件,我们也对我们的扩增子流程中的qiime2做了更新,更新到了最新的2022.8这个版本。另外,我们也对qiime2的物种注释分类数据库进行了同步更新以避免数据库与scikit-learn版本不一致时的报错,因此,老版本的qiime2分类数据库和新版的qiime2分类数据库不通用;同时我们也更新了真菌数据库的版本:真菌unite数据库的版本则从8.99升级到了9.99,silva数据库的版本官方没有更新,依然是138。对于以上更新,我们用测试数据集都进行了测试,代码测试一切顺利,大家可以放心使用;这次qimme2版本更新我们放到omicsclass/ampliseq-q2:v1.5版本的扩增子分析docker镜像中,下载及使用方式如下:
#下载多样性分析docker镜像
docker pull omicsclass/ampliseq-q2:v1.5
#启动docker容器并交互式进入
docker run --rm -it -m 20G --cpus 2 -v D:\qiime2-16s:/work omicsclass/ampliseq-q2:v1.5
数据库更新说明
在我们分享的百度网盘资源中有如下4个文件,其中《更新说明.txt》文件为对于本次更新的描述,data.tar.gz和amplicon_demo.tar.gz 基本未做改动(amplicon_demo中的work.sh文件,对应本次更新修改了数据库路径部分)
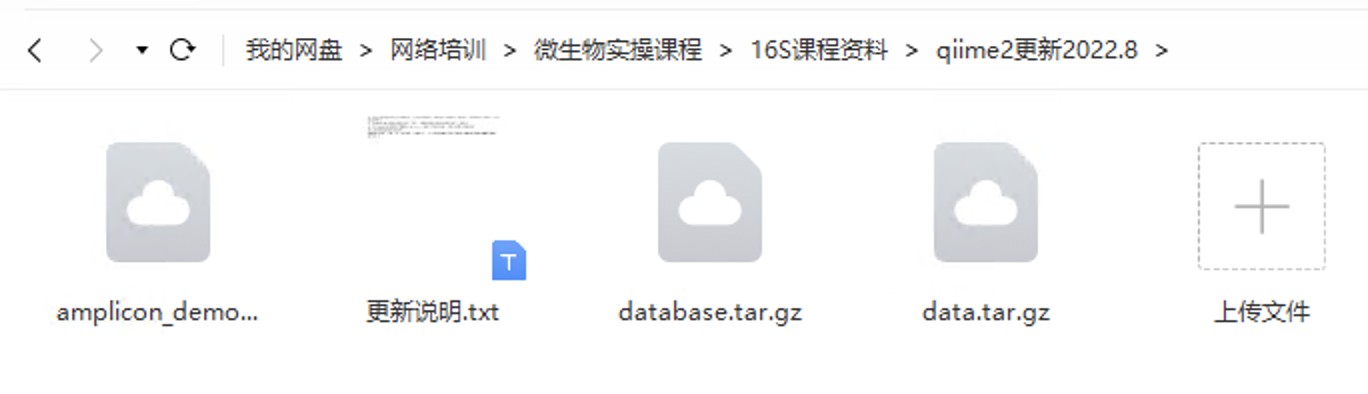
database.tar.gz的文件解压后即可得到更新后的分类数据库。为了使文件夹更简洁,我们将各个数据库单独放置
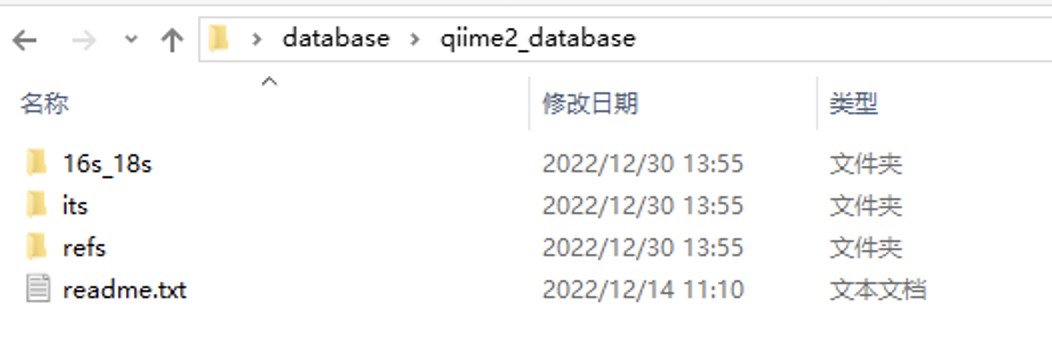
文件夹refs:为降噪参考数据库,是从官网直接下载并转换的数据库
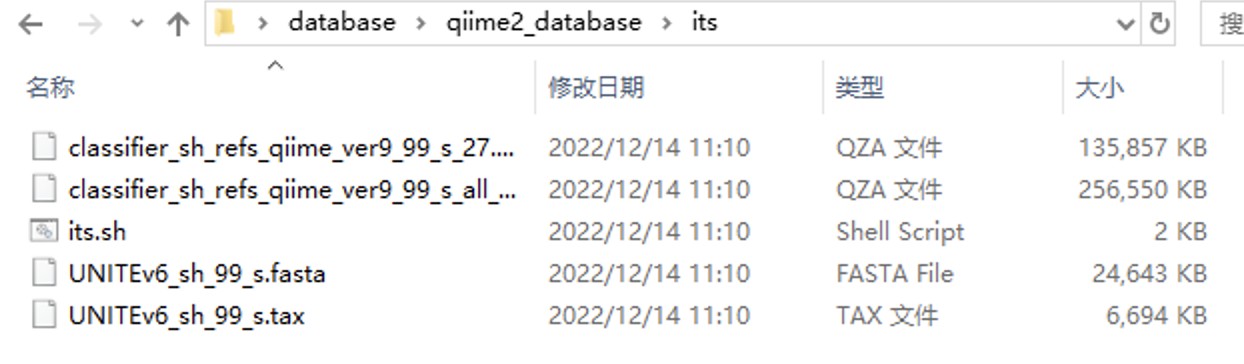
文件夹its:为用于真菌的分类数据库,其中classifier开头的两个文件分别为qiime2官网最新的数据库(带all的用于所有真核生物,不带all的仅限真菌)可直接使用。Unite开头的两个文件为之前官网下载的数据,可以使用its.sh里的命令自己构建数据库,官网暂时无法进入,开放后可以自行下载最新的数据 https://unite.ut.ee
文件夹16s_18s:考虑到目前16s最常用的数据库也支持18s的注释,我们便将他们整合到了一起放到了16s_18s文件夹,其中分别是官网直接下载的silva和greengene的数据库,注意,只有sliva的数据可同时用于16s及18s。基于greengene构建的数据库只能用于16s数据分析
而my_classifier中的文件则是特异引物构建的物种分类数据库,我们从官网下载数据并自己构建的数据库,原始数据已删除,如有需要可以在各自的.sh文件中使用wget下载,您同样也可以通过两个.sh后缀的脚本自己构建各自的数据库
picrust2更新
此外,我们将picrust2从2.3.0版本升级到了最新的2.5.1版本,他解决了此前可能存在的运行报错的问题,更新内容如下

经过测试,新版PICRUSt2拥有更快的运行速度且更为稳定。
详细的更新说明请见官方文档 https://github.com/picrust/picrust2/releases
视频课程
扫描下方二维码进入组学大讲堂网校首页:

注:1. 本系列课程为录播课程,已经全部制作完成,购买后即可观看全部内容;2. 购买之后可以通过手机或者电脑访问study.omicsclass.com,登录账号进行学习;3. 购买或者观看遇到问题,可以加微信“532812110”联系我们。
延伸阅读:
1. 微生物测序原理2. 肠道“君”与人类二三事(科普)3. 什么是OTU4. alpha多样性5. GraPhlAn树状图6. OTU网络图MENA7. beta多样性解读
8. 一文读懂微生物多样性中的各种排序分析方法异同点9. 微生物组间差异分析之LEfSe分析10. 用测序研究微生物多样性,您选对区了吗?11.微生物多样性分析docker镜像使用12.生成OTU方法如何选择,去噪还是聚类?13.微生物多样性组间差异分析—STAMP使用!
14.16SrDNA你知道多少?15.机器学习预测土壤枯萎病

- 发表于 2023-01-06 13:26
- 阅读 ( 3251 )
- 分类:宏基因组
你可能感兴趣的文章
- 微生物多样性扩增子(16S/18S/ITS)课程更新v2.0 1778 浏览
- qiime2 q2-picrust2 报错问题 1222 浏览
- qiime2分类器的训练 2889 浏览
- qiime2 安装-使用国内镜像避免网络原因报错: 3147 浏览
- 利用qiime2对微生物扩增子测序数据文库根据barcode信息拆分数据 9229 浏览
