OrthoFinder 输出结果文件说明
OrthoFinder 工作流程
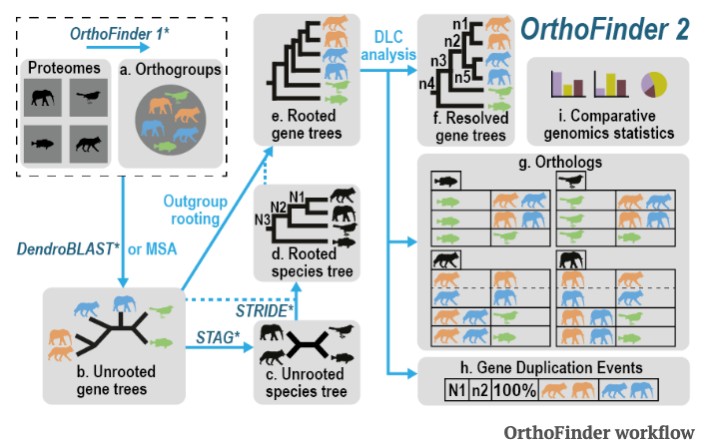
使用 DIAMOND 软件 对输入序列进行 all-vs-all 序列比对。
使用 MCL(Markov Cluster Algorithm)算法 根据比对结果进行聚类,得到 直系同源组(orthogroup)。其中每个 orthogroup 的蛋白及序列信息存放在 Orthogroup_Sequences 文件夹中,单拷贝 orthogroup 的蛋白及序列信息存放在 Single_Copy_Orthologue_Sequences 文件夹中,orthogroup 的统计信息存放在 Comparative_Genomics_Statistics、Orthogroups 文件夹中。
如 OrthoFinder 自带案例(ExampleData)中总共包含 2733 个基因,MCL 将 2202 个基因划分为 604 个 orthogroups(gene_num > 2),剩余 531 个基因为离散点(每个基因独立成组)。
使用 FastTree 软件 对每个 orthogroup(gene_num >= species_num)构建 无根基因树(gene tree)。如自带案例中总共生成 324 个基因树文件。
使用 STAG(Species Tree Inference from All Genes)软件 根据 orthogroups(包含所有物种,如自带案例推断出的 604 个 orthogroups 中只有 316 个 orthogroups 中的同源基因在所有物种中均有分布)推断 无根物种树(species tree)。
通过参数 -M dendroblast 或 -M msa,OrthoFinder 可以调用 STAG 中两种构建物种树的方法:DendroBLAST(默认) 和 CMSA(Concatenated Multiple Sequence Alignment,联合多序列比对)。
使用 STRIDE(Species Tree Root Inference from Gene Duplication Events) 通过基因复制事件的不可逆性为无根物种树、无根基因树赋根,得到有根物种树、有根基因树、基因间的直系同源关系、基因复制事件。结果存放在文件夹 Species_Tree、Gene_Tree、Orthologues、Gene_Duplication_Events、Comparative_Genomics_Statistics 中。
OrthoFinder 输出文件说明
OrthoFinder 的标准输出包括:直系同源组,直系同源基因,有根基因树,解析基因树,无根物种树、有根物种树,基因重复事件以及相关的统计数据。
1. Orthogroups 文件夹
Orthogroups.tsv、Orthogroups.txt:记录了 MCL 中 成功聚类(直系同源组中基因数 >= 2)的每个 Orthogroup 所包含的基因。
Orthogroups_UnassignedGenes.tsv:记录了 MCL 中 未成功聚类(直系同源组中基因数 >= 1)的离散基因。
Orthogroups.GeneCount.tsv:记录了每个 Orthogroup 中基因在物种间的分布情况,可以用于分析同源基因在物种间的收缩和扩张。
Orthogroups_SingleCopyOrthologues.txt:记录了 单拷贝直系同源组。
2. Orthogroup_Sequences 文件夹
均为 FASTA 格式文件,记录了每个 Orthogroup 所包含的基因 / 蛋白的序列信息
3. Single_Copy_Orthologue_Sequences 文件夹
均为 FASTA 格式文件,记录了每个单拷贝 Orthogroup 所包含的基因 / 蛋白的序列信息
4. Phylogenetic_Hierarchical_Orthogroups 文件夹
由于复制本在进化之间存在突变速率的异质性,所以在研究同源基因时更希望所研究的同源基因来自相同的复制本。Hierarchical Orthogroups(HOG)就是为这一目的而设立的概念,HOG 指由最近共同祖先中某一基因进化而来的一组直系同源基因,进化过程中不涉及基因复制,所以 HOG 中不包含旁系同源。如下图中红框所标注的 Orthogroup。
OrthoFinder 以物种树中的节点(LCA)为标准,寻找有根基因树内由 LCA 中基因进化来的 HOG,对原先 MCL 算法得到 orthogroup 进行细分。输出文件 N0.tsv,N1.txt,N2.tsv,… 分别指以物种树 N0,N1,N2,… 节点为标准推断出的 HOG。
5. MultipleSequenceAlignments 文件夹
此文件夹仅在 -M msa 模式下输出,均为 FASTA 格式文件。
记录了每个 orthogroup 中序列间的多序列比对结果。
记录了程序通过 CMSA 算法过滤后的 orthogroup 中各序列串联后的多序列比对结果,同时比对结果中空位数 > 50% 的列已被删除。
6. Species_Tree 文件夹
SpeciesTree_rooted.txt:STAG、STRIDE 算法计算出的有根物种树结构。
SpeciesTree_rooted_node_labels.txt:相比上树在节点处具有标签(N 0 , N 1 , . . . , N m ),让后续的分析中可以方便的指定物种树节点。
Orthogroups_for_concatenated_alignment.txt:仅在 -M msa 模式下输出,列出了所有串联起来用于推断物种树的 orthogroup ID。
7. Gene_Trees 文件夹
记录了每个 orthogroup(gene_num >= 4)的有根基因树结构。
8. Gene_Duplication_Events 文件夹
注意!OrthoFinder 只统计记录支持值(Support) >= 50% 的的复制事件。支持值是指复制后两个基因副本未被丢失的比例,Support >= 50% 表示复制后至少有一半基因在演化中保留了下来。
Duplications.tsv:记录了程序推测出的所有基因复制事件的信息。其中 Species Tree Node 表示基因复制事件发生时所对应的物种树节点(即复制是在该物种内发生的);Gene tree node 表示基因复制事件发生时所对应的基因树节点与基因复制事件对应的节点;Support 表示复制后两个基因副本未被丢失的比例;Type 中 Terminal 表示重复发生在物种树的末端分支上,Non-Terminal 表示重复发生在物种树的内部分支上,被多个物种共享;Genes 1、Genes 2 为基因列表,其中 Genes 1 表示来自复制后基因的一个副本;Genes 2 表示来自复制后基因的另一个副本。
SpeciesTree_Gene_Duplications_0.5_Support.txt:记录了物种树每个节点、分枝上包含的基因复制事件的总和,格式为节点或物种名 + 数字(基因复制事件数量)。
9. Orthologues 文件夹
以物种为单位,记录了每个物种与其他物种间的直系同源基因。
10. Comparative_Genomics_Statistics 文件夹
Statistics_Overall.tsv:记录了有关 orthogroup 的常规统计信息。
Statistics_PerSpecies.tsv:以物种为单位,记录了有关 orthogroup 的常规统计信息。
Orthogroups_SpeciesOverlaps.tsv:记录了每个物种对之间共享的 orthogroup 数。
Duplications_per_Species_Tree_Node.tsv:记录了物种树中每个节点、物种中发生基因重复事件的数量。
Duplications_per_Orthogroup.tsv:记录了每个 orthogroup 中推断出的基因重复事件数量。
OrthologuesStats _ *:记录了每对物种之间一对一、一对多和多对多关系的直向同源物数量。
11. WorkingDirectory 文件夹
OrthoFinder 运行所需的必须中间文件, 如 DIAMOND 比对结果,STAG 输出的无根物种树等。
- 发表于 2023-03-13 14:12
- 阅读 ( 5534 )
- 分类:基因组学
