TCGA和GEO套路文章解说
文章,重在研究思路。有好的思路,才能有精彩的故事。很多时候,我们想找一个好点子,比如性状,疾病相关的关键分子,需要设计实验,做大量的分子筛选的工作,对于土豪实验室可以可以大把的花钱做芯片或者高通量测序做前期的筛选工作。但是如果你所处的实验室或者科室经费少,样本也不多,而无法开展研究,而又想发SCI文章,此时我们最经济有效的方法就是:分析一下别人的数据,筛选出一些有价值的分子(mRNA,miRNA,lncRNA,circRNA,lncRNA),之后在自己的小样本中验证一下,发篇SCI文章还是轻轻松松的。接下来我们来介绍一篇挖掘GEO,TCGA数据库,筛选关键分子的文章。
文章1:
文章今年(2018)发表在:Cancer Management and Research 上IF=3.851。该文章发表的杂志影响因子虽然不高,但是分析思路还是可以借鉴的:文章主要分析结论是:利用公共数据(GEO,TCGA数据),通过数据筛选分析后发现两个miRNA(miR-182 and miR-20a)可以作为结直肠癌(colorectal cancer CRC)诊断的分子标志物,然后在组织和血液中进行验证,最终确定诊断参数。主要的分析思路分为三大步,如下图所示图所示:
第一步:利用公共数据进行筛选biomarker(Discovery phase)
首先,作者利用GEO数据库当中关于CRC的4个miRNA表达芯片数据,寻找患病样品和正常样品中差异表达的miRNA,通过取交集初步筛选到8个miRNA,再利用TCGA数据库中的CRC样本进行筛选,和qRT-PCR验证,其验证不仅在组织样中,同时还在血液中进行筛选,方便后续通过血液检测筛查CRC患者。最终确定了两个miRNA在CRC患者的组织和血液中表达都有上调,可作为CRC的分子诊断候选标志物(miR-182 and miR-20a)。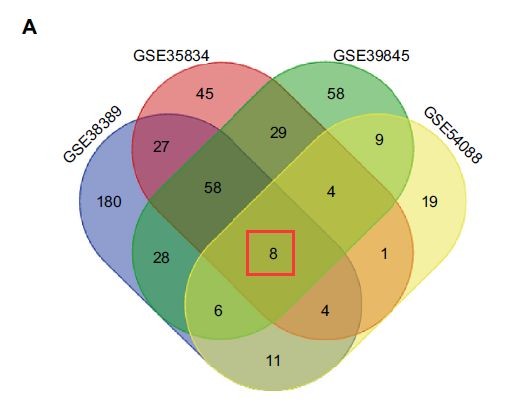
第二步:做训练数据集,确定诊断参数(Training phase)
之后,作者在自己收集的CRC患者血液样本中进行验证,40个健康样品,40个CRC患者样本,发现这两个miRNA在患者和对照中表达都存在差异,最终通过分析ROC线确定分类参数。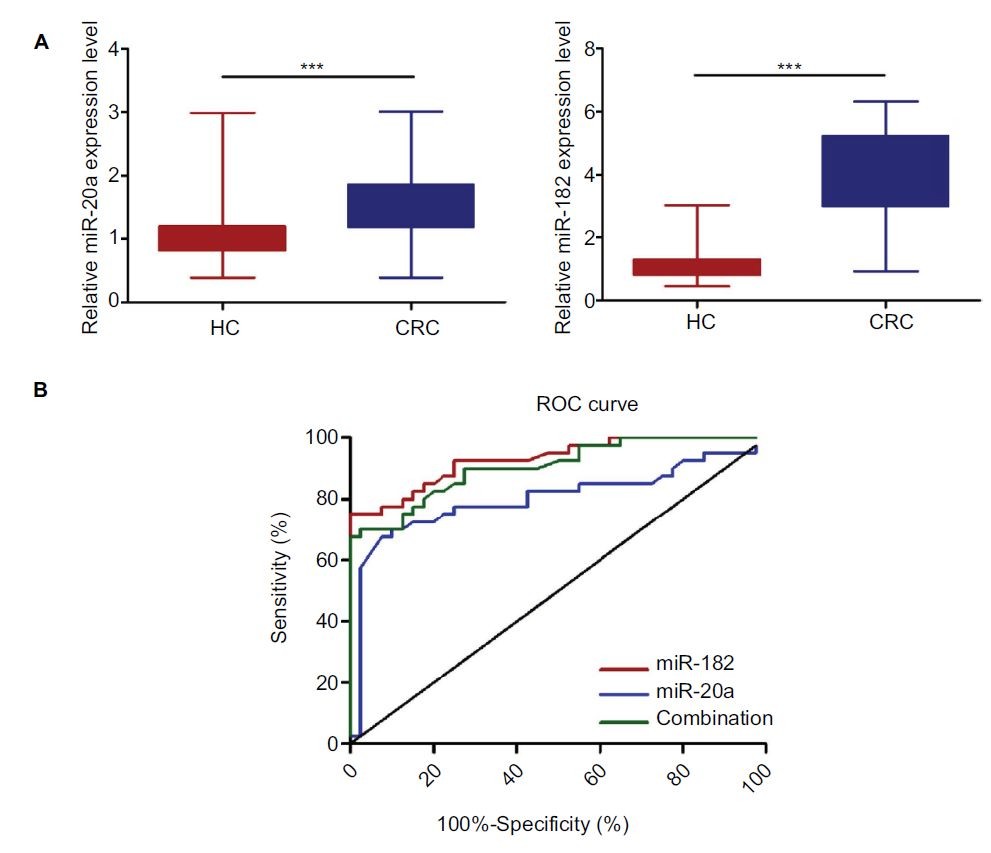
第三步:扩大数据集,确定最佳诊断参数(Validation phase)
最后作者扩大样品到150个,分别为健康样品50个,Adenoma(腺瘤)样品50个和CRC患者(stage I)进行验证并绘制ROC曲线,最终确定,诊断参数:
miR-182,miR-20a, and 2-miRNA combination were 2.620, 1.355,and 2.147, respectively.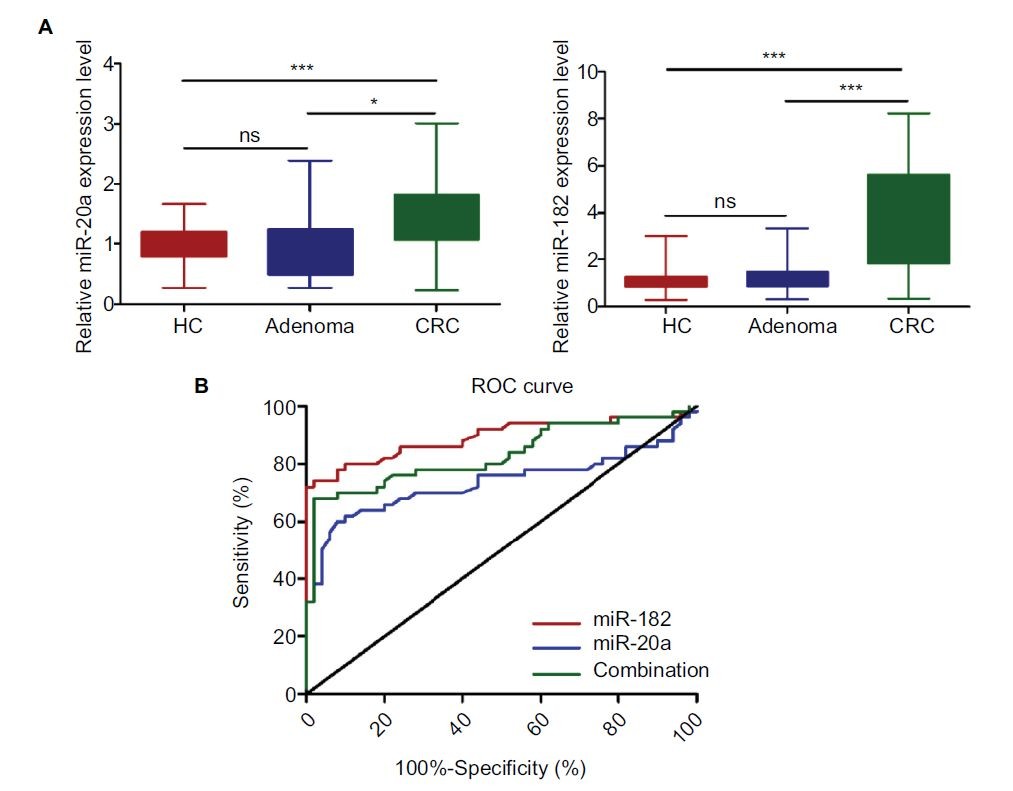
文章2:
题目:Expression profiles analysis identifies anovel three-mRNA signature to predict overall survival in oral squamous cellcarcinoma
期刊:Am J Cancer Res
分数:3.2
疾病:口腔鳞细胞癌(OSCC)
文章思路:
利用GSE13601,GSE30784, GSE37991三个mRNA芯片表达数据以及TCGA中OSCC表达数据分别做对照和患者差异基因分析,筛选到显著差异表达的76个上调基因、106个下调基因;然后进行GO和KEGG通路富集分析、PPI网络分析,IPA通路分析,生存分析等联合分析最终筛选到PLAU, CLDN8 and CDKN2A与OSCC预后相关,并且在另一个GEO数据GSE41613中得到了验证。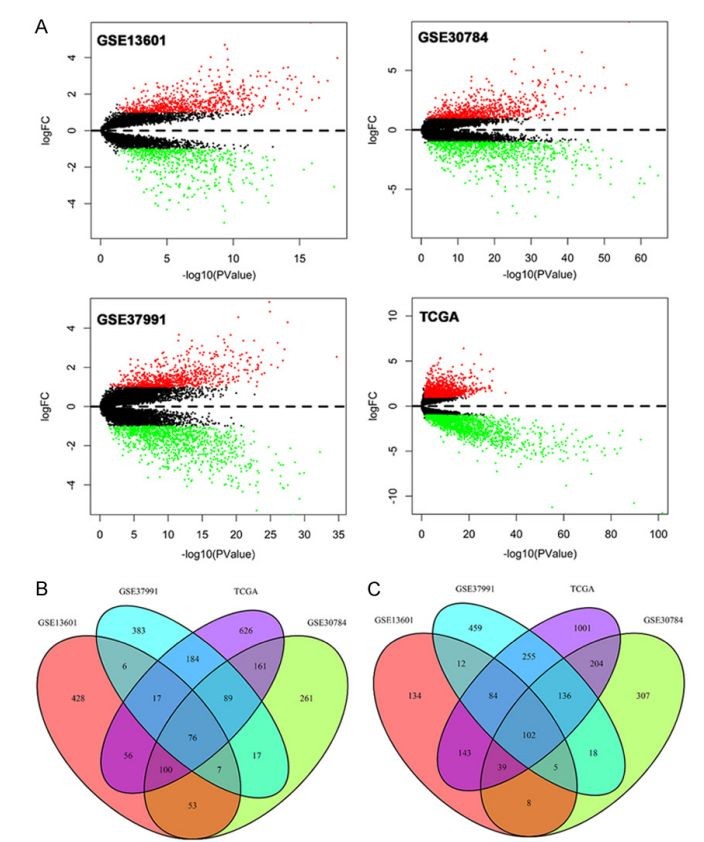
总结:
大家可以发现,GEO数据挖掘,以及TCGA数据的挖掘就是这个套路,先确定自己要分析的疾病,然后下数据,差异表达分析,筛选得到几个关键的分子(可以是lncRNA,mRNA,miRNA,circRNA),然后进行GO富集分析,Pathway富集分析,GSEA富集分析,PPI分析,IPA通路分析等,再做生存分析,ROC曲线分析等,就可以得到与疾病诊断相关的biomarker。如果要进一步发高分的文章,知其然更要知其所以然。可以再做一下小鼠模型,细胞模型,敲除,过表达等,对分子机理进行深入研究,干湿结合,文章就能再上一个层次。另外,还有其他套路组合,例如做一下WGCNA,或者联合Oncomine数据挖掘等等。
相关课程链接:
参考文獻:
[1] Liu X, Xu T, Hu X, et al. Elevated circulating miR-182 acts as a diagnostic biomarker for early colorectal cancer. Cancer Management and Research. 2018;10:857-865. doi:10.2147/CMAR.S158016.
[2] Zhao X, Sun S, Zeng X, Cui L. Expression profiles analysis identifies a novel three-mRNA signature to predict overall survival in oral squamous cell carcinoma. American Journal of Cancer Research. 2018;8(3):450-461.
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、biolinux搭建生物信息分析环境、linux命令处理生物大数据、perl入门到精通、perl语言高级、R语言画图、R语言快速入门与提高
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、 GEO芯片数据不同平台标准化 、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代fastq测序数据解读、
延伸阅读
GEO芯片数据挖掘|GSEA原理详解|利用好WGCNA|TCGA数据挖掘
- 发表于 2018-06-23 14:08
- 阅读 ( 8183 )
- 分类:文献解读
你可能感兴趣的文章
- 用AI写代码批量整理GEO数据库中的单细胞数据 354 浏览
- ROC 曲线绘制,多个和x轴空白 290 浏览
- 单细胞转录组数据挖掘流程记录-头颈癌(HNSCC)(GSE181919) 875 浏览
- R语言中转换成dummy dummy-variables 虚拟变量 1816 浏览
- 细胞焦亡、铁死亡及铜死亡等基因集 3857 浏览
- 记录一个数据下载:GSE12417 1378 浏览
相关问题
- 请教一下TCGA的蛋白数据怎么理解,它是有正负值的,这个正负值代表什么意思,为什么蛋白表达会有负值?我只想给它分成高表达,低表达和表达缺失组应该怎么处理呀? 0 回答
- wgCNA一个模块中找hub基因 我把cytoScapeinput-edges文件输入cytoscape中 用cytohubba计算 但是每次出来的结果都不一样 试了很多遍也不行 求解答 谢谢! 0 回答
- 我得到转录组分析的wgcna数据需通过cytoscape筛选关键模块的hub基因,边的权重应该设定多少来筛好 1 回答
- WGCNA中过滤低表达量(0.5)时datExpr0=datExpr0[1:n,datExpr0[n+1,] > meanFPKM]不成功,且变量从上步30769变成了0. ? 2 回答
- 跑WGCNA,用的独享服务器,报错 1 回答
- 我想咨询一下wgcna问题 0 回答
