转录组个性化分析之可变剪切分析工具:rMATS turbo
可变剪接(Alternative splicing):一个基因的外显子以不同的组合方式剪接形成不同的成熟RNA,从而被翻译成不同的蛋白质构体,因此,一个基因可能编码多种蛋白质。这个过程极大的增加了mRNA和蛋白质的多样性。可变剪切是一种后转录生物学过程,对细胞活动和疾病过程具有重要的且广泛的影响。到目前为止,也有很多软件可对其进行检测,今天我们就来了解一下这款常用可变剪切软件rMATS的最新版详情。

rMATS介绍
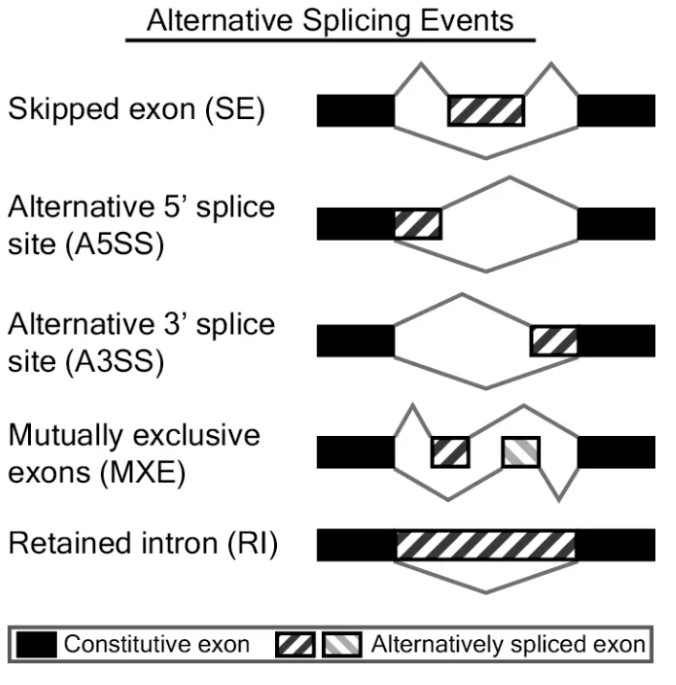
rMATS是检测可变剪切事件的常用软件之一,其可以从RNA测序数据中,检测出多种类型的可变剪切事件,并提供了定量和组间差异分析的功能,可对生物学重复的样本进行组间分析。rMATS可识别的可变剪切事件有5种:
Skipped exon (SE):外显子跳跃
Alternative 5' splice site (A5SS):5’端可变剪切
Alternative 3' splice site (A3SS):3’端可变剪切
Mutually exclusive exons (MXE):外显子互斥
Retained intron (RI):内含子保留
定量
rMATS采用exon inclusion level 来定义样本中可变剪切事件的表达量,以外显子跳跃(Skipped Exon)为例,正常的转录本称之为Exon Inclusion Isofrom, 发生了外显子跳跃的转录本则称之为Exon Skipping Isofrom。
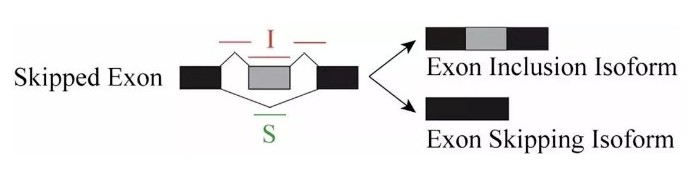
用 I 表示比对到Exon Inclusion Isofrom上的reads,S表示比对到Exon Skipping Isofrom上的reads, 则该外显子跳跃的可变剪切事件比例可以表示为:

exon inclusion level实际上是inclusion isofrom所占的比例,计算时,用长度校正了原始的reads数。其他类型的可变剪切事件也可以划分成上述两种isoform, 示意图如下:
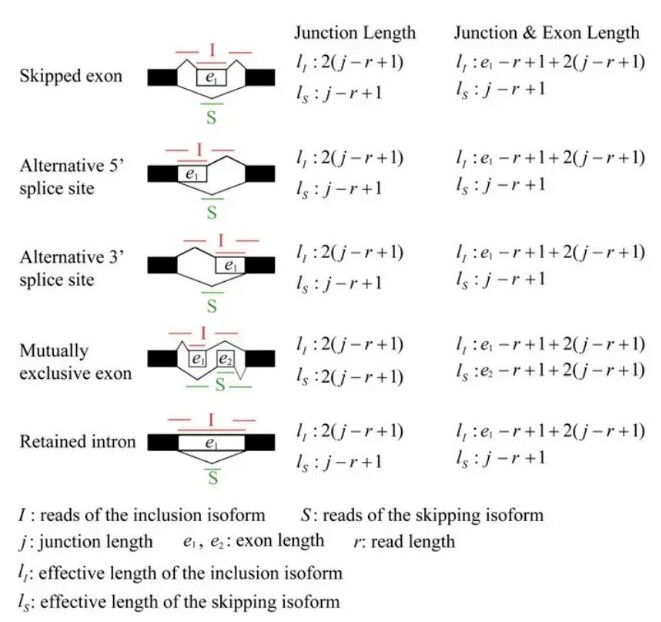 可以看到,rmats在计算isofrom的长度时,提供了两种方式,二者的区别就在于是否考虑跳过的exon的长度。
可以看到,rmats在计算isofrom的长度时,提供了两种方式,二者的区别就在于是否考虑跳过的exon的长度。
软件安装
rMATS turbo是rMATS的C/Cython版本。主要的差别在于速度和存储资源上,rMATS turbo相较于rMATS速度要快100倍,存储使用要小1000倍,因此我们安装rMATS turbo。
 安装依赖:Python (either 2.7 or 3.6),BLAS,LAPACK,GNU Scientific Library,GCC,gfortran,CMake等。保证以上依赖均存在的情况下就可以进行安装了。其实安装好conda,这些基础的包均已包括了。
安装依赖:Python (either 2.7 or 3.6),BLAS,LAPACK,GNU Scientific Library,GCC,gfortran,CMake等。保证以上依赖均存在的情况下就可以进行安装了。其实安装好conda,这些基础的包均已包括了。
conda create -n my_rmats_env
conda activate my_rmats_env
conda install rmats-turbo
rmats-turbo --version
软件使用
参数说明:
python rmats.py -h
usage: rmats.py [options]
optional arguments:
-h, --help show this help message and exit
--version show program's version number and exit
--gtf GTF An annotation of genes and transcripts in GTF format
--b1 B1 A text file containing a comma separated list of the
BAM files for sample_1. (Only if using BAM)
--b2 B2 A text file containing a comma separated list of the
BAM files for sample_2. (Only if using BAM)
--s1 S1 A text file containing a comma separated list of the
FASTQ files for sample_1. If using paired reads the
format is ":" to separate pairs and "," to separate
replicates. (Only if using fastq)
--s2 S2 A text file containing a comma separated list of the
FASTQ files for sample_2. If using paired reads the
format is ":" to separate pairs and "," to separate
replicates. (Only if using fastq)
--od OD The directory for final output
--tmp TMP The directory for intermediate output such as ".rmats"
files from the prep step
-t {paired,single} Type of read used in the analysis: either "paired" for
paired-end data or "single" for single-end data.
Default: paired
--libType {fr-unstranded,fr-firststrand,fr-secondstrand}
Library type. Use fr-firststrand or fr-secondstrand
for strand-specific data. Default: fr-unstranded
--readLength READLENGTH
The length of each read
--variable-read-length
Allow reads with lengths that differ from --readLength
to be processed. --readLength will still be used to
determine IncFormLen and SkipFormLen
--anchorLength ANCHORLENGTH
The anchor length. Default is 1
--tophatAnchor TOPHATANCHOR
The "anchor length" or "overhang length" used in the
aligner. At least "anchor length" NT must be mapped to
each end of a given junction. The default is 6. (Only
if using fastq)
--bi BINDEX The directory name of the STAR binary indices (name of
the directory that contains the SA file). (Only if
using fastq)
--nthread NTHREAD The number of threads. The optimal number of threads
should be equal to the number of CPU cores. Default: 1
--tstat TSTAT The number of threads for the statistical model.
Default: 1
--cstat CSTAT The cutoff splicing difference. The cutoff used in the
null hypothesis test for differential splicing. The
default is 0.0001 for 0.01% difference. Valid: 0 <=
cutoff < 1. Does not apply to the paired stats model
--task {prep,post,both,inte}
Specify which step(s) of rMATS to run. Default: both.
prep: preprocess BAMs and generate a .rmats file.
post: load .rmats file(s) into memory, detect and
count alternative splicing events, and calculate P
value (if not --statoff). both: prep + post. inte
(integrity): check that the BAM filenames recorded by
the prep task(s) match the BAM filenames for the
current command line
--statoff Skip the statistical analysis
--paired-stats Use the paired stats model
--novelSS Enable detection of novel splice sites (unannotated
splice sites). Default is no detection of novel splice
sites
--mil MIL Minimum Intron Length. Only impacts --novelSS
behavior. Default: 50
--mel MEL Maximum Exon Length. Only impacts --novelSS behavior.
Default: 500
运行:
##/path/to/b1.txt
/path/to/1_1.bam,/path/to/1_2.bam
##/path/to/b2.txt
/path/to/2_1.bam,/path/to/2_2.bam
###运行
python rmats.py --b1 /path/to/b1.txt --b2 /path/to/b2.txt --gtf Gallus_gallus.GRCg6a.101.gtf --od A_vs_B --tmp A_vs_B/tmp -t paired --variable-read-length --readLength 150 --cstat 0.0001 --libType fr-unstranded --novelSS --nthread 4
--b1 为组别1的bam文件的路径,若有生物学重复则bam文件路径用逗号隔开;
--b2 为组别2的bam文件的路径,若有生物学重复则bam文件路径用逗号隔开;
--gtf 为已知的基因及转录本的gtf文件;
--od 即为输出路径;
-t 测序类型为单端或者双端 ;
--variable-read-length 能够允许不同长度的长度的reads进行分析;
--readLength 若长度不一致时,可使用该参数将reads截取到给定的数值;
--libType 文库类型,可选择是否为链特异性;
--tmp 暂存目录;
--nthread 线程数。
输出文件详情请查看:https://github.com/Xinglab/rmats-turbo/blob/v4.2.0/README.md
- 发表于 2024-04-01 11:36
- 阅读 ( 4597 )
- 分类:软件工具
你可能感兴趣的文章
- rMATS turbo使用 1924 浏览
