单细胞转录组数据挖掘流程记录-BRCA乳腺癌(E-MTAB-8107)
单细胞转录组数据挖掘流程记录-BRCA乳腺癌(E-MTAB-8107)
数据介绍:
这个数据的表达量来自一篇泛癌文章,里面有很多项目的数据,其中就有这个数据,我们可以从中分离出来(GSE210347):
文章地址PanCAF :https://www.nature.com/articles/s41467-022-34395-2
数据下载:
wget -c https://ftp.ncbi.nlm.nih.gov/geo/series/GSE210nnn/GSE210347/suppl/GSE210347%5Fcounts.Rds.gz -O GSE210347_counts.Rds.gz wget -c https://ftp.ncbi.nlm.nih.gov/geo/series/GSE210nnn/GSE210347/suppl/GSE210347%5Fmeta.txt.gz -O GSE210347_meta.txt.gz wget -c https://ftp.ncbi.nlm.nih.gov/geo/series/GSE210nnn/GSE210347/suppl/GSE210347%5Fstudy%5Fmetadata.xls.gz -O GSE210347_study_metadata.xls.gz #合并两个表格按sample ID合并,注意保存txt格式 Rscript $scripts/merge_tsv_files.r -i GSE210347_meta.txt GSE210347_study_metadata.tsv -b SampleID -p metadata_all #筛选乳腺癌数据的metadata cat metadata_all.tsv |awk 'NR==1|| ($0~"E-MTAB-8107" && $0~/Breast/){print $0}'>E-MTAB-8107_BRCA_metadata.tsv
数据分析:
会根据metadata数据筛选乳腺癌数据:
Rscript $scripts/seurat_sc_qc.r --rds GSE210347_counts.Rds \ --project BRCA_E-MTAB-8107 \ --nUMI.min 500 \ --nUMI.max 50000 \ --nGene.min 250 \ --mito.gene.pattern "^MT.*-" \ --percent_mito 20 \ --log10GenesPerUMI 0.8 \ -o 01.qc -p BRCA_E-MTAB-8107 --metadata E-MTAB-8107_BRCA_metadata.tsv Rscript $scripts/seurat_sc_cluster.r --rds 01.qc/BRCA_E-MTAB-8107.afterQC.rds \ -p BRCA_E-MTAB-8107 --resolution 0.5 -d 30 -o 02.cluster \ --vars.to.regress nUMI percent_mito --high.variable.genes 2000
结果:
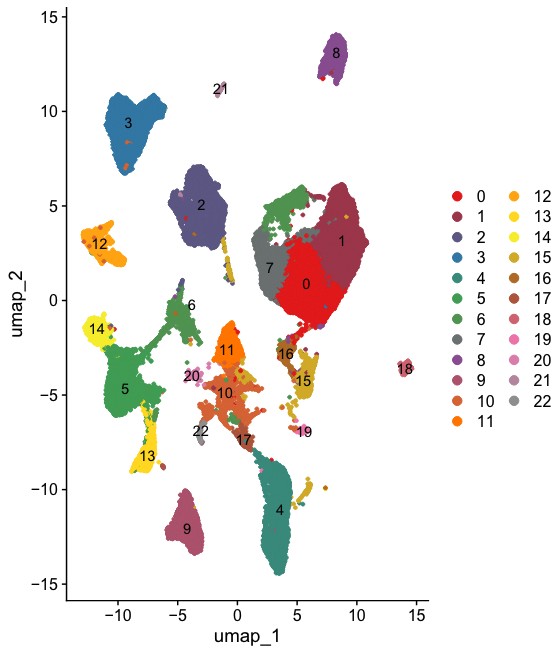
- 发表于 2024-06-27 13:33
- 阅读 ( 1860 )
- 分类:转录组
你可能感兴趣的文章
- 植物单细胞转录组文章 1358 浏览
- 拟南芥叶单细胞分析流程 1361 浏览
- 单细胞转录组镜像代码更新4.0 2022 浏览
- 单细胞转录组数据挖掘流程记录-头颈癌(HNSCC)(GSE181919) 2168 浏览
- 非负矩阵分解(cNMF) 3697 浏览
- 单细胞转录组如何将UCell CytoTRACE2 infercnv等结果添加到总的rds中 1637 浏览
相关问题
0 条评论
请先 登录 后评论
