单细胞转录组数据挖掘流程记录-非小細胞肺癌(NSCLC)(GSE127465)
单细胞转录组数据挖掘流程记录-癌(NSCLC)(GSE127465)
数据介绍:
来自谷歌翻译:
肿瘤浸润性髓系细胞 (TIM) 包括单核细胞、巨噬细胞、树突状细胞和中性粒细胞,已成为癌症生长的关键调节因子。这些细胞可以分化成一系列状态,这些状态可能会促进或限制肿瘤生长,但目前仍知之甚少。在这里,我们使用单细胞 RNA 测序来绘制非小细胞肺癌患者中的 TIM。我们发现了 25 种 TIM 状态,其中大多数在患者中可重复发现。为了促进这些群体的转化研究,我们还对小鼠中的 TIM 进行了分析。在比较不同物种的 TIM 时,我们发现树突状细胞和单核细胞之间的群体结构几乎完全一致;中性粒细胞亚群保守;巨噬细胞之间存在物种差异。相比之下,患者血液中的髓系细胞群体结构与 TIM 的重叠有限。这项研究确定了肺 TIM 概况,并为未来研究 TIM 作为免疫治疗靶点的潜力奠定了基础。
数据下载与准备:
https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE127465
数据预处理代码:
以下为处理代码
wget -c "https://www.ncbi.nlm.nih.gov/geo/download/?acc=GSE127465&format=file" -O GSE127465_RAW.tar wget -c https://ftp.ncbi.nlm.nih.gov/geo/series/GSE127nnn/GSE127465/suppl/GSE127465%5Fgene%5Fnames%5Fhuman%5F41861.tsv.gz wget -c https://ftp.ncbi.nlm.nih.gov/geo/series/GSE127nnn/GSE127465/suppl/GSE127465%5Fhuman%5Fcell%5Fmetadata%5F54773x25.tsv.gz #metadata文件处理:由于不同样本之间barcode ID可能重复。因此,细胞ID提取到第一列,并添加样本ID到第一列 zcat GSE127465_human_cell_metadata_54773x25.tsv.gz |awk -F '\t' 'BEGIN{OFS="\t"}{print $4$5,$0}' |gzip - >metadata.tsv.gz #解压包 tar xvf GSE127465_RAW.tar # 由于不同样本之间barcode ID可能重复,这里需要添加样本ID到barcode中 for id in `zcat metadata.tsv.gz |awk 'NR>1{print $5}'|sort|uniq`;do zcat *_human_${id}_raw_counts.tsv.gz|awk -F '\t' 'BEGIN{OFS="\t"}{print "'$id'"$0}' |gzip - >$id.counts.tsv.gz done #合并人的所有的counts数据,awk只输出第一个文件的表头 zcat *.counts.tsv.gz |awk -F"\t" 'NR==1 || $1!~ /barcode/ {print $0}' |gzip - > all_human_counts.tsv.gz
单细胞分析
这个数据有点特殊为indrop的单细胞数据, 也是测的3’端,因此和10X的数据处理类似
#count数据和metadata数据无需解压可直接读入 #读入数据并质控时间久,由于indrop的数据行是细胞列是基因需要转置,173954个细胞的矩阵大约消耗 100G-200G 左右内存 Rscript $scripts/seurat_sc_qc.r --count all_human_counts.tsv.gz --transpose \ --project GSE127465 \ --nUMI.min 500 \ --nUMI.max 50000 \ --nGene.min 250 \ --mito.gene.pattern "^MT.*-" \ --percent_mito 30 \ --log10GenesPerUMI 0.8 \ -o 01.qc \ -p GSE127465 --metadata metadata.tsv.gz #单细胞聚类分群分析 Rscript $scripts/seurat_sc_cluster.r --rds 01.qc/GSE127465.afterQC.rds \ -p GSE127465 --resolution 0.5 -d 30 \ -o 02.cluster --integrate.method harmony --batch.id Library \ --vars.to.regress nUMI percent_mito --high.variable.genes 2000
结果展示:
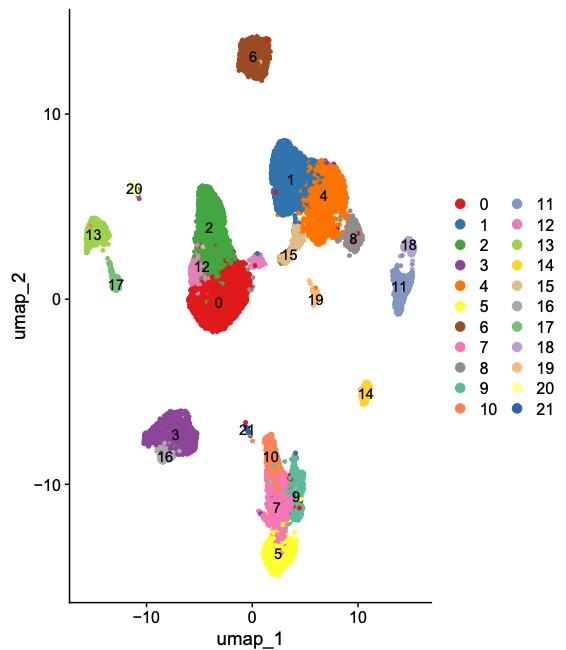
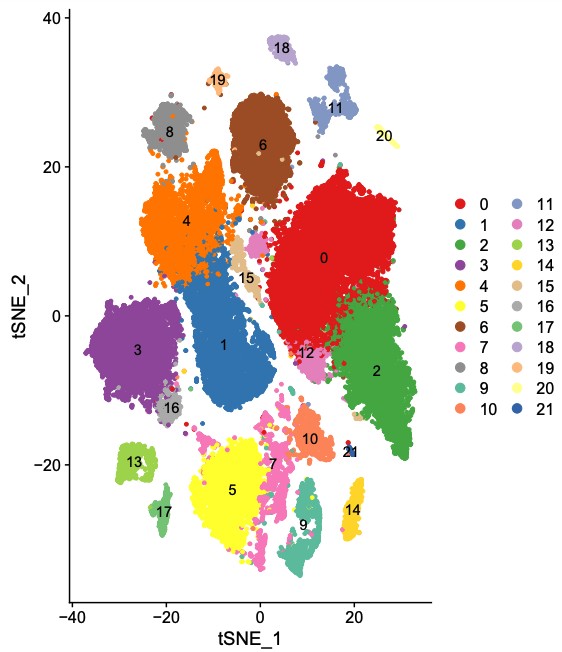
单细胞转录组分析课程推荐:https://bdtcd.xetslk.com/s/4i88K6
- 发表于 2024-07-24 11:11
- 阅读 ( 1350 )
- 分类:转录组
你可能感兴趣的文章
- 拟南芥叶单细胞分析流程 497 浏览
- 单细胞转录组镜像代码更新4.0 1158 浏览
- 单细胞转录组数据挖掘流程记录-头颈癌(HNSCC)(GSE181919) 879 浏览
- 非负矩阵分解(cNMF) 1737 浏览
- 单细胞转录组如何将UCell CytoTRACE2 infercnv等结果添加到总的rds中 894 浏览
- 单细胞转录组-infer CNV拷贝数变异分析介绍 1734 浏览
0 条评论
请先 登录 后评论