Scanpy数据结构:AnnData
Scanpy数据结构:AnnData
AnnData是python中存储单细胞数据的一种格式
1. AnnData数据结构:
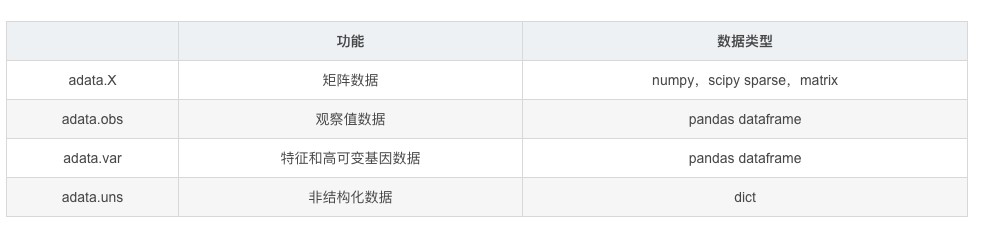
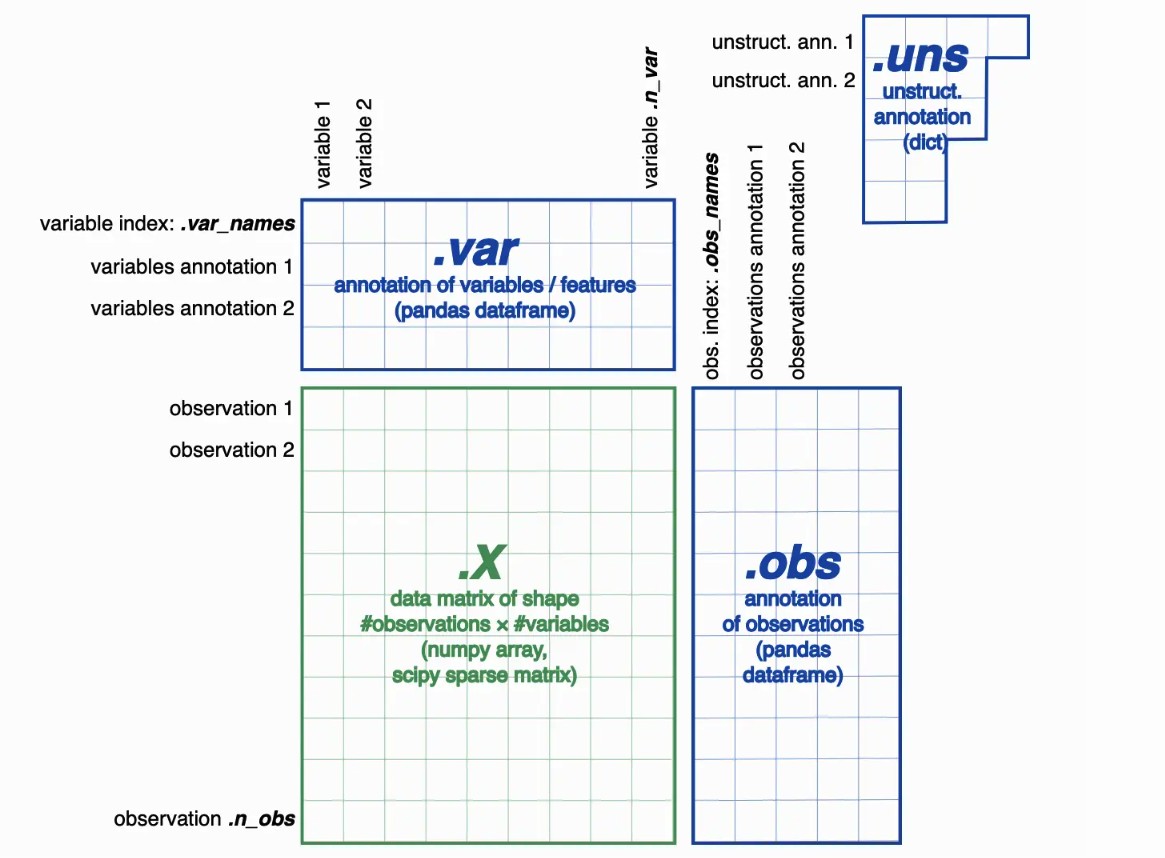
主要包含四个slots:
X(数据矩阵): 存储单细胞数据的核心矩阵,通常是一个二维数组,其中行表示细胞,列表示特征(基因或其他测量值)。
obs(观测信息): 包括每个细胞的元信息,如样本名称、细胞类型、质量信息等。obs 是一个观测特征的字典 metadata。
var(变量信息): 包括每个特征(基因)的元信息,如基因名、功能注释等。var 是一个变量特征的字典。
layers(层): 可以存储其他数据层,如归一化后的数据或差异表达分析的结果。
uns(未排序的数据): 用于存储其他未排序的数据和元信息
AnnData object with n_obs × n_vars = 6050 × 32285
obs: 'in_tissue', 'array_row', 'array_col', 'gender', 'age', 'tissue', 'Strain', 'n_genes_by_counts', 'log1p_n_genes_by_counts', 'total_counts', 'log1p_total_counts', 'pct_counts_in_top_50_genes', 'pct_counts_in_top_100_genes', 'pct_counts_in_top_200_genes', 'pct_counts_in_top_500_genes', 'total_counts_mt', 'log1p_total_counts_mt', 'pct_counts_mt', 'n_counts', 'n_genes'
var: 'highly_variable', 'means', 'dispersions', 'dispersions_norm'
uns: 'spatial', 'log1p', 'hvg', 'pca', 'neighbors', 'umap', 'tsne', 'tissue_colors'
obsm: 'spatial', 'X_pca', 'X_umap', 'X_tsne'
varm: 'PCs'
obsp: 'distances', 'connectivities'
AnnData 解释
1. obs
- 含义 : 存储每个观测样本(通常是spot/细胞)的metadata信息。
- 示例内容 :
- 'in_tissue': 表示细胞是否属于组织的一部分(布尔值或其他标签)。
- 'array_row', 'array_col': 表示细胞在空间阵列中的位置(行和列索引),通常用于空间转录组学数据。
- 用途 : 这些信息是对每个细胞的描述性特征,可以用于分组、过滤或可视化。
2. uns
- 含义 : 存储未结构化的metadata信息,通常是与整个数据集相关的全局信息,运行参数等
- 示例内容 :
- 'hvg': 高变基因(Highly Variable Genes)的相关信息,可能是一个列表或字典。
- 'spatial': 空间转录组学相关的全局信息,例如图像数据或空间坐标系的参考。
- 'tsne', 'umap': t-SNE 和 UMAP 的参数或配置信息,可能包括降维算法的超参数。
- 用途 : 这些信息通常是对数据集的整体描述,而不是针对单个细胞或基因。
3. obsm
- 含义 : 存储每个spot/细胞 的多维信息(multi-dimensional embeddings)。
- 示例内容 :
- 'X_pca': PCA 降维后的结果,形状为 (n_cells, n_components)。
- 'X_tsne': t-SNE 降维后的结果,形状为 (n_cells, 2) 或 (n_cells, 3)。
- 'X_umap': UMAP 降维后的结果,形状为 (n_cells, 2) 或 (n_cells, 3)。
- 'spatial': 空间转录组学中细胞的空间坐标,形状为 (n_cells, 2) 或 (n_cells, 3)。
- 用途 : 这些嵌入用于可视化或进一步分析,例如聚类、轨迹推断等。
4. varm
- 含义 : 存储每个基因 的多维信息。
- 示例内容 :
- 'PCs': 主成分分析(PCA)中每个基因的主成分载荷(loadings),形状为 (n_genes, n_components)。
- 用途 : 这些信息通常用于解释基因在降维空间中的贡献。
5. layers
- 含义 : 存储不同类型的表达矩阵(expression matrices)。
- 示例内容 :
- 'counts': 原始计数矩阵(raw counts),表示每个基因在每个细胞中的表达量。
- 'data': 经过预处理的表达矩阵(normalized 或 log-transformed 数据)。
- 用途 : 不同的层允许用户在同一 AnnData 对象中存储和切换不同的数据表示形式。
6. obsp
- 含义 : 存储spot/细胞 的成对关系(pairwise relationships)。
- 示例内容 :
- 'connectivities': 细胞之间的连接性矩阵,通常基于 k-最近邻图(kNN graph)构建。
- 'distances': 细胞之间的距离矩阵,通常用于构建 kNN 图。
- 用途 : 这些矩阵用于聚类分析、轨迹推断等任务。
总结
以下是对各字段的简要总结:
字段
含义
示例内容
obs
每个细胞的元信息
'in_tissue','array_row','array_col'
uns
全局元信息
'hvg','spatial','tsne','umap'
obsm
每个细胞的多维嵌入
'X_pca','X_tsne','X_umap','spatial'
varm
每个基因的多维信息
'PCs'
layers
不同类型的表达矩阵
'counts','data'
obsp
细胞之间的成对关系
'connectivities','distances'
这些字段共同构成了一个完整的 AnnData 对象,使得用户可以在同一个对象中存储、操作和分析单细胞数据的不同方面。
1.2 创建 Anndata 对象: 可以使用 Anndata 构造函数创建一个 Anndata 对象。
import anndata as ad
adata = ad.AnnData(X=data_matrix, obs=obs_info, var=var_info)
1.3 数据操作: Anndata 允许您执行多种数据操作,包括切片、过滤、转置、连接数据、添加元信息等。
# 切片数据
subset_data = adata[:, list_of_genes]
# 过滤细胞
adata = adata[adata.obs['quality'] > 0.9]
# 转置数据
adata_T = adata.T
1.4 数据可视化: Anndata 可以与 scanpy 或其他可视化工具结合使用,以可视化数据、绘制UMAP、t-SNE图等。
import scanpy as sc
sc.tl.pca(adata)
sc.pl.umap(adata, color='cell_type')
1.5 数据存储: Anndata 可以将数据存储为HDF5文件,以便将数据持久化和共享。
adata.write('my_data.h5ad')
- 发表于 2024-10-11 15:42
- 阅读 ( 1463 )
- 分类:转录组
你可能感兴趣的文章
- 空间转录组数据挖掘流程记录-甲状腺癌(GSE250521) 176 浏览
- 10X Visium空间转录组图像 956 浏览
相关问题
0 条评论
请先 登录 后评论
