单细胞转录组数据挖掘流程记录-头颈癌(HNSCC)(GSE181919)
数据介绍:
来自谷歌翻译:
肝细胞癌(HCC)代表了肿瘤微环境与肿瘤发展之间关系的范例。在这里,我们为来自四个相关部位的 10 名 HCC 患者生成了超过 70,000 个单细胞转录组:原发肿瘤、门...
数据介绍:
来自谷歌翻译:
头颈鳞状细胞癌(HNSCC)从正常组织逐步进展为转移性肿瘤。为了全面描述肿瘤细胞的异质性及其在 HNSCC 进展过程中的相互作用,我们对正常、白斑、HNSCC 和转移性 HNSCC 组织进行了单细胞 RNA 测序 (scRNA-seq) 分析。我们鉴定了表达 LGALS7B 的恶性细胞和表达 CXCL8 的成纤维细胞的新克隆,肿瘤组织中这些细胞的丰度与较差的预后结果相关。此外,我们证明白斑和肿瘤组织中的成纤维细胞表达 COL1A1,它与 CD44+ 恶性细胞相互作用,促进 HNSCC 进展。 CD4+FOXP3+调节性T细胞在白斑中扩增并在胶原刺激下表达LAIR2,这可能为肿瘤进展提供有利的微环境。总之,基质和免疫细胞相互作用在 HNSCC 的逐步进展中发挥着关键作用,我们已经确定了 HNSCC 进展过程中可能的治疗干预措施的步骤
数据下载与准备:
数据下载地址:https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE181919
数据预处理代码:
#下载数据 wget -c "https://www.ncbi.nlm.nih.gov/geo/download/?acc=GSE181919&format=file&file=GSE181919%5FBarcode%5Fmetadata%2Etxt%2Egz" -O GSE181919_Barcode_metadata.txt.gz wget -c "https://www.ncbi.nlm.nih.gov/geo/download/?acc=GSE181919&format=file&file=GSE181919%5FUMI%5Fcounts%2Etxt%2Egz" -O GSE181919_UMI_counts.txt.gz #metadata中的barcode ID和表达矩阵的barcode ID有区别,这里矫正一下 zcat GSE181919_Barcode_metadata.txt.gz |sed '2,$s/-/./' |gzip - >GSE181919_Barcode_metadata.txt1.gz
单细胞分析:
#读入数据并质控 #读入数据并质控 Rscript $scripts/seurat_sc_qc.r --count data/singcell_GSE181919/GSE181919_UMI_counts.txt.gz --project GSE181919 \ --sep "\t" \ --nUMI.min 500 \ --nGene.min 500 \ --mito.gene.pattern "^MT.*-" \ --percent_mito 20 \ --log10GenesPerUMI 0.65 \ -o 01.qc -p HNSCC_GSE181919 --metadata data/singcell_GSE181919/GSE181919_Barcode_metadata.txt.gz Rscript $scripts/seurat_sc_cluster.r --rds 01.qc/HNSCC_GSE181919.afterQC.rds \ -p HNSCC_GSE181919 --resolution 0.5 -d 30 -o 02.cluster --sctransform --integrate.method harmony --batch sample.id \ --vars.to.regress nUMI percent_mito --high.variable.genes 2000 --cpu 10
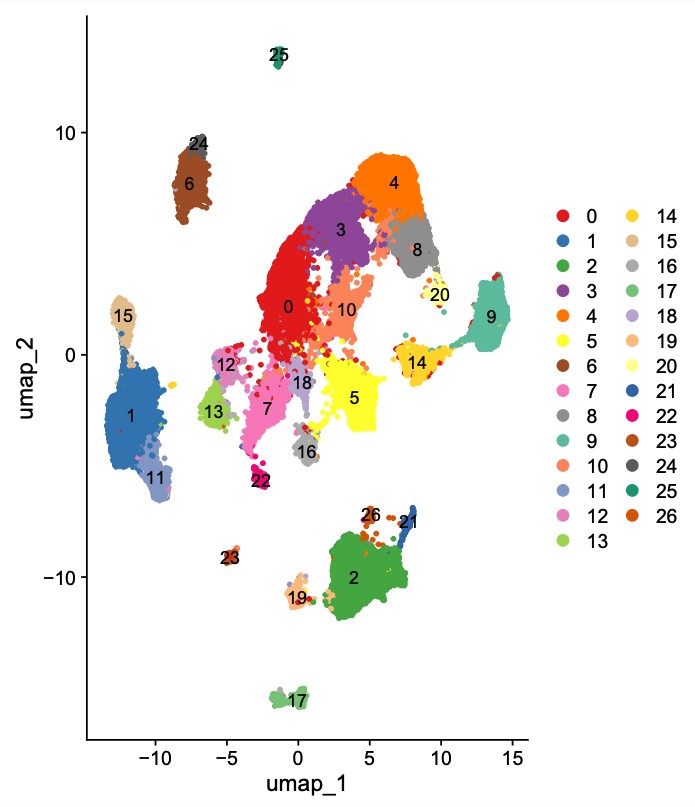
结果展示:
单细胞转录组分析课程推荐:https://bdtcd.xetslk.com/s/4i88K6
- 发表于 2024-10-31 14:05
- 阅读 ( 860 )
- 分类:转录组
你可能感兴趣的文章
- 用AI写代码批量整理GEO数据库中的单细胞数据 341 浏览
- 拟南芥叶单细胞分析流程 478 浏览
- 单细胞转录组镜像代码更新4.0 1130 浏览
- 非负矩阵分解(cNMF) 1675 浏览
- 单细胞转录组如何将UCell CytoTRACE2 infercnv等结果添加到总的rds中 879 浏览
0 条评论
请先 登录 后评论