eQTL分析之协变量计算
准备eQTL分析的协变量文件
参考文章来源
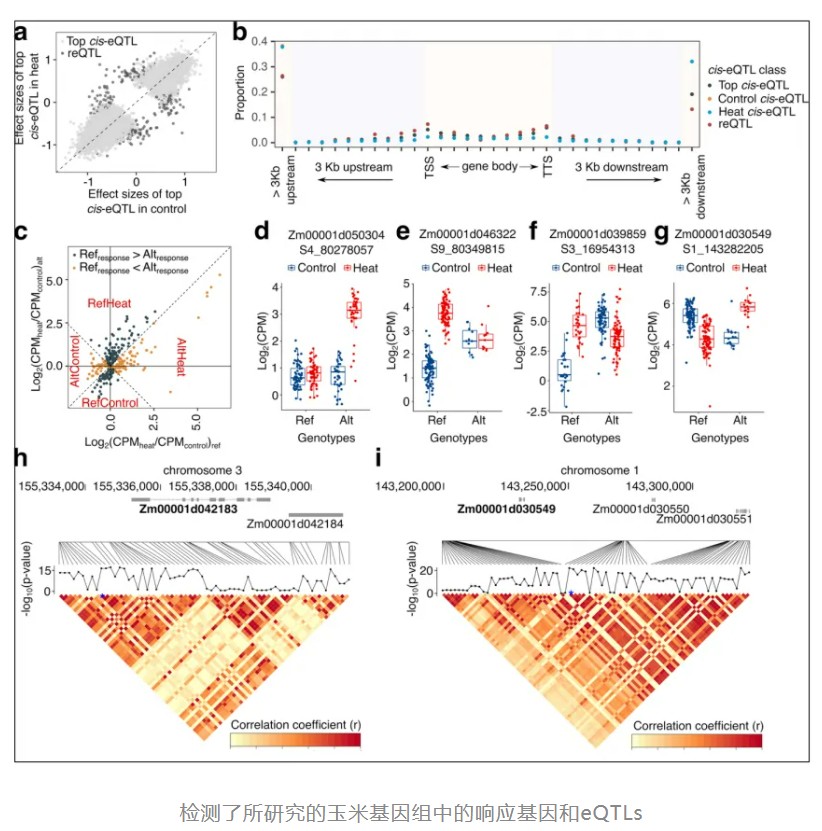
 通过分析 100 多个玉米近交系的转录组数据,文章对基因组中热胁迫表达响应的相关顺式(cis)和反式(trans)作用的 eQTLs进行了鉴定。并在热胁迫差异表达基因以及仅在热胁迫下表达的基因中,发现了引起热胁迫响应的cis-eQTLs,推断热胁迫响应表达可能是由于cis-eQTLs对启动子活性的影响。
通过分析 100 多个玉米近交系的转录组数据,文章对基因组中热胁迫表达响应的相关顺式(cis)和反式(trans)作用的 eQTLs进行了鉴定。并在热胁迫差异表达基因以及仅在热胁迫下表达的基因中,发现了引起热胁迫响应的cis-eQTLs,推断热胁迫响应表达可能是由于cis-eQTLs对启动子活性的影响。
 eQTL相关协变量计算
eQTL相关协变量计算
我们在之前的文章中分享了eQTL的分析流程,其中提到了需要准备协变量文件作为输入,在这篇文章中,作者也对协变量计算部分进行了描述:
 文章中提到协变量主要由主成分(PCs)和隐藏因子(hidden factor)两部分组成,其中主成分的计算我们在这里就不进行详细的描述了,有现成的软件可以进行主成分的计算,比如plink:plink使用手册https://zzz.bwh.harvard.edu/plink/dist/plink-doc-1.07.pdf
文章中提到协变量主要由主成分(PCs)和隐藏因子(hidden factor)两部分组成,其中主成分的计算我们在这里就不进行详细的描述了,有现成的软件可以进行主成分的计算,比如plink:plink使用手册https://zzz.bwh.harvard.edu/plink/dist/plink-doc-1.07.pdf
我们主要来看一下他的隐藏因子是如何计算的,根据文章内容,隐藏因子计算时用到了peer()这个R包,peer()的核心原理是利用贝叶斯法从基因表达谱中推断因子的隐藏决定因素及影响:https://github.com/PMBio/peer/wiki/

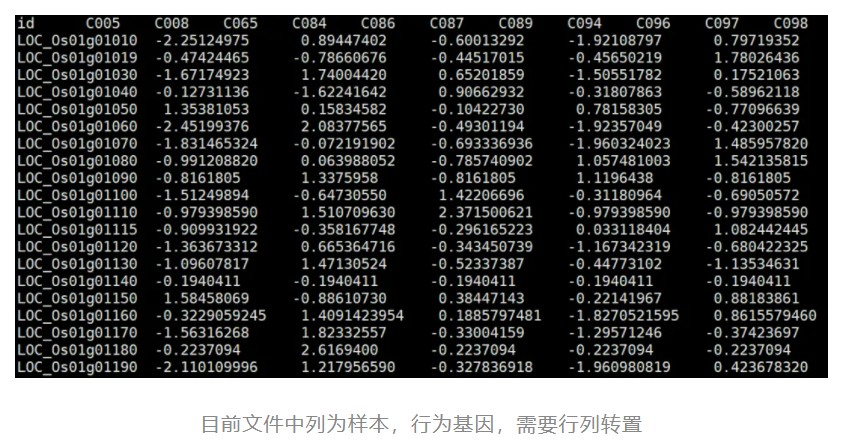
使用时只需要提供样本的基因表达量文件,文件中每一行为一个样本,每列为一个基因,我们通常获得的表达量的矩阵是每行是一个基因,每列是一个样本,所以要对数据进行转置,转置之前的表达量文件格式如下:
行列转置和peer进行隐藏因子推断的脚本如下:
input<-"表达量文件.txt"
##计算表达量的隐藏因子,作为eQTL分析的协变量之一
#expr = read.csv(input, header=TRUE)
expr = read.table(input,sep="\t",header=TRUE)
sample_list <- colnames(expr)
expr<-expr[,-1]
index <- which(sample_list == "id")
sample_list <- sample_list[-index]
print(sample_list)
expr<-t(expr)
print(expr)
library(peer)
#创建model
model = PEER()
#如果结果是NULL表示数据没有问题
PEER_setPhenoMean(model,as.matrix(expr))
#设置想要推断的隐藏因子的数目
#The number of PEER factors was selected as function of sample size (N):
#15 factors for N < 150
#30 factors for 150 ≤ N < 250
#45 factors for 250 ≤ N < 350
#60 factors for N ≥ 350
PEER_setNk(model,15)
PEER_getNk(model)
#根据模型进行推演(需要得到收敛的结果,如果没有,调整隐藏因子数量)
PEER_update(model)
#得到隐藏因子结果
factors = PEER_getX(model)
factors<-t(factors)
colnames(factors) <- sample_list
write.table(factors,"Express_hidden_factors.csv",sep="\t",quote = FALSE,col.names = TRUE,row.names = FALSE)
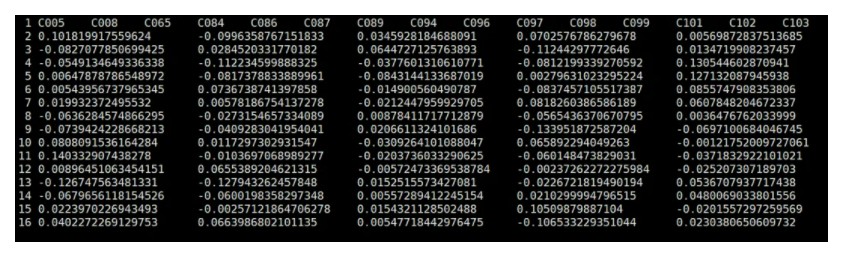
结果如下,我们在这里推断了前15个隐藏因子:
- 发表于 2024-11-12 17:37
- 阅读 ( 681 )
- 分类:软件工具
你可能感兴趣的文章
- TWAS分析的原理和工具 2041 浏览
相关问题
0 条评论
请先 登录 后评论
