绘制Upset 图
Upset 图(Upset Plot)是一种用于可视化集合间交集的图表,是传统维恩图的替代方法,特别适用于集合个数多或交集关系复杂的场景。它通过柱状图、点图和辅助条形图来直观展示集合交集的大小及分布。
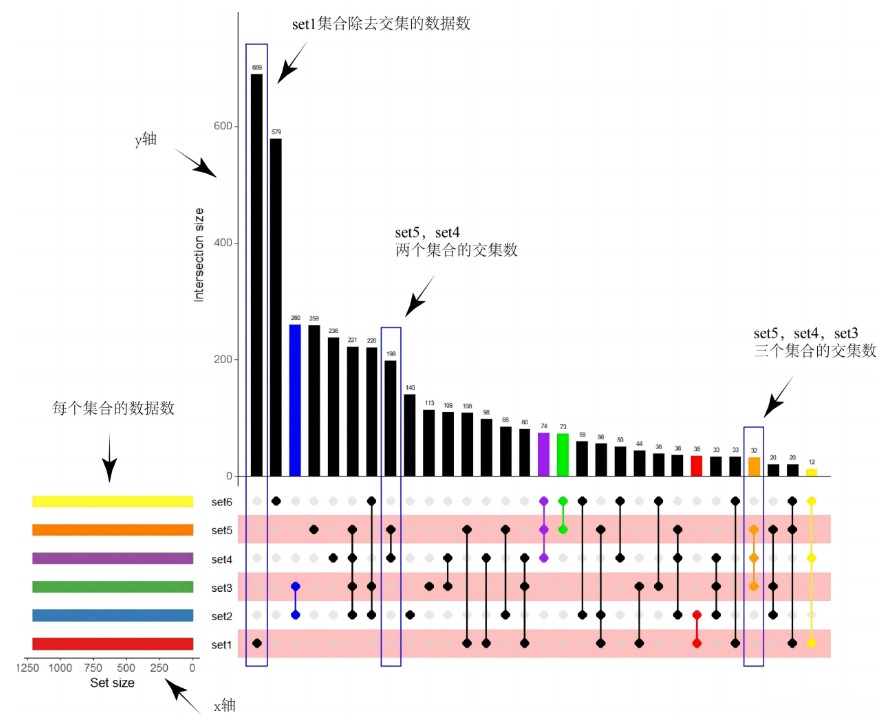
1.Upset 图的结构
主交集柱状图:显示每种交集的大小。
X 轴:表示集合交集组合。
Y 轴:表示对应交集的大小(元素数量)。
集合连接点图:在柱状图下方显示交集涉及的集合。
用点连接线标识参与交集的集合。
点表示集合是否参与特定交集。
集合条形图(可选):在侧面显示每个集合的总大小。
2.Upset 图的优点
能清晰地展示多达几十个集合之间复杂的交集关系。
比维恩图更高效,避免因集合过多导致图表混乱。
提供交集大小的定量信息,而维恩图通常只能粗略估计。
3.UpSetR包绘图
准备的数据文件格式如下:
all_deg_id CK_vs_A CK_vs_B CK_vs_C CK_vs_D CK_vs_E ENSRNA049471043 1 1 0 0 1 ENSRNA049471090 1 0 0 0 0 ENSRNA049471102 1 1 0 0 0 ENSRNA049471535 1 0 1 1 0 ENSRNA049473455 1 0 0 0 0 ENSRNA049475677 1 1 0 0 0 ENSRNA049476201 1 0 1 1 1
第一列为基因ID,后面每一列是不同的集合,集合中有此基因为1,没有便为0
。
下载需要的包:
# 1.安装CRAN来源常用包
#设置镜像,
local({r <- getOption("repos")
r["CRAN"] <- "http://mirrors.tuna.tsinghua.edu.cn/CRAN/"
options(repos=r)})
# 依赖包列表:自动加载并安装
package_list <- c("ggplot2","UpSetR","devtools")
# 判断R包加载是否成功来决定是否安装后再加载
for(p in package_list){
if(!suppressWarnings(suppressMessages(require(p, character.only = TRUE, quietly = TRUE, warn.conflicts = FALSE)))){
install.packages(p, warn.conflicts = FALSE)
suppressWarnings(suppressMessages(library(p, character.only = TRUE, quietly = TRUE, warn.conflicts = FALSE)))
}}
读入数据:
#读入文件
mydata<-read.table("Venn.txt",header=T,fill=T,check.names=FALSE,row.names = 1,na.strings="")
head(mydata)
过滤基因:
#去掉都不存的基因
mydata <- mydata[rowSums(mydata) != 0, ]
绘图:
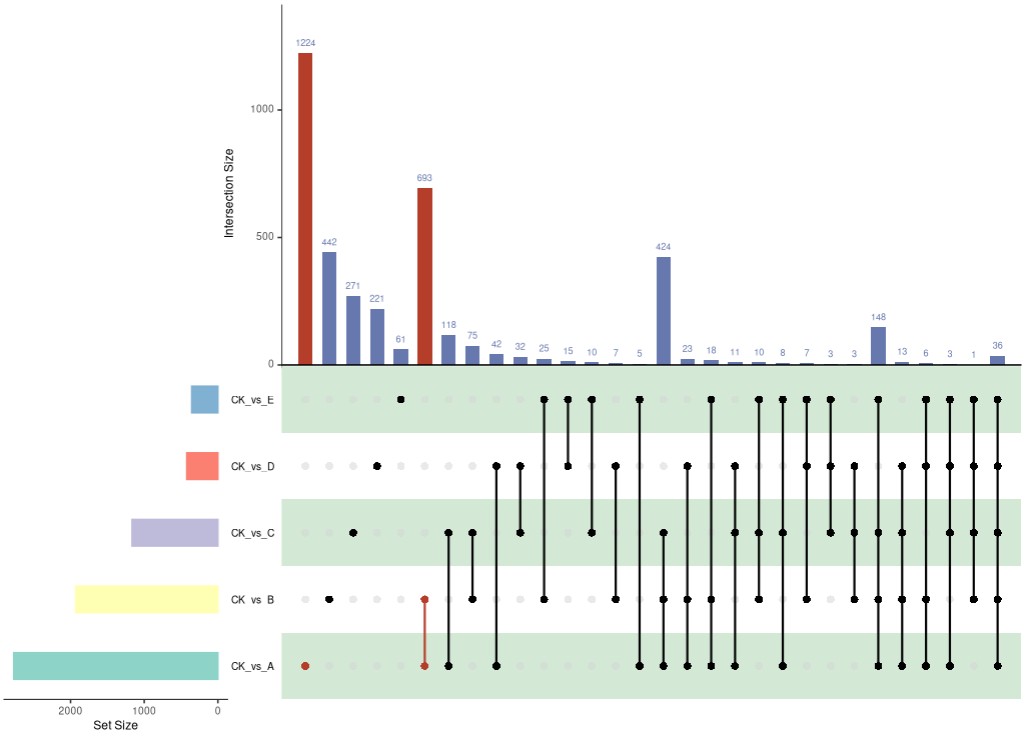
upset(mydata,
nsets = 10, #可视化数据集数量
nintersects= 60, #显示前多少个交集
main.bar.color = '#6778AE', #柱状图颜色
matrix.color="black", #集合点的颜色
#sets.bar.color= "#F3B1A0", #条形图条形的颜色
sets.bar.color= c(brewer.pal(5,"Set3")), #颜色数量需与集合数量一致
set_size.show = F, #是否在条形图上显示集合大小
shade.color = "#53A85F",#矩阵点图阴影的颜色
mb.ratio = c(0.5, 0.5), #矩阵图与主柱图之比
queries = list(list(query = intersects, #着重展示的交集
params = list("CK_vs_A","CK_vs_B"),
active = T,color="#B53E2B"),
list(query = intersects,
params = list("CK_vs_A"),
active = T,color="#B53E2B")
)
)
绘图如下:
好了,小编就先给大家介绍到这里。希望对您的科研能有所帮助!祝您工作生活顺心快乐!
- 发表于 2024-11-28 11:01
- 阅读 ( 1491 )
- 分类:R
你可能感兴趣的文章
- 已知集合交集数量绘制韦恩图 339 浏览
- R公式中如何加入变量 714 浏览
- R语言基础入门—数据结构(2) 715 浏览
- R语言基础入门—数据结构(1) 685 浏览
- R语言基础入门—R语言概述与安装! 881 浏览
