inferCNV 测试
Rscript ../infercnv.r -i ../BCC_GSE123813.qs \
-r "T_cells" --annotations_file ../cellanno_selected.tsv --gene_location ../hg38_gencode_v27.txt \
--cpu 20 --hmm --denoise --analysis_mode subclusters --cluster_by_groups
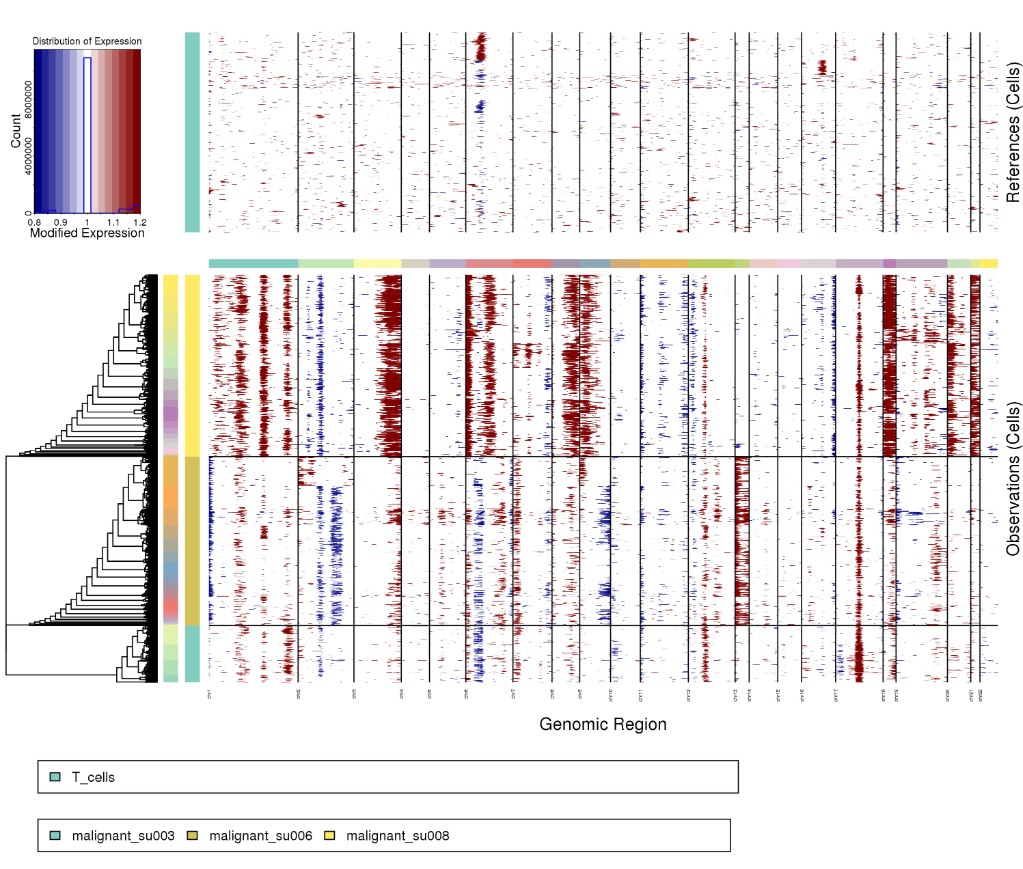
Rscript ../infercnv.r -i ../BCC_GSE123813.qs \
-r "T_cells" --annotations_file ../cellanno_selected.tsv --gene_location ../hg38_gencode_v27.txt \
--cpu 20 --hmm --denoise --analysis_mode subclusters
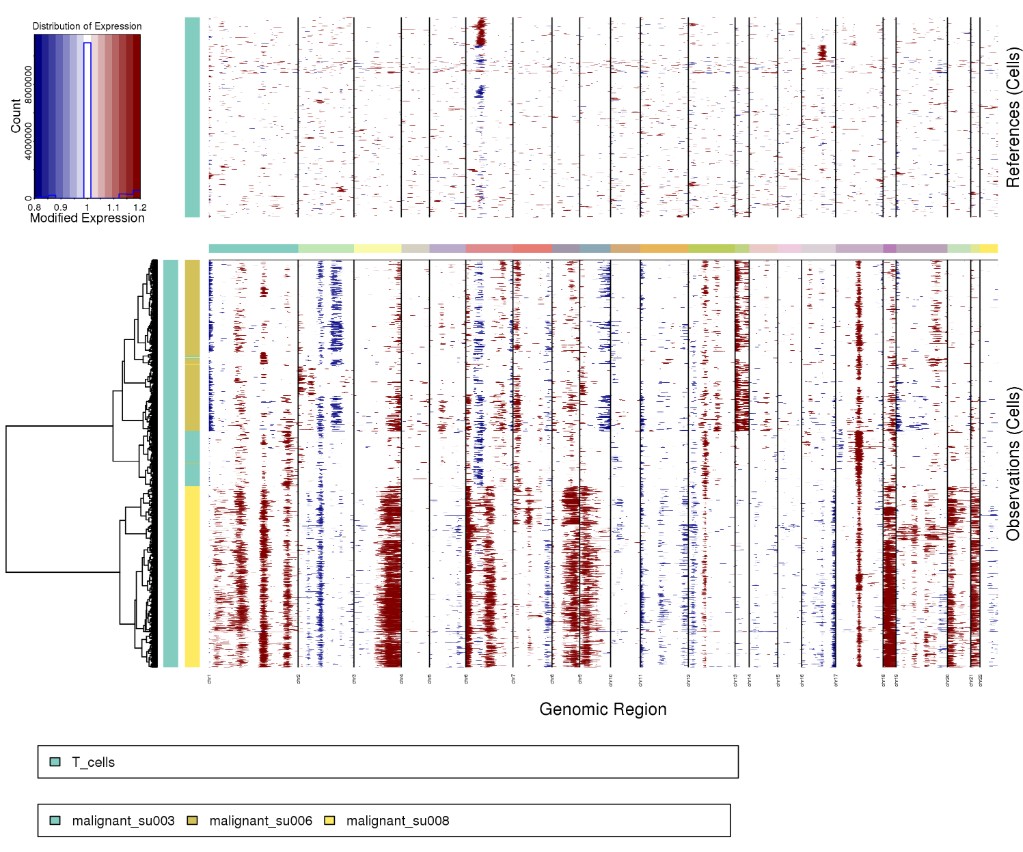
Rscript ../infercnv.r -i ../BCC_GSE123813.qs \
-r "T_cells" --annotations_file ../cellanno_selected.tsv --gene_location ../hg38_gencode_v27.txt \
--cpu 20 --hmm --denoise --analysis_mode samples
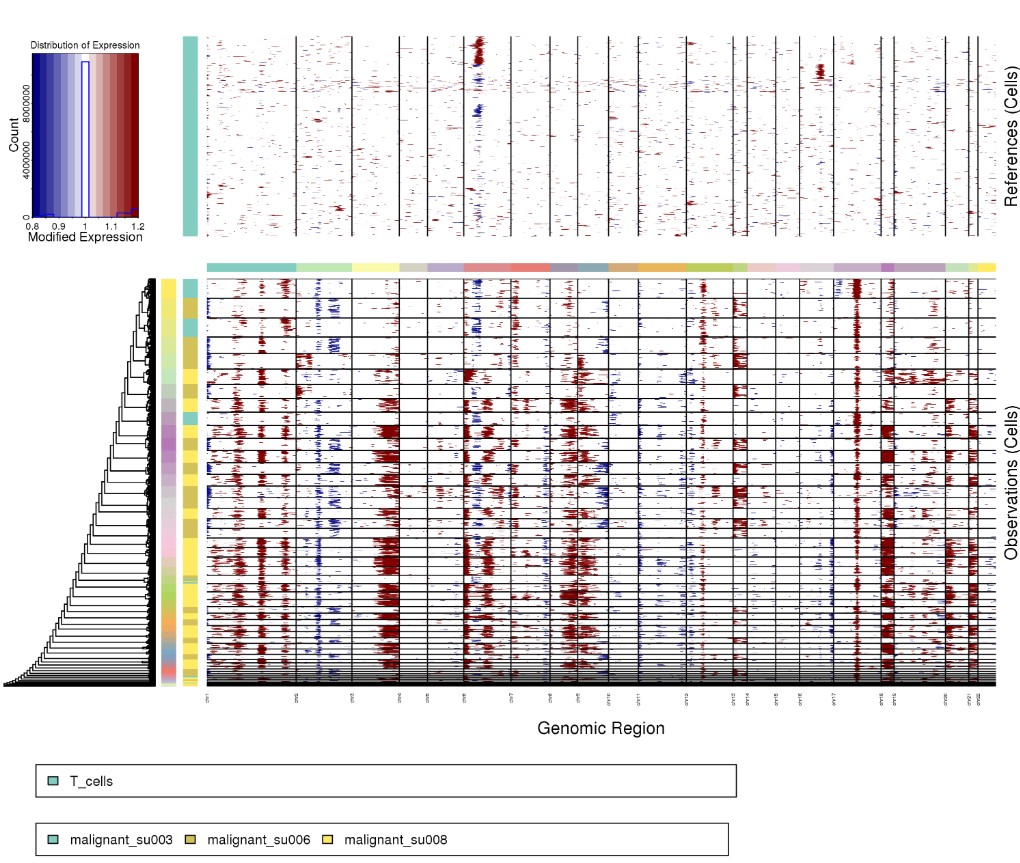
Rscript ../infercnv.r -i ../BCC_GSE123813.qs \
-r "T_cells" --annotations_file ../cellanno_selected.tsv --gene_location ../hg38_gencode_v27.txt \
--cpu 20 --hmm --denoise --analysis_mode samples --cluster_by_groups
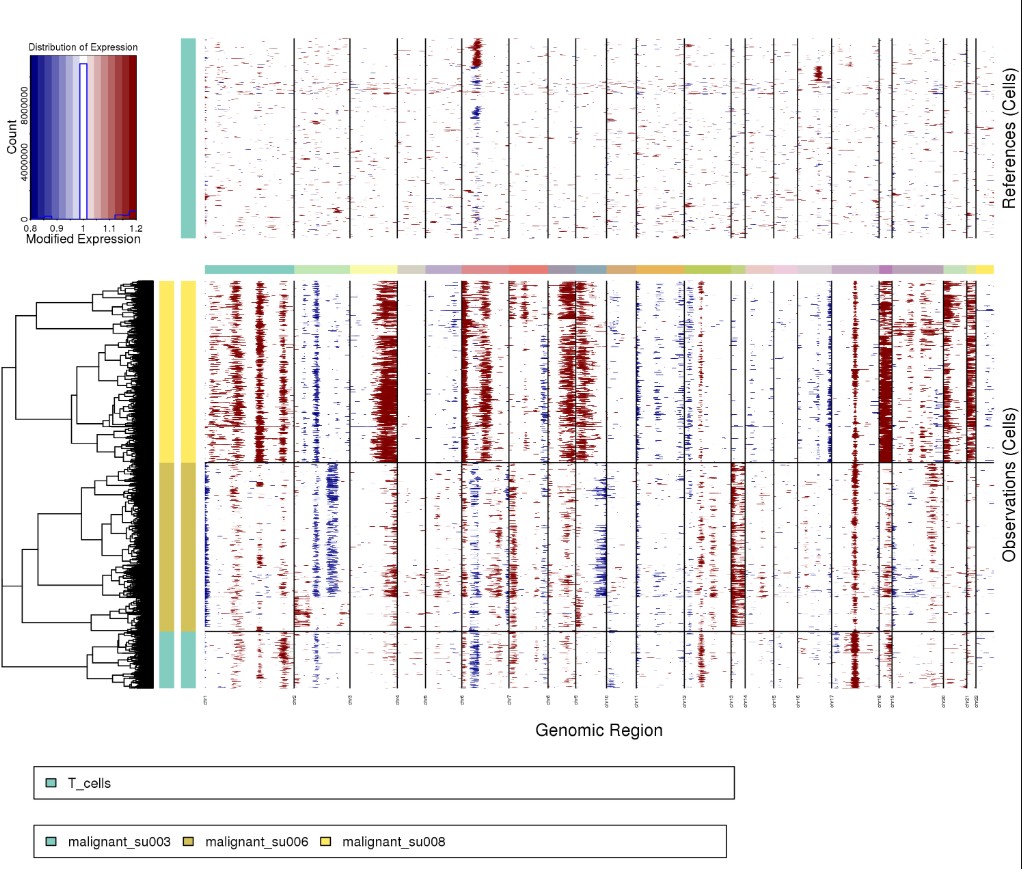
Rscript ../infercnv.r -i ../BCC_GSE123813.qs \
-r "T_cells" --annotations_file ../cellanno_selected.tsv --gene_location ../hg38_gencode_v27.txt \
--cpu 20 --hmm --denoise --analysis_mode subclusters --cluster_by_groups --tumor_subcluster_partition_method random_trees
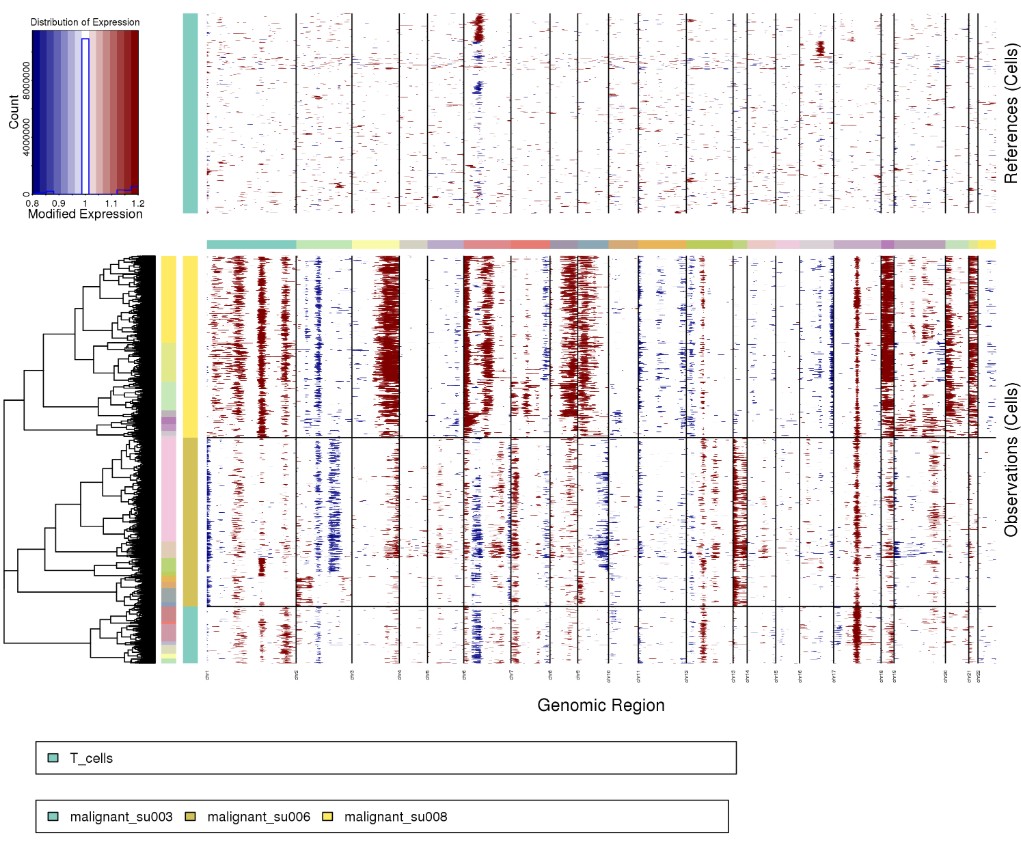
Rscript ../infercnv.r -i ../BCC_GSE123813.qs \
-r "T_cells" --annotations_file ../cellanno_selected.tsv --gene_location ../hg38_gencode_v27.txt \
--cpu 20 --hmm --denoise --analysis_mode subclusters --tumor_subcluster_partition_method random_trees
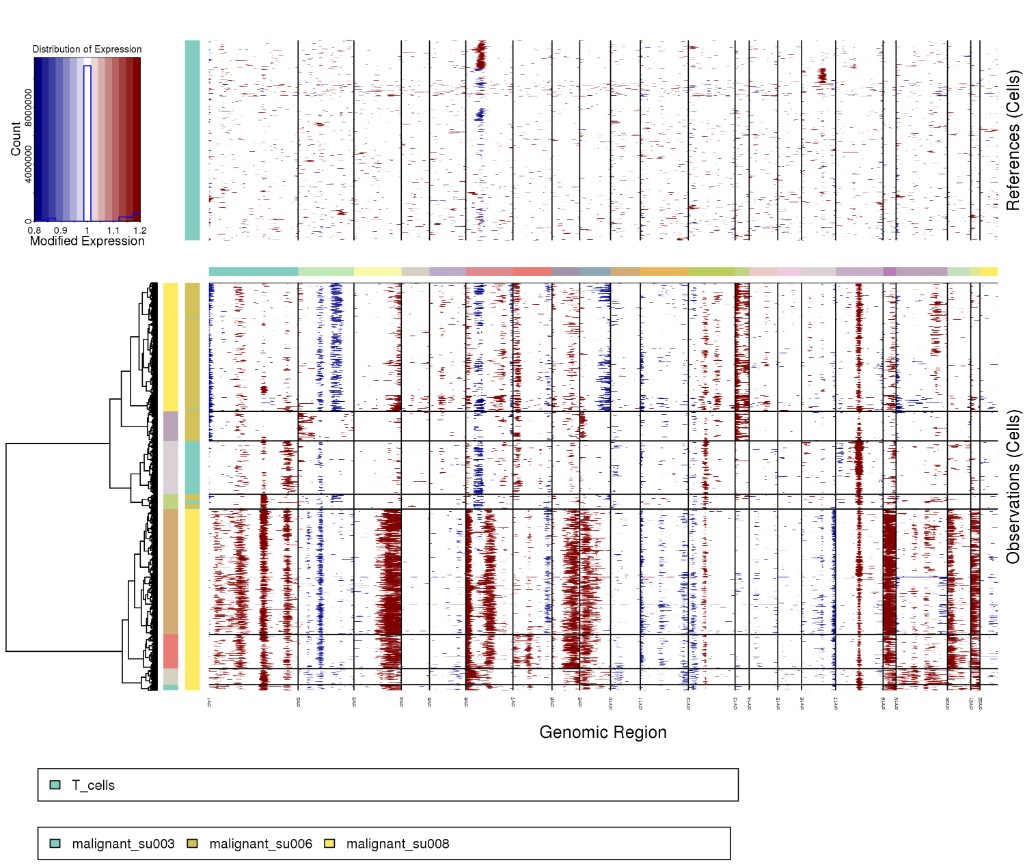
Rscript ../infercnv.r -i ../BCC_GSE123813.qs \
-r "T_cells" --annotations_file ../cellanno_selected.tsv --gene_location ../hg38_gencode_v27.txt \
--cpu 20 --hmm --denoise --analysis_mode samples --cluster_by_groups --tumor_subcluster_partition_method random_trees
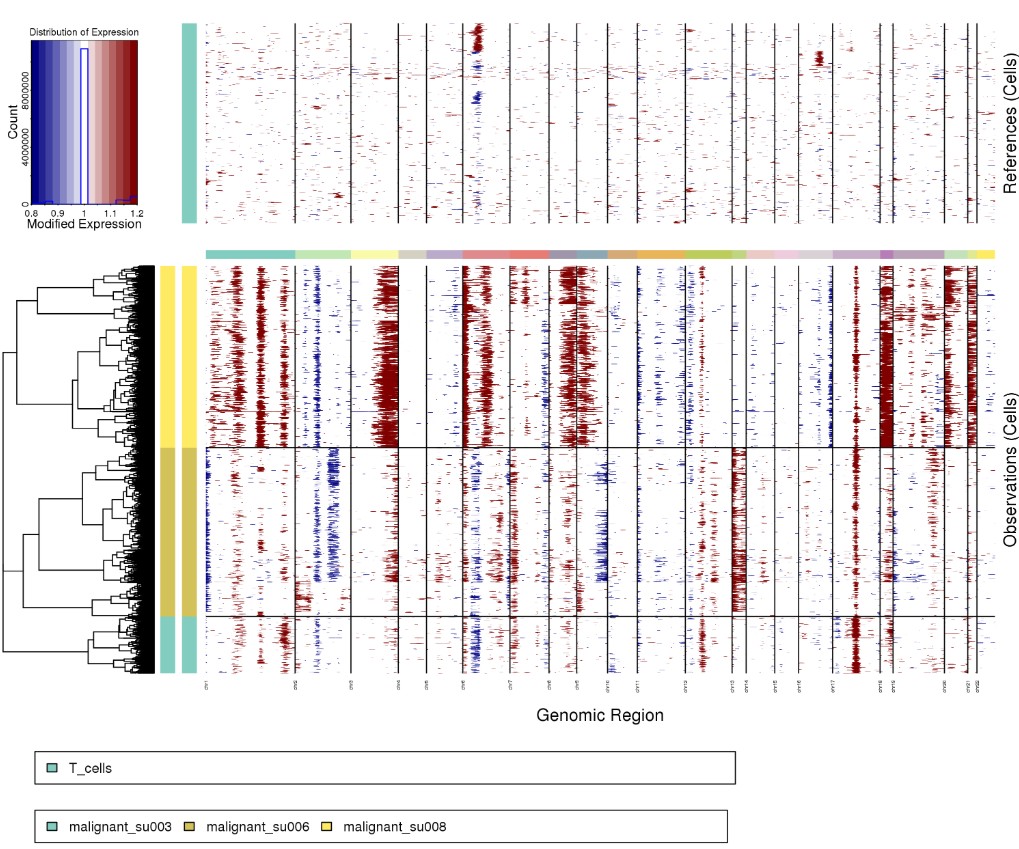
Rscript ../infercnv.r -i ../BCC_GSE123813.qs \
-r "T_cells" --annotations_file ../cellanno_selected.tsv --gene_location ../hg38_gencode_v27.txt \
--cpu 20 --hmm --denoise --analysis_mode samples --tumor_subcluster_partition_method random_trees


- 发表于 2025-01-10 11:26
- 阅读 ( 503 )
- 分类:转录组
