TCGA数据发表5分文章
医学研究的目的就是研究疾病的致病机理,找到相关的biomarker,对疾病的预防,诊断和治疗进行指导。癌症做为一种重要的疾病,其致病机理非常的复杂,目前大量研究是从大规模的病例数据中找一些跟治疗,生存预后相关的biomarker。我们曾介绍了一个癌症相关的数据库TCGA,其中含有33多种癌症,11000多个病例,包含多组学数据和临床数据。今天就跟大家介绍一篇发表在Gynecologic Oncology(IF:4.95)上的挖掘TCGA数据的文章“A 15-long non-coding RNA signature to improve prognosis prediction of cervical squamous cell carcinoma”。
研究思路
该文章的研究目的是找到宫颈鳞癌生存相关的biomarker,构建生存分析相关模型,为疾病的治疗和预后进行指导。首先来看一下文章整个思路的流程图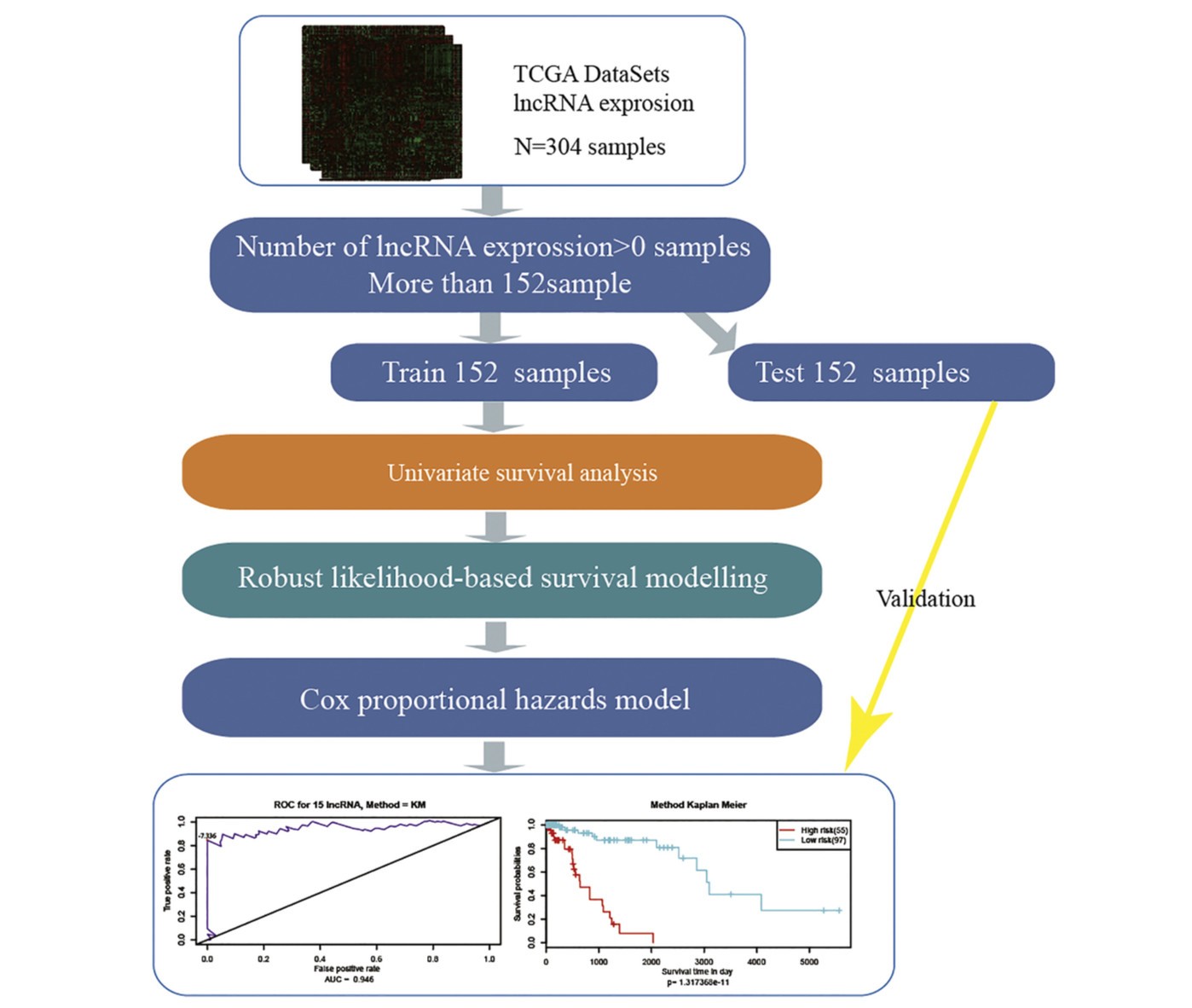
整个的分析流程非常的清晰,是一个值得借鉴的研究思路,下面我们介绍一下基于这个分析流程的分析结果。
文章具体内容
从TCGA下载公开数据
从TCGA下载到304例宫颈鳞癌的转录组数据,从中提取lncRNA的表达量,并将样品随机分成训练样本和测试样本两组。分成两组的目的主要是为了检验生存预测模型是质量。lncRNA 表达量筛选
剔除掉低表达量的lncRNA,保留高表达量的lncRNA。从TCGA下载到14,488个lncRNA, 经过筛选,获得6855个表达量高的lncRNA。鉴定和筛选预后相关的lncRNA
在测试数据集中,通过单因素cox分析,筛选出809个跟预后显著相关的lncRNA,筛选标准为P值小于0.05。由于单因素的分析结果太多,需要采用鲁棒性分析,进一步筛选。单因素鲁棒性分析
鲁棒性筛选,就是对数据集进行多次抽样分析,筛选出那些在多次抽样分析结果中出现频率较高的lncRNA。选择其中显著性lncRNA,再进行下面的多因素分析。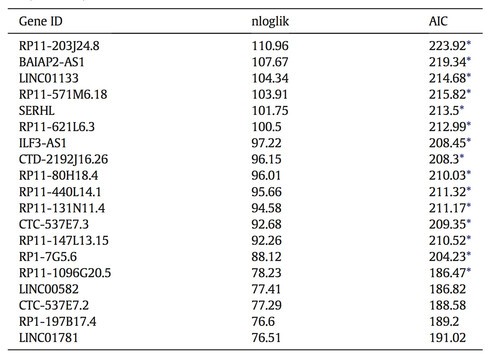
多因素分析,构建风险值评估函数
构建多因素的cox分析模型,并基于多个因素对生存率的影响贡献,构建多因素的风险值预测函数。
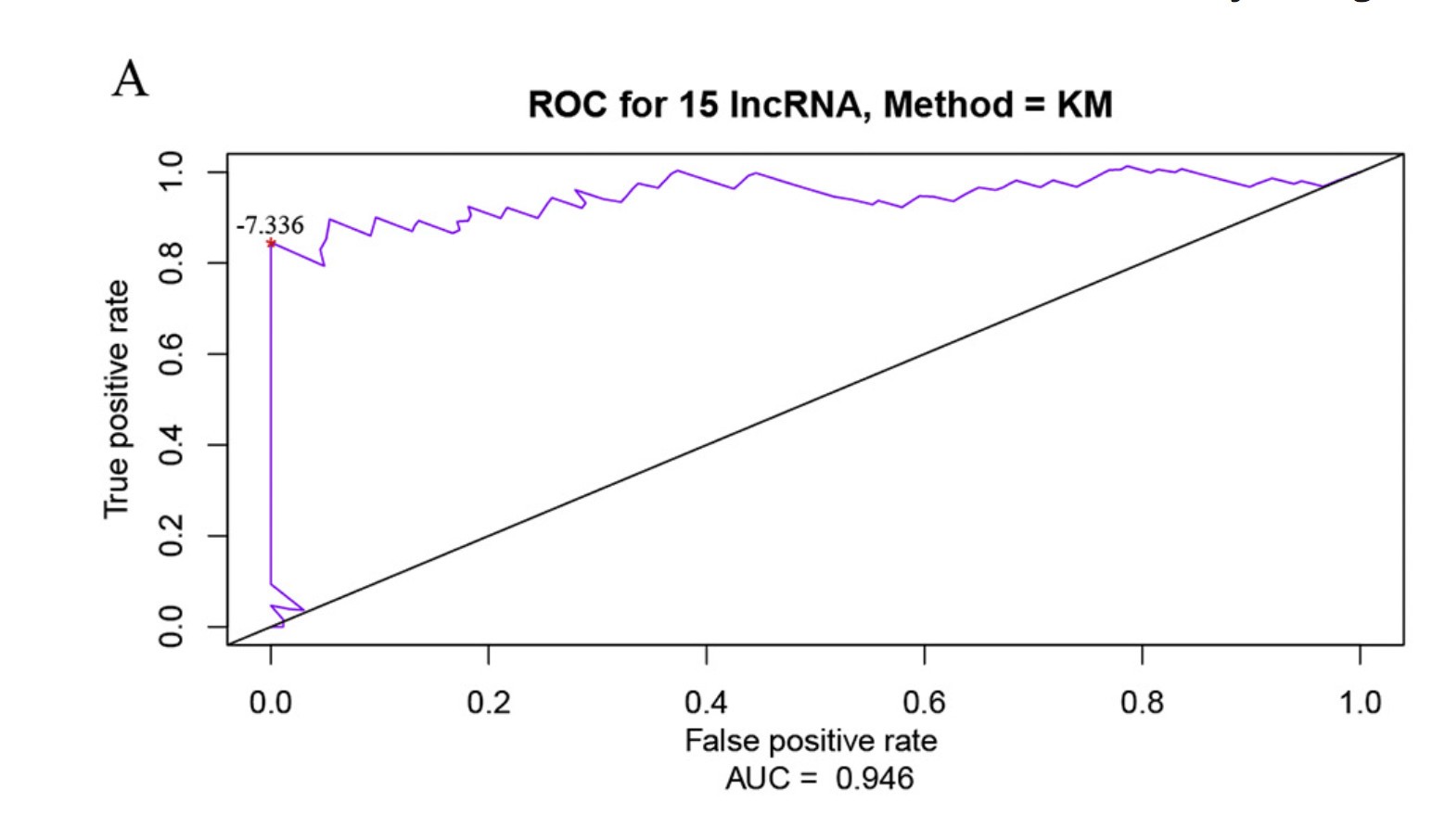
采用ROC曲线优化最佳分类阀值,并在测试数据上验证模型针对风险函数,进行二分类,筛选出一个最优阀值,能将病例样本分成高,低风险两类。分类效果很好,AUC达到0.946。
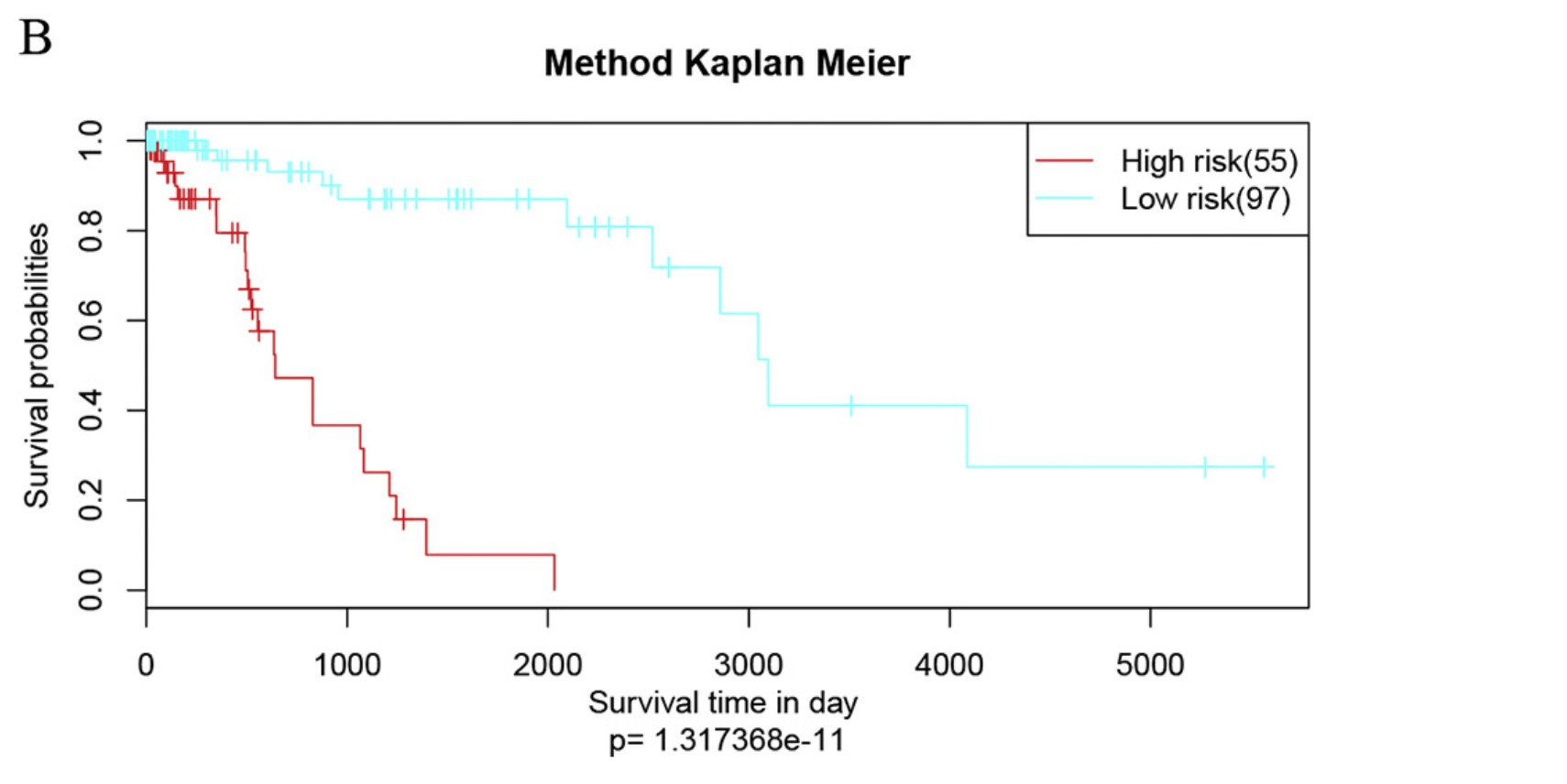
在测试数据集上采用生存分析对风险模型进行验证,发现模型能显著的将样本进行很好的区分。
总结
总体来看,该篇文章思路清晰,结果可靠,操作性强,值得借鉴。目前公共数据库里类似的数据实在有太多了,下载公共数据库里的数据,采取类似的思路整理些SCI文章,其实并不是一个困难的事情!怎么样?您想发一篇类似的SCI论文吗?想的话,这里我给您推荐一门《TCGA-生存分析》的自学课程,该课程主要介绍利用公共数据库进行生存分析,内容覆盖上面文章的整个分析流程,讲解详尽,简单易学,欢迎一起学习!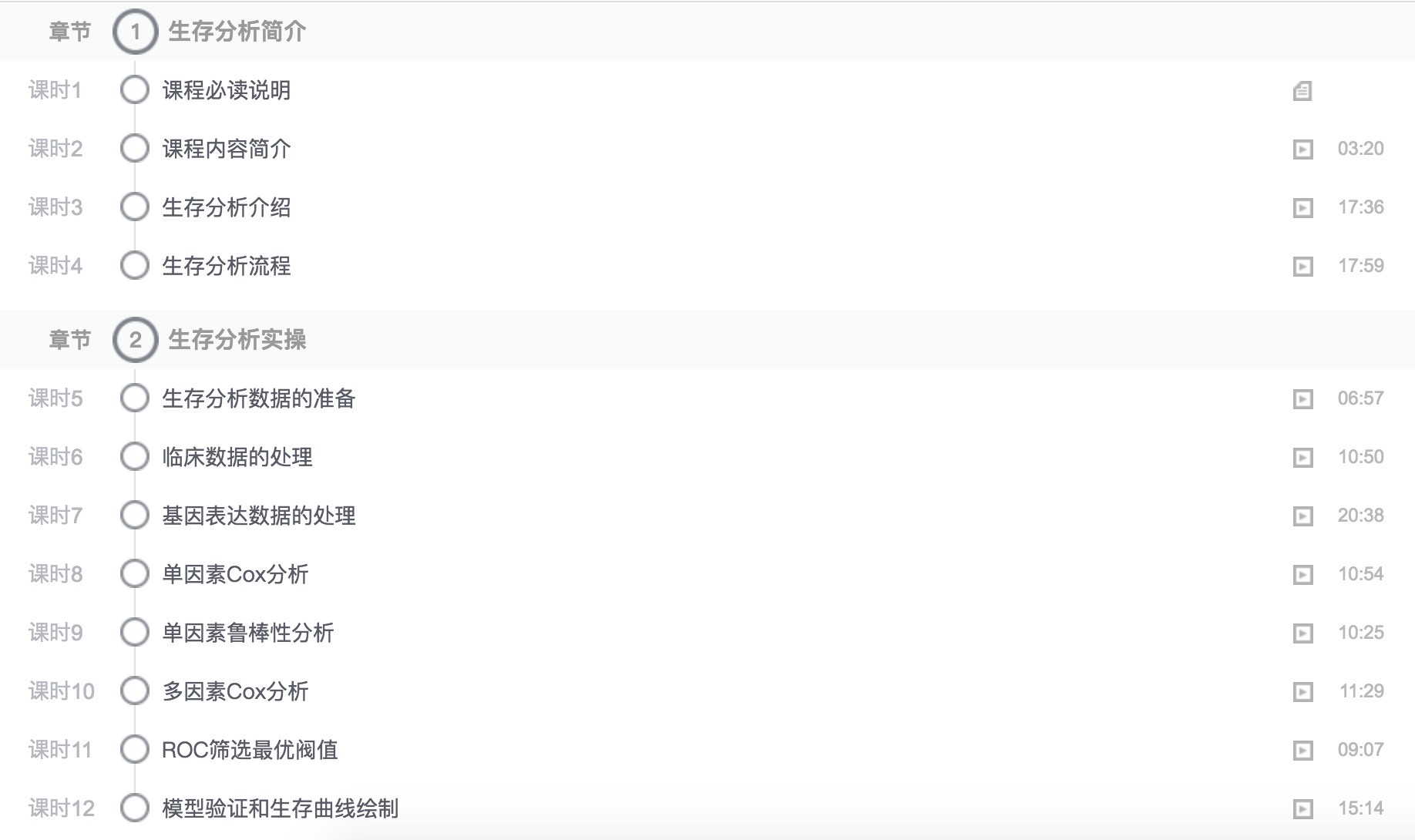
如果您对TCGA数据挖掘感兴趣,请学习我的TCGA系列课程:
- 发表于 2018-07-06 17:22
- 阅读 ( 6533 )
- 分类:TCGA
你可能感兴趣的文章
相关问题
- 请教一下TCGA的蛋白数据怎么理解,它是有正负值的,这个正负值代表什么意思,为什么蛋白表达会有负值?我只想给它分成高表达,低表达和表达缺失组应该怎么处理呀? 0 回答
- TCGA下载 docker报错 0 回答
- TCGA差异分析如何根据临床信息分组? 1 回答
- TCGA用R数据下载错误 1 回答
- 老师,想请教一下TCGA基因表达数据的问题,我从xena.ucsc网页上下载了基因表达数据TCGA-CESC.htseq_counts.tsv;然后发现该数据中只有Ensembl格式的基因ID ,没有SYMBOL格式的。所以接下来进行基因ID格式转换,却发现同一个SYMBOL ID对应的多个Ensembl格式的ID,想问下老师,这种情况该怎么处理?同一个SYMBOL ID所对应的多个Ensembl格式ID的基因表达数据应该留下哪一个? 1 回答
- TCGA的RNA-seq count annotation过滤,有些不知道是否需要删除 0 回答
