MCScanX 在biolinux上安装及使用
内容已经更新,可观看《基因家族分析实操课程》MCScanX分析使用;
一、软件安装
1.安装软件下载地址主页:MCScanX
MCScanX源码地址,把这个文件下载下来:http://chibba.pgml.uga.edu/mcscan2/MCScanX.zip
下面的代码,可在我们的biolinux系统上直接执行,创建目录及下载:
cd /biosoft
sudo wget http://chibba.pgml.uga.edu/mcscan2/MCScanX.zip
2,下载完成之后,就可以解压安装了:
unzip MCScanX.zip
cd MCScanX make
因为/biosoft 为root的目录,所以遇到权限问题时在命令前面加sudo 之后再运行就没问题了;
如果安装过程中报错,可参照:https://www.omicsclass.com/article/104
二、基因家族复制加倍分析(练习)
官方网站上提供测试代码,还有测试数据,存放在MCScanX安装目录的data目录当中,非常适合初学者练习:
data目录数据:
1.先来运行一下共线性分析(物种内,拟南芥为例第一个例子)
使用拟南芥的测试数据at开头的文件,用了data目录下的两个文件,at.gff 和at.blast 分别为基因的位置信息和blast比对结果:
官方例子:http://chibba.pgml.uga.edu/mcscan2/examples/example1.php
sudo ./MCScanX data/at
运行完成之后生成文件如下:
其中at.collinearity 为共线性结果,at.tandem为串联重复基因结果,这两个文件最重要;
2.结合基因家族分析基因加倍与复制
上面做完全基因组共线性分析后,可根据自己的基因家族信息,绘制基因家族圈图:
官方例子:http://chibba.pgml.uga.edu/mcscan2/examples/example12.php
代码如下
http://chibba.pgml.uga.edu/mcscan2/examples/family.ctl wget http://chibba.pgml.uga.edu/mcscan2/examples/MADS_box_family.txt sudo java family_circle_plotter -g ../data/at.gff -s ../data/at.collinearity -c family.ctl -f ../data/MADS_box_family.txt -o MADS.circle.PNG
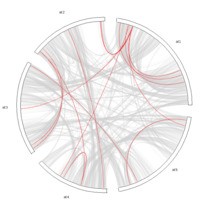 灰色背景为拟南芥全基因组的共线性结果,红色为基因家族的共线性结果;
灰色背景为拟南芥全基因组的共线性结果,红色为基因家族的共线性结果;
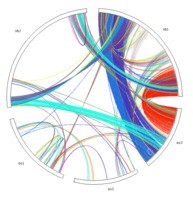
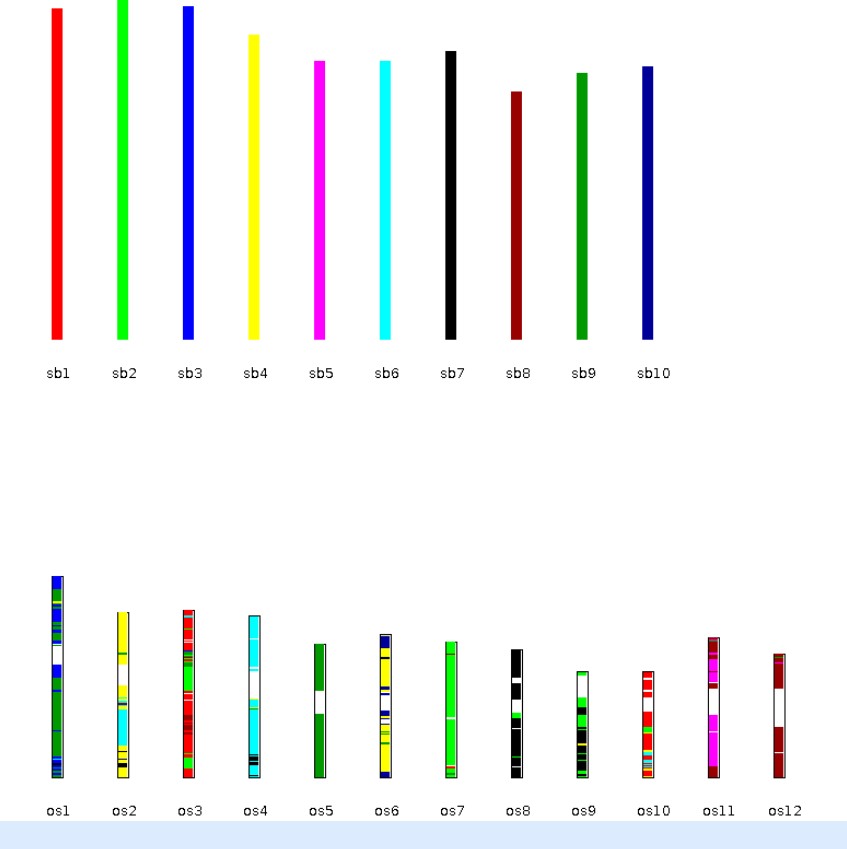
4.再来运行物种间的共线性分析(第二个例子,Rice (os) and sorghum (sb)共线性分析)
官方例子地址:http://chibba.pgml.uga.edu/mcscan2/examples/example7.php
sudo ./MCScanX data/os_sb
运行结果文件:os_sb.collinearity , os_sb.tandem
结果可视化:
首先,切换到downstream_analyses目录下,然后下载四个绘图控制文件,*ctl,之后就可以利用结果文件绘图了,代码如下:
http://chibba.pgml.uga.edu/mcscan2/examples/dot.ctl
http://chibba.pgml.uga.edu/mcscan2/examples/dual_synteny.ctl
http://chibba.pgml.uga.edu/mcscan2/examples/circle.ctl
http://chibba.pgml.uga.edu/mcscan2/examples/bar.ctl
java dot_plotter -g ../data/os_sb.gff -s ../data/os_sb.collinearity -c dot.ctl -o dot.PNG
java dual_synteny_plotter -g ../data/os_sb.gff -s ../data/os_sb.collinearity -c dual_synteny.ctl -o dual_synteny.PNG
java circle_plotter -g ../data/os_sb.gff -s ../data/os_sb.collinearity -c circle.ctl -o circle.PNG
java bar_plotter -g ../data/os_sb.gff -s ../data/os_sb.collinearity -c bar.ctl -o bar.PNG
结果图展示:
当然网站上还有其他示例代码和数据(Examples中),大家可以自行操作练习。
还是不会,可观看《基因家族分析实操课程》,里面有MCScanX的详细使用和操作说明;
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、biolinux搭建生物信息分析环境、linux命令处理生物大数据、perl入门到精通、perl语言高级、R语言画图、R语言快速入门与提高
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、 GEO芯片数据不同平台标准化 、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代fastq测序数据解读、
9.更多课程可点击:组学大讲堂视频课程
- 发表于 2018-07-09 13:58
- 阅读 ( 25827 )
- 分类:软件工具
你可能感兴趣的文章
- IF=4.3 | 秋茄树SOS1基因家族分析 701 浏览
- IF=4.9 | 多倍体割手密IAA基因家族分析鉴定 1084 浏览
- 紧扣2025国自然热点--10+研究培训总有你需要的! 4481 浏览
- 2024最新版《基因家族分析实操》直播培训开始报名了! 1557 浏览
- 基因家族分析课程更新2.0 3520 浏览
- 2023年秋季线下/直播培训火热报名中! 4126 浏览

我做sudo ./MCScanX data/at这一步时提示sudo: ./MCScanX: command not found是哪出问题了呢?谢谢
目录 路径不对,或者 软件安装不对,建议单独提问,或者学习Linux基础课