pacbio 三代全长转录组数据分析流程 Iso-Seq 3
pacbio 三代全长转录组数据分析流程 Iso-Seq 3
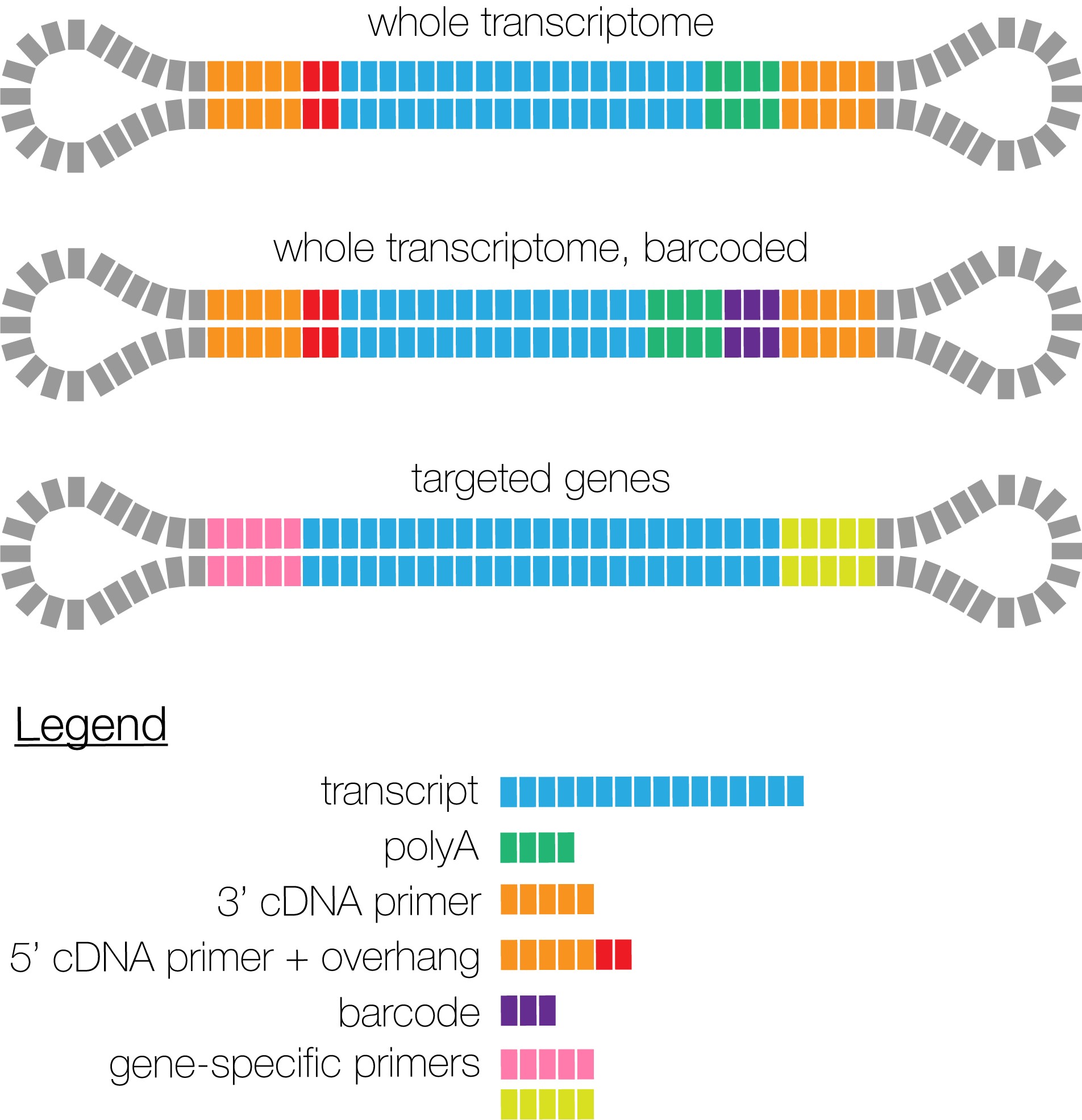
Iso-Seq 建库
Iso-Seq的建库方案有如下三类:
- 整个库都是一个样品的全长转录组,不需要加barcode区分样品
- 不同样品的全长转录组,加上不同barcode ,可以放在一起进行建库测序
- 一些靶向获得的部分基因也可以进行全长转录组的测序

Iso-Seq 3进行数据分析
Iso-Seq3 进行全长转录组的分析,运行流程如下图所示:
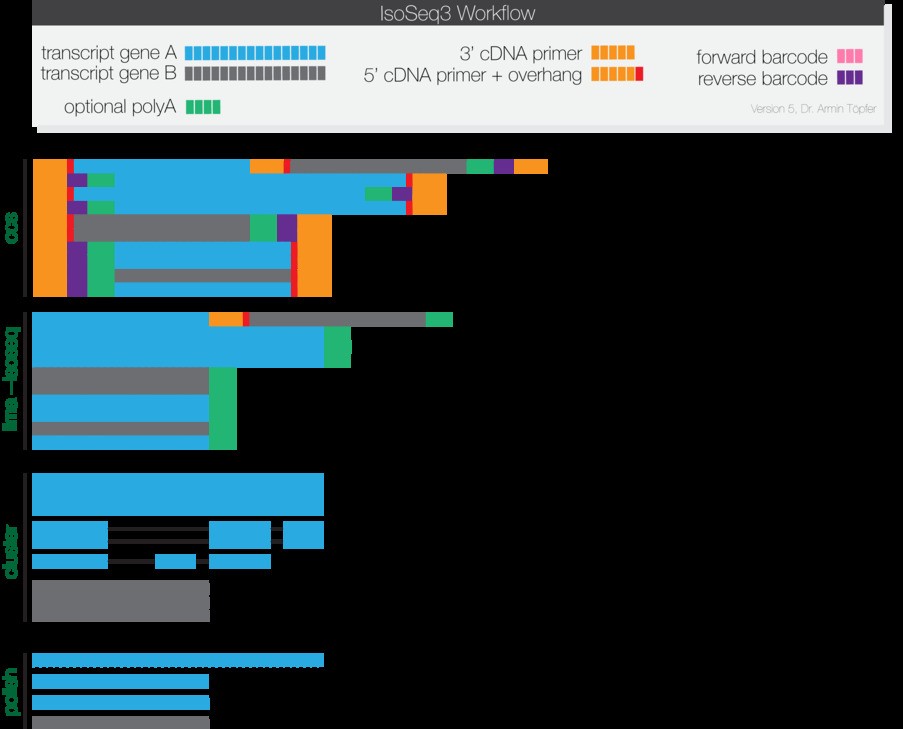
1 ccs(Circular Consensus Calling)
ccs 获取一致性的序列,要求每一条测序的reads至少有一端含有引物序列。
ccs test.subreads.bam ccs.bam --noPolish --minPasses 1
2 测序引物和barcode的去除
这一步是采用lima 来完成的,这个软件也是官方最新开发出来的程序,速度和准确度都较以往的算法有了很大的提升。还能够基于barcode序列区分不同的样品,
lima ccs.bam barcoded_primers.fasta demux.ccs.bam --isoseq --no-pbi
3 聚类(cluster)
聚类采用IsoSeq3 软件来完成,这一步会首先将ployA尾巴去除掉,并将连环结构去除掉,再对相似的序列进行聚类,最好形成全长的reads。
isoseq3 cluster demux.primer_5p--primer_3p.bam unpolished.bam --verbose
4 抛光(polish)
这一步主要是将聚类的转录本,合并成一个完整的一致性序列。
isoseq3 polish -j 16 unpolished.bam test.subreads.bam polished.bam
- 发表于 2018-08-03 10:54
- 阅读 ( 20171 )
- 分类:转录组
你可能感兴趣的文章
- Pacbio三代全长转录组(Iso-Seq ) 采用CD-HIT合并转录本 6284 浏览
- Pacbio三代无参全长转录组(Iso-Seq) 的转录本合并策略 6344 浏览
- Pacbio 数据相关的几个重要概念 6906 浏览
- Pacbio三代全长转录组(Iso-Seq ) 需要测序多少合适? 5656 浏览
- Pacbio三代全长转录组(Iso-Seq ) 纠错软件-HECIL 3782 浏览
- Pacbio三代全长转录组(Iso-Seq ) 数据纠错软件-proovread 6134 浏览
1 条评论
请先 登录 后评论
