正选择分析之 Site Models
在做遗传进化分析时,通常需要进行选择压力分析,筛选一下正选择基因。通常的做法是计算Ka/Ks,但是这种方法是比较严格的,因为正选择通常在短时间内作用于基因的几个位点,并且会被其余位点上的负选择抵消。因此,会使用其它一些方法来计算正选择基因,比如今天小编要说的Site Models。话不多说,下面就来介绍如何利用CODEML中的 Site Models 进行正选择基因分析。
Site Models 简介
Site Models是PAML软件CODEML程序的一个正选择作用分析模型,其主要观点是同一序列不同位点的ω值不同,其中ω = dN/dS,表示非同义/同义替换率。ω比率用于度量正选择作用。简而言之,ω值 < 1,= 1,> 1表示负的纯净选择,中性进化和正选择。然而所有位点的平均ω比率和所有的种系几乎从不 > 1,因为正选择不可能在漫长的时间中作用于所有的位点。因此,真正要探究的只是一些种系和一些位点所受的正选择影响。
在进行Site Models分析时,需要设置control file中的Model=0。Site Models中有多个不同的模型,可由Nssites参数指定,根据不同Model的选择设置不同的值。值得注意的是,以此可以选择多个Site Models。如Nssites=0 1 2 3 7 8。
不同的Site Models 表示什么意思?
M0即one-ratio Model,值得是所有位点的ω值是恒定的;
M1表示加假定有一部分位点的ω值为0,其他位点的ω值为1;
M2是在M1的基础上增加了第三类ω值,该类ω是通过数据计算得到的,有可能大于1;
M3假定所有位点的ω值呈简单的离散分布趋势;
M7假定所有位点的ω属于矩阵(0,1)且呈beta分布;
M8是在M7的基础上增加另一类ω值,该值可通过计算得到,可以大于1;
其对应的参数如下:
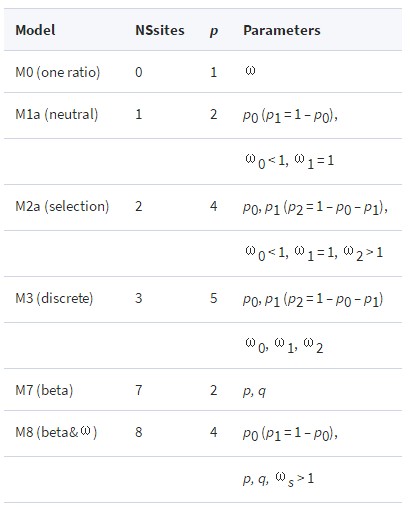
不同Model的比较可以得到什么样的结果?
在Site Models 中,M0表示one ratio for all sites, M3表示所有位点的ω值呈简单的离散分布。对于这两个模型的比较并非用于正选择作用的检测,而是用于位点间ω值是否一致的检测。
M1 and M2 以及M7 and M8是用于正选择作用的检测,作者推荐使用这两组比较进行LRT检验来验证正选择。不过Prof.Yang认为,The M1-M2 comparison 与 the M7- M8 comparison相比,更加的稳定。M7和M8俩模型计算时间会长一些,如果要进行分析的基因较多,可以考虑不比较 M7- M8。
如何检测positive sites?
在CODEML中,positive sites 的检测流程主要如图1所示:
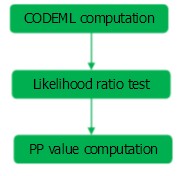
CODEML computation:主要是对control file中的命令值进行设定之后,运行CODEML程序。运行CODEML程序需要三个文件,分别是序列文件(PHYLIP格式),树文件和控制文件。控制文件示例:
seqfile = Fungi.fasta * sequence data file name
treefile = Fungi.tree * tree structure file name
outfile = mlc * main result file name
noisy = 3 * 0,1,2,3,9: how much rubbish on the screen
verbose = 0 * 1: detailed output, 0: concise output
runmode = 0 * 0: user tree; 1: semi-automatic; 2: automatic
* 3: StepwiseAddition; (4,5):PerturbationNNI; -2: pairwise
seqtype = 1 * 1:codons; 2:AAs; 3:codons-->AAs
CodonFreq = 2 * 0:1/61 each, 1:F1X4, 2:F3X4, 3:codon table
clock = 0 * 0: no clock, unrooted tree, 1: clock, rooted tree
aaDist = 0 * 0:equal, +:geometric; -:linear, {1-5:G1974,Miyata,c,p,v}
model = 0
NSsites = 0 3 1 2 7 8
* 0:one w; 1:NearlyNeutral; 2:PositiveSelection; 3:discrete;
* 4:freqs; 5:gamma;6:2gamma;7:beta;8:beta&w;9:betaγ10:3normal
icode = 0 * 0:standard genetic code; 1:mammalian mt; 2-10:see below
Mgene = 0 * 0:rates, 1:separate; 2:pi, 3:kappa, 4:all
fix_kappa = 0 * 1: kappa fixed, 0: kappa to be estimated
kappa = .3 * initial or fixed kappa
fix_omega = 0 * 1: omega or omega_1 fixed, 0: estimate
omega = 1.3 * initial or fixed omega, for codons or codon-based AAs
ncatG = 10 * # of categories in the dG or AdG models of rates
getSE = 0 * 0: don't want them, 1: want S.E.s of estimates
RateAncestor = 0 * (0,1,2): rates (alpha>0) or ancestral states (1 or 2)
Small_Diff = .45e-6
cleandata = 1 * remove sites with ambiguity data (1:yes, 0:no)?
fix_blength = 0 * 0: ignore, -1: random, 1: initial, 2: fixed, 3: proportional
Likelihood ratio test: 即对两个模型进行显著性水平比较,可以使用PAML软件自带Chi2子程序进行计算。先计算两个对应模型 Ln L 差值,并取绝对值后的数值乘以2,即2△Ln L =|Ln L 1-Ln L2|。利用Chi2程序计算P值,命令为:Chi2 2 2.03(2为自由度df,site models中df通常使用2;2.03为2△Ln L)。
输出值 prob <0.05视为显著,可认为该基因受到正选择。
PP value computation:主要是指位点后验概率的计算,该结果是显示在主输出文件 mlc中。标 "*" 号的位点为显著的正选择位点。CODEML程序中常见的计算后验概率的方法有BEB和NEB。与BEB相比,NEB在计算的过程中往往会忽略抽样误差。因此,Prof.Yang建议在读取运算结果时,可以直接将NEB result忽略,但值得注意的是,BEB只能在M2a和M8 model下运行。
参考文献:
Yang Z. PAML 4: phylogenetic analysis by maximum likelihood[J]. Molecular biology and evolution, 2007, 24(8): 1586-1591.
Yang Z. PAML: phylogenetic analysis by maximum likelihood[J]. University College London, London, 2000.
延伸阅读
- 发表于 2018-04-22 10:39
- 阅读 ( 16742 )
- 分类:软件工具
