NCBI上传高通量测序数据到SRA数据库 aspera 支持续传
当有大量的数据需要上传NCBI的SRA数据库时,网页上传很费劲,而且支持的文件数量有限;一旦链接断开就得重新来:
这里介绍用linux中aspera的命令行来执行上传数据,会更稳定:
linux当中安装与使用:
1.安装,下载地址:https://downloads.asperasoft.com/en/downloads/62
得到文件如下:
64-bit Linux: aspera-cli-x.x.x.xxx.xxxxxxx-linux-64-release.sh 32-bit Linux: aspera-cli-x.x.x.xxx.xxxxxxx-linux-32-release.sh
Note: 加可执行权限之后,直接运行这个sh就安装好了
$ chmod +x aspera-cli-x.x.x.xxx.xxxxxxx-linux-xx-release.sh $ ./aspera-cli-x.x.x.xxx.xxxxxxx-linux-xx-release.sh
安装到自己的home目录下面: Aspera CLI in the ~/.aspera/cli directory.
2. 添加环境变量
$ export PATH=~/.aspera/cli/bin:$PATH
3. 添加说明文档,然后就可以使用了:
$ export MANPATH=~/.aspera/cli/share/man:$MANPATH
下面是使用说明:
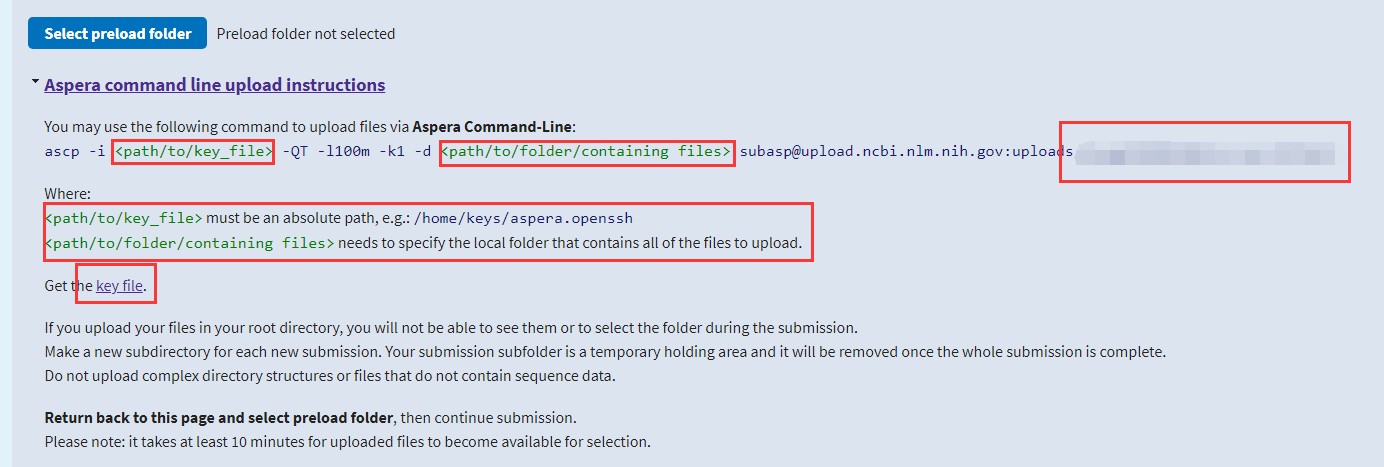
主要有三个参数:要上传的数据路径; keyfile 在网页下载得到,还有就是上传地址:
准备好这些就可以上传了:
在 windows当中安装与使用:
1.安装
下载对应windows的版本:https://downloads.asperasoft.com/en/downloads/62
aspera-cli-x.x.x.xxx.xxxxxxx-win-xxxx-32-release.zip 然后解压
然后将解压的路径下的:C:\aspera-cli-3.7.7.608.927cce8-win-v140_xp-32-release\cli\bin 目录添加到windows的环境变量Path当中;具体方法和java添加环境变量相近:https://www.omicsclass.com/article/298
2.使用和linux类似:
注意输入的路径当中不要有中文,否则或报错:
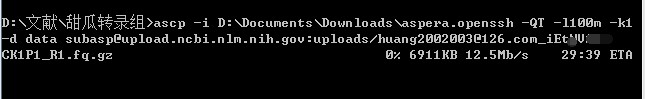
windows当中打开cmd, 输入如下命令。
最好用有线网络上传,无线网络经常掉线,笔记本电脑建议接上网线后关闭无线网络在上传;
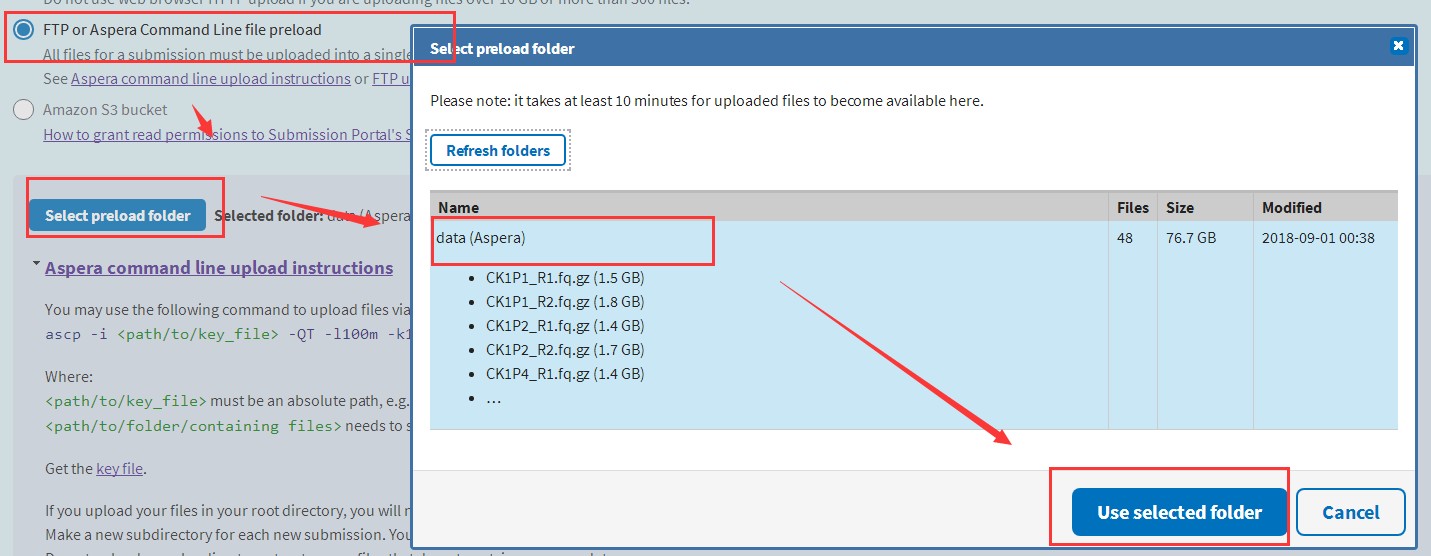
所有数据上传完成之后;最后选择,上传好的目录,提交就可以了:
NCBI详细上传视频见:《NCBI和GEO数据库上传课程》
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、perl入门到精通、perl语言高级、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、 GEO芯片数据不同平台标准化 、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读、
- 发表于 2018-08-30 20:03
- 阅读 ( 14795 )
- 分类:软件工具
你可能感兴趣的文章
- 探索基因大数据的利器——SRA Explorer 2480 浏览
- NCBI 基因组文件下载——datasets 2367 浏览
- 使用NCBI——Batch Entrez批量下载蛋白序列 11750 浏览
- NCBI的SRA Lite和SRA Normalized数据有什么区别 7562 浏览
- NCBI下载SRR并转换为fastq文件 12419 浏览
- NCBI下载物种叶绿体或线粒体基因组fasta,gff3,gtf,gb文件等 10500 浏览
相关问题
- SRA下载 1 回答
- SRA数据下载 2 回答
- 请问老师怎么将NCBI里的gbff文件转化为gff格式的文件,因为里面找不到gff文件 0 回答
- 利用命令行进行sra数据下载有如下报错 1 回答
- NCBI序列上传 1 回答
- sra文件转换为fastq文件 1 回答