多基因组基因水平共线性分析
多基因组基因水平共线性分析
在 多基因间共线性及局部基因水平的共线性 和 基因组间共线性分析 里讲了MCscan如何做基因组共线性分析,这里再说说多基因组基因水平的共线性分析。
方法是两两基因组做共线性分析。还是以桃子,葡萄和可可为例,方法如下:
$ python -m jcvi.compara.synteny mcscan grape.bed grape.peach.lifted.anchors --iter=1 -o grape.peach.i1.blocks
$ python -m jcvi.compara.synteny mcscan grape.bed grape.cacao.lifted.anchors --iter=1 -o grape.cacao.i1.blocks
$ python -m jcvi.formats.base join grape.peach.i1.blocks grape.cacao.i1.blocks --noheader | cut -f1,2,4,6 > grape.blocks
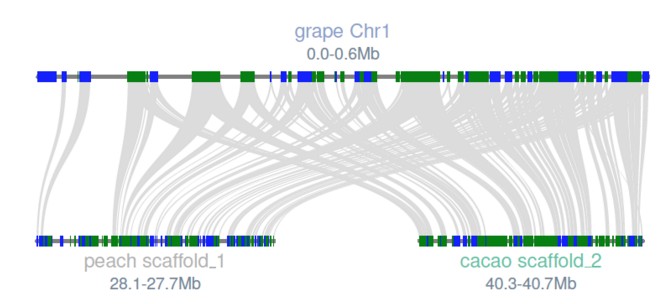
筛选关注的区域进行展示,这里展示前50列:
$ head -50 grape.blocks > blocks2
blocks2文件格式如下:
GSVIVT01012261001 . . GSVIVT01012259001 ppa005716m . GSVIVT01012258001 . . GSVIVT01012257001 ppa002846m . GSVIVT01012255001 ppa000919m Thecc1EG011472t1 GSVIVT01012253001 ppa010733m Thecc1EG011473t1 GSVIVT01012252001 ppa015194m Thecc1EG011474t1
配置文件如下:
# x, y, rotation, ha, va, color, ratio, label 0.5, 0.6, 0, center, top, , 1, grape Chr1 0.3, 0.4, 0, center, bottom, , .5, peach scaffold_1 0.7, 0.4, 0, center, bottom, , .5, cacao scaffold_2 # edges e, 0, 1 e, 0, 2
运行画图:
$ cat grape.bed peach.bed cacao.bed > grape_peach_cacao.bed
$ python -m jcvi.graphics.synteny blocks2 grape_peach_cacao.bed blocks2.layout
结果如下:
- 发表于 2018-10-19 13:39
- 阅读 ( 6460 )
- 分类:软件工具
你可能感兴趣的文章
0 条评论
请先 登录 后评论
