cutree对pheatmap返回结果实现聚类cluster划分
此前给大家介绍过pheatmap聚类后的矩阵数据重排:https://www.omicsclass.com/article/507
其中给大家介绍到了pheatmap返回结果是一个列表,其中包括行列聚类的返回结果,而这一结果又是基于hclust聚类返回的结果,这里刚好可以利用cutree函数对其聚类结果进行划分。
首先基于pheatmap参数cutree_row查看一下,将行聚类结果划分成3个cluster实现的图片结果如何(数据参考上一链接:https://www.omicsclass.com/article/507
pheatmap(mat,scale="row",cutree_rows =3)
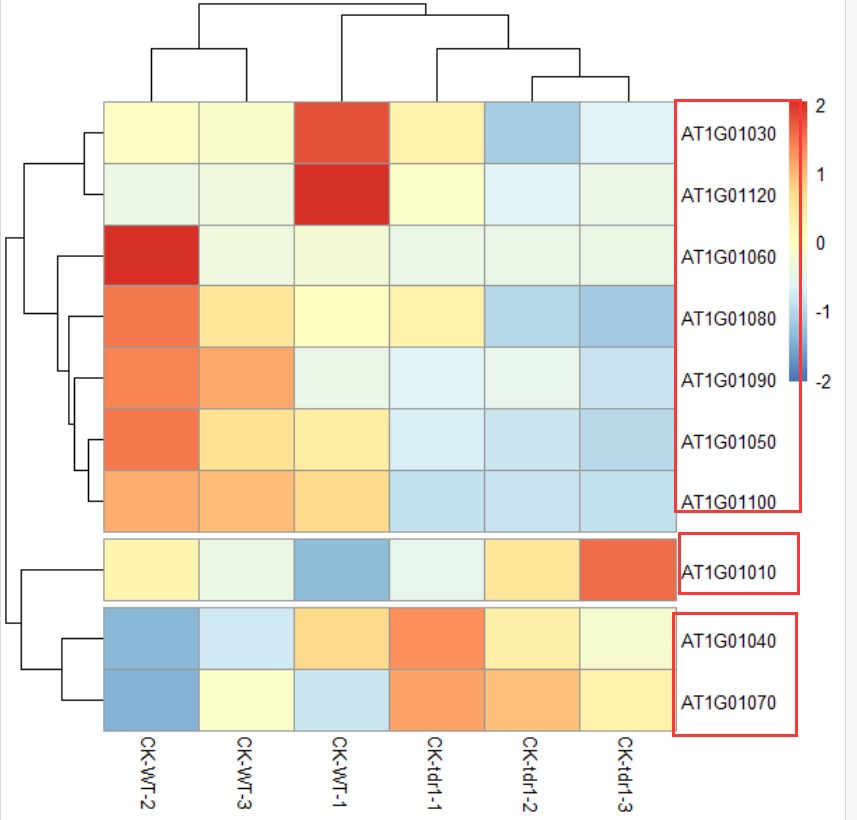 返回的图片结果图上所示,对应不同的cluster中包含不同的基因。而如何获得文本格式的内容呢?——cutree()函数实现
返回的图片结果图上所示,对应不同的cluster中包含不同的基因。而如何获得文本格式的内容呢?——cutree()函数实现
cutree()可以直接利用hclust返回结果进行cluster划分,
Usage
cutree(tree, k = NULL, h = NULL)
Arguments
| tree | a tree as produced by hclust. cutree() only expects a list with components merge,height, and labels, of appropriate content each. |
| k | an integer scalar or vector with the desired number of groups |
| h | numeric scalar or vector with heights where the tree should be cut. |
所以将热图结果中的list$tree_row作为tree输入即可:
list=pheatmap(mat,scale = "row")
row_cluster=cutree(list$tree_row,k=3)
> row_cluster
AT1G01010 AT1G01030 AT1G01040 AT1G01050 AT1G01060 AT1G01070 AT1G01080 AT1G01090
1 2 3 2 2 3 2 2
AT1G01100 AT1G01120
2 2
返回结果可以看到row_cluster即包含了哪些基因属于哪些cluster,将此结果和重排序后的矩阵整理至一起,就可以和热图对应
newOrder=mat[listp$tree_row$order,]
newOrder[,ncol(newOrder)+1]=row_cluster[match(rownames(newOrder),names(row_cluster))]
colnames(newOrder)[ncol(newOrder)]="Cluster"
> newOrder
CK-WT-1 CK-WT-2 CK-WT-3 CK-tdr1-1 CK-tdr1-2 CK-tdr1-3 Cluster
AT1G01030 5.235280 2.7707000 2.6685900 3.2263200 1.3210500 1.9672600 2
AT1G01120 173.996000 51.0019000 52.3322000 66.9486000 41.1148000 49.7169000 2
AT1G01060 1.873769 16.9090246 0.9559375 0.4774184 0.5273923 0.4333881 2
AT1G01080 30.261105 33.6571056 31.4669073 30.9689028 28.0334025 27.6215018 2
AT1G01090 86.640500 116.4700000 111.7380000 82.8809000 85.7292000 79.6027000 2
AT1G01050 118.660000 140.1430000 123.3830000 97.2229000 95.2539000 91.8525000 2
AT1G01100 1448.627845 1634.0417300 1566.5625600 648.4734402 683.4113500 647.2749880 2
AT1G01010 3.741490 7.3618000 5.8173400 5.7113100 7.9705400 10.3762000 1
AT1G01040 2.821317 1.5633947 1.9316282 3.1948090 2.6008540 2.3012776 3
AT1G01070 1.710346 0.7802436 2.7996091 4.7297117 4.3637146 3.3732144 3
此结果和图片排列对应
如果想提升自己的绘图技能,我们推荐:R语言绘图基础(ggplot2)
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、perl入门到精通、perl语言高级、R语言入门、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、GEO芯片数据标准化、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读
- 发表于 2018-10-19 14:53
- 阅读 ( 16836 )
- 分类:R
你可能感兴趣的文章
- deg_heatmap.r 差异表达基因热图展示 3342 浏览
- rocks7 集群安装笔记 3956 浏览
- pheatmap绘制热图详解 29726 浏览
- pheatmap NA/NaN/Inf 聚类错误 15952 浏览
- pheatmap绘制聚类热图 24801 浏览
- plot单独画出pheatmap返回的聚类结果(聚类树) 8137 浏览
