Vennerable包venn返回结果绘图
1、Vennerable包绘图过程 是由Venn函数 对各组数据构成的列表 进行处理返回一个Venn 对象,该对象是s4
譬如:
library(Vennerable)
data(StemCell)
w <- Venn(Sets=StemCell[1:2])
plot(w, type="squares")
查看w显示其为Venn object:
> w
A Venn object on 2 sets named
OCT4,SOX2
00 10 01 11
0 219 875 404
绘图plot的调用实际上是两个独立函数的方便包装,这两个函数首先计算绘图的几何形状,然后返回类VennDrawingand的对象,然后呈现该对象
所以代码等同于下方,获取Venn object 之后计算几何形状compute.Venn 再绘图:
library(Vennerable)
library(grid)
w <- Venn(Sets=StemCell[1:2])
w <- compute.Venn(w, type ="squares")
grid.newpage()
plot(w)
都是获得下图结果:
注意:第二种类型中,需要加载grid包,每次运行grid.newpage() 否则下一次绘图结果会叠加到上一次图片上
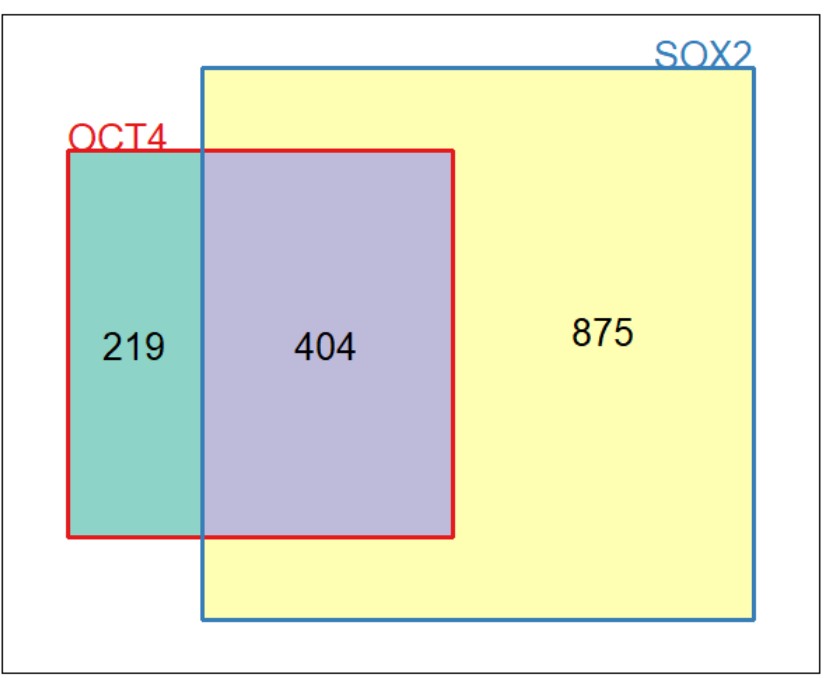
2、Vennerable中有一个函数Weights可以直接获取每个交集的对应的编号 以及交集内具体的count数,也就是图片上显示出来的数据
> Weights(w)
00 10 01 11
0 219 875 404
以上图结果为例,编号中0表示否,1表示是 及涉及与否的问题
00 表示两组都不包含的区域,也就是最外围,对应数量0
10 表示包含第一组 但是不涉及第二组,对应数量219 和图片中蓝绿色一致
01 表示不包含第一组,包含第二组,对应数据875 和图片中黄色一致
11 表示两组同时涉及,也就是交集,对应数据404 和图片茄色(?)一致
如果组别数量不同,编号会编号,如果是三组,编号为三位,以此类推。
如果想提升自己的绘图技能,我们推荐:R语言绘图基础(ggplot2)
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、perl入门到精通、perl语言高级、R语言入门、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、GEO芯片数据标准化、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读
- 发表于 2018-11-02 15:16
- 阅读 ( 4580 )
- 分类:R
你可能感兴趣的文章
- r绘图0不在原点的问题 2495 浏览
- 生物视频教程:生物统计和绘图必备的R语言技能 7108 浏览
- UpSetR包绘制多维的venn图 10249 浏览
- R包venn绘制韦恩图 5209 浏览
- rainbow函数取色 12717 浏览
