iTOL修改进化树的标签基因ID名称
iTOL如何修改标签
iTOL入门:
有些情况下我们需要对进化树的标签进行修改,iTOL可以很好地实现这一功能。如下图所示进化树:
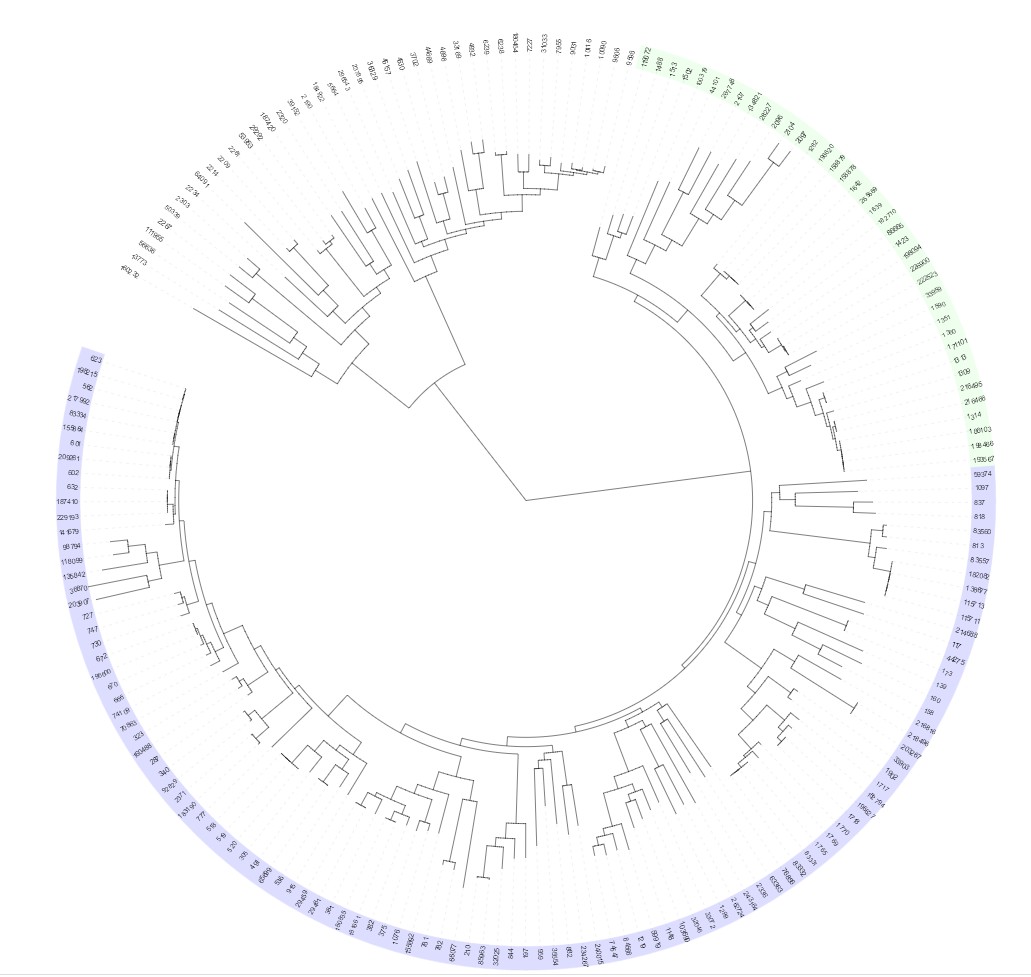
只需要添加一标签的注释文件即可,文件格式如下:
LABELS
#use this template to change the leaf labels, or define/change the internal node names (displayed in mouseover popups)
#lines starting with a hash are comments and ignored during parsing
#=================================================================#
# MANDATORY SETTINGS #
#=================================================================#
#select the separator which is used to delimit the data below (TAB,SPACE or COMMA).This separator must be used throughout this file (except in the SEPARATOR line, which uses space).
SEPARATOR TAB
#SEPARATOR SPACE
#SEPARATOR COMMA
#Internal tree nodes can be specified using IDs directly, or using the 'last common ancestor' method described in iTOL help pages
#=================================================================#
# Actual data follows after the "DATA" keyword #
#=================================================================#
DATA
#NODE_ID,LABEL
#Examples
#defined a name for an internal node
9606|9031Metazoa
#change the label for leaf node 9606
4530 Oryza sativa
45157 Cyanidioschyzon merolae
237895 Cryptosporidium hominis
184922 Giardia lamblia
56636 Aeropyrum pernix
3702 Arabidopsis thaliana
33169 Eremothecium gossypii
222523 Bacillus cereus ATCC 10987
1423 Bacillus subtilis
216816 Bifidobacterium longum
518 Bordetella bronchiseptica
29461 Brucella suis
118099 Buchnera aphidicola APS
6239 Caenorhabditis elegans
155892 Caulobacter crescentus
83560 Chlamydia muridarum
115711 Chlamydia pneumoniae AR39
115713 Chlamydia pneumoniae CWL029
138677 Chlamydia pneumoniae J138
DATA之前的部分都是格式声明,照抄即可。之后就是具体标签修改数据,数据分两列,第一列为原始标签,第二列是修改后的标签,之间以Tab键分隔。准备好数据后就可以加到进化树中了。
刘改后进化树如下:
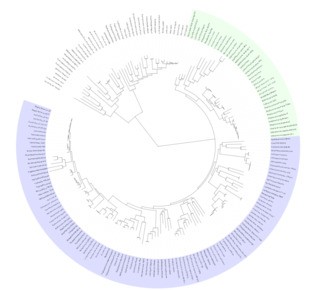
修改之后可以导出nwk格式:
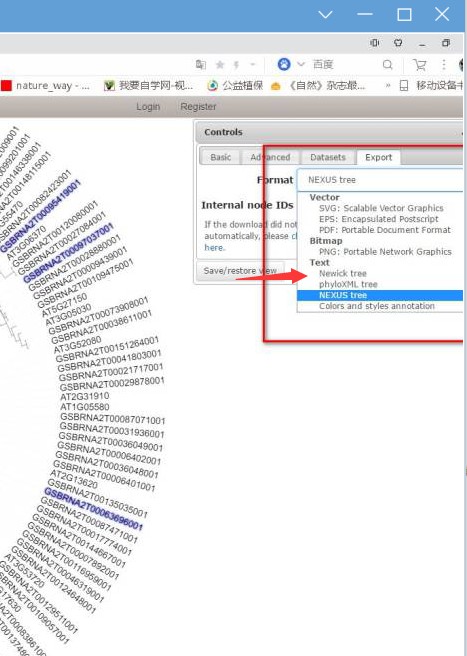
然后又可以到evolview中编辑:https://www.omicsclass.com/article/671
此外,我们在网易云课堂上有各种教学视频,有兴趣可以了解一下:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘
6. 更多学习内容:linux、perl、R语言画图,更多免费课程请点击以下链接:
- 发表于 2018-12-07 10:32
- 阅读 ( 17296 )
- 分类:软件工具
你可能感兴趣的文章
- IF=4.3 | 秋茄树SOS1基因家族分析 795 浏览
- IF=4.9 | 多倍体割手密IAA基因家族分析鉴定 1188 浏览
- 紧扣2025国自然热点--10+研究培训总有你需要的! 4575 浏览
- 2024最新版《基因家族分析实操》直播培训开始报名了! 1609 浏览
- 快速生成iTOL配置文件美化你的进化树 2485 浏览
- 基因家族分析课程更新2.0 3637 浏览
0 条评论
请先 登录 后评论
