iTOL进化树外层添加圆环
iTOL进化树外层添加圆环
我们经常会看到进化树外层有圆环用以区分样品分组等,大家可能不知道怎么画,iTOL可以很好地实现这一功能。
方法很简单,首先准备好进化树。
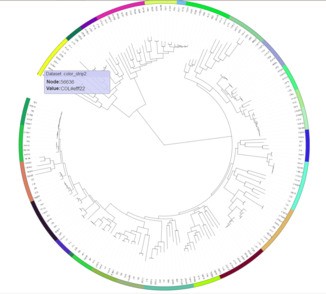
然后准备注释文件,其格式如下:
DATASET_COLORSTRIP
#lines starting with a hash are comments and ignored during parsing
#select the separator which is used to delimit the data below (TAB,SPACE or COMMA).This separator must be used throught this file (except in the SEPARATOR line, which uses space).
#SEPARATOR TAB
SEPARATOR SPACE
#SEPARATOR COMMA
#label is used in the legend table (can be changed later)
DATASET_LABEL color_strip2
#dataset color (can be changed later)
COLOR #ff0000
#optional settings
#all other optional settings can be set or changed later in the web interface (under 'Datasets' tab)
#maximum width
STRIP_WIDTH 25
#left margin, used to increase/decrease the spacing to the next dataset. Can be negative, causing datasets to overlap.
MARGIN 0
#border width; if set above 0, a black border of specified width (in pixels) will be drawn around the color strip
BORDER_WIDTH 1
BORDER_COLOR #000
#show internal values; if set, values associated to internal nodes will be displayed even if these nodes are not collapsed. It could cause overlapping in the dataset display.
SHOW_INTERNAL 0
#In colored strip charts, each ID is associated to a color. Color can be specified in hexadecimal, RGB or RGBA notation
#Internal tree nodes can be specified using IDs directly, or using the 'last common ancestor' method described in iTOL help pages
#Actual data follows after the "DATA" keyword
DATA
#ID1 value1
#ID2 value2
160232 #efff22 COL#efff22
13773 #efff22 COL#efff22
56636 #efff22 COL#efff22
111955 #efff22 COL#efff22
2287 #efff22 COL#efff22
50339 #efff22 COL#efff22
2303 #efff22 COL#efff22
2234 #efff22 COL#efff22
DATA之前的部分都是格式声明,照抄即可。之后就是添加圆环数据,数据分两列,第一列为原始标签,第二列是圆环对应颜色,之间以Ta空格分隔。准备好数据后就可以加到进化树中了。
此外,我们在网易云课堂上有各种教学视频,有兴趣可以了解一下:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘
6. 更多学习内容:linux、perl、R语言画图,更多免费课程请点击以下链接:
- 发表于 2018-12-07 15:18
- 阅读 ( 18768 )
- 分类:软件工具
你可能感兴趣的文章
- 快速生成iTOL配置文件美化你的进化树 3458 浏览
- itol添加圆环分组和节点标签时位置调整 6213 浏览
- 如何利用iTOL绘制漂亮的进化树 18804 浏览
- 使用evolview美化进化树 107107 浏览
- 批量修改进化树中的名称及添加分组名称 20994 浏览
- 轻松搞定iTOL注释文件 19059 浏览
0 条评论
请先 登录 后评论
