WGCNA筛选Biomarker
Biomarker有助于疾病诊断、判断疾病分期或者用来评价新药或新疗法在目标人群中的安全性及有效性。基于WGCNA,对疾病相关的基因表达数据构建网络,鉴定关键模块和hub基因,是筛选Biomarker的有效方法,也有助于我们进行后期湿实验验证。
接下来,就给大家讲解一篇过敏性哮喘相关文献,了解其分析思路。
研究背景
过敏性哮喘是一种具有多种分子机制的下呼吸道Th2炎症性疾病,而上下呼吸道疾病需要整体对待,因此,上呼吸道上皮细胞的基因表达相关研究对过敏性哮喘biomarker的筛选也有一定的价值。
数据来源
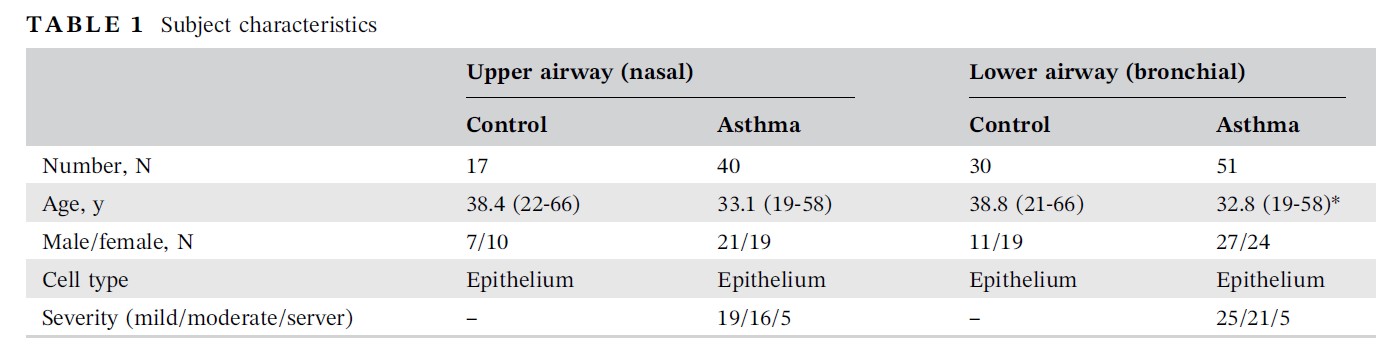
通过GEO数据库下载过敏性哮喘相关数据:GSE41861中的57个鼻黏膜上皮细胞样本(40个患者,17个健康人员),下载数据为CEL文件(原数据分上呼吸道和下呼吸道样品数据,此处仅利用上呼吸道样品数据构建网络)。
筛选差异基因
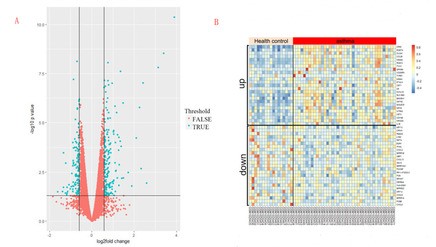
基于原始数据CEL文件,进行数据预处理,通过RMA算法进行标准化,并利用DESeq筛选差异表达的基因:最终基于1.5-fold change (P<0.05)为阈值筛选到307 DEGs(up:190;down:117,见下图A);基于1.2-fold change (P<0.05)筛选到 2804 DEGs (up:1183;down:1621),最显著上调或下调的25个DEGs表达情况见下图B。
WGCNA网络构建过程
以筛选到的2804 DEGs的表达数据进行加权基因共表达网络构建。
剔除离群样品
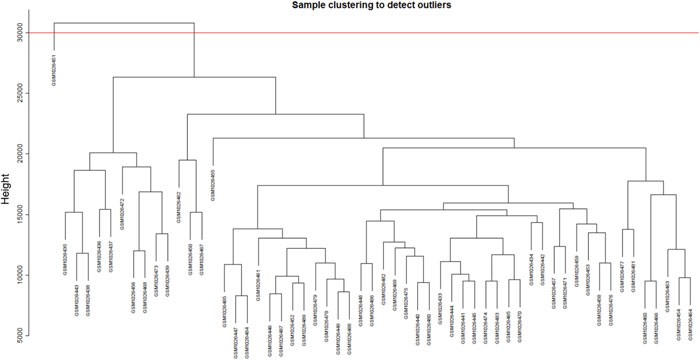
以2804 DEGs 在57个样品中的表达数据进行样品聚类分析,从而剔除离群样品,确保结果的准确性,最终剔除了GSM1026451样品(见下图)。
网络构建与模块划分
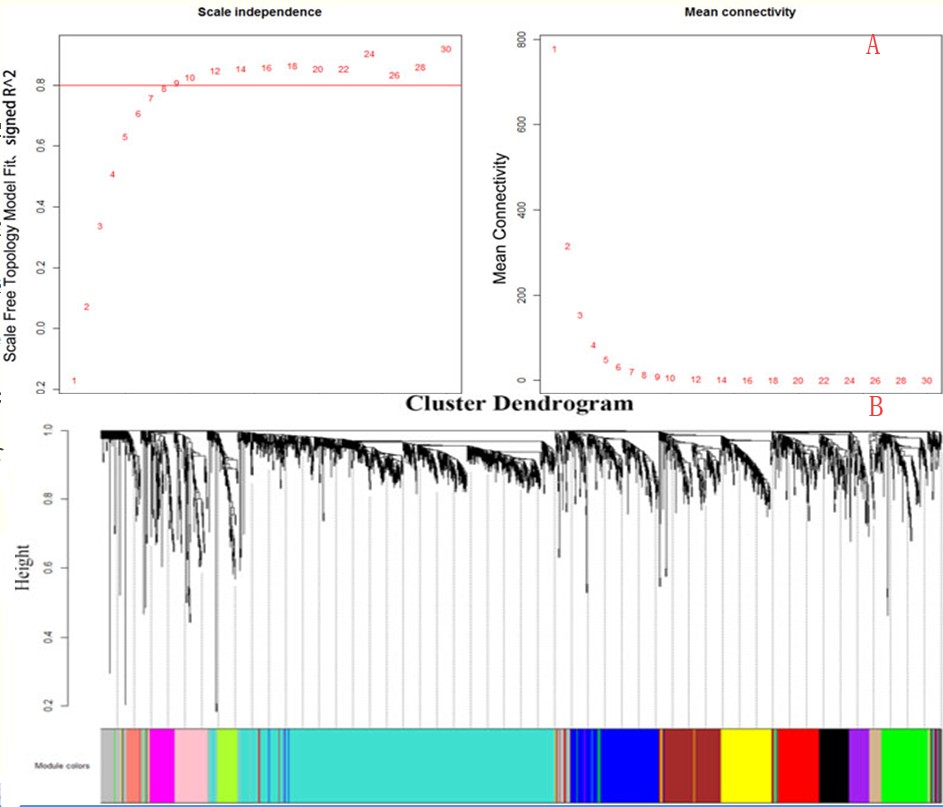
按照无尺度网络的标准确定power值为9(下图A),进行网络的构建,并划分模块,最终获得13个有效模块(下图B)
模块性状关联分析
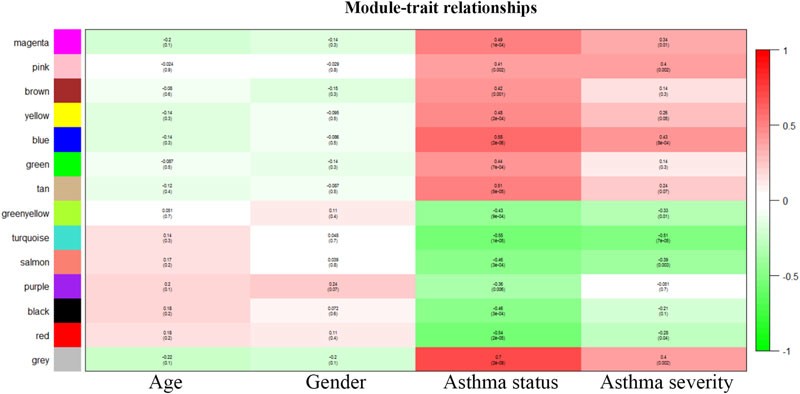
基于GSE41861性状信息(下表1),进行模块性状关联分析,寻找和性状显著相关的模块,进行后期的挖掘分析。最终发现和过敏性哮喘状态严重程度明显正相关的四个模块:magenta、pink、yellow、 blue以及明显负相关的turquoise、 salmon、 red、greenyellow(下图)。
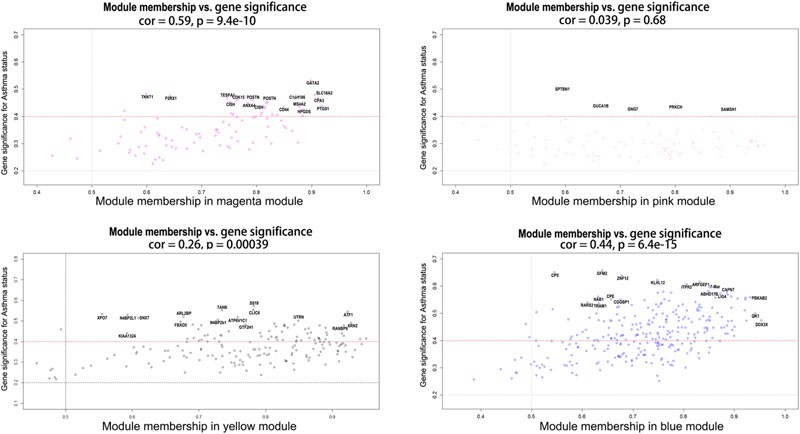
针对和临床性状正相关的四个模块进一步分析其基因显著性(GS)和模块成员关系(MM)之间的相关性,绘制对应的散点图(下图),当基因和性状之间的越相关(GS越大),并且在该模块内的作用越明显(MM越大),该基因对应的点往往出现在散点图的右上方,这类基因在模块网络内往往起到了重要作用。通过这一步的分析最终发现几个重要基因:GATA2, PTGS1, POSTN, and CD44 具有这类特征,这几个基因的功能也和过敏性哮喘紧密相关。
富集分析
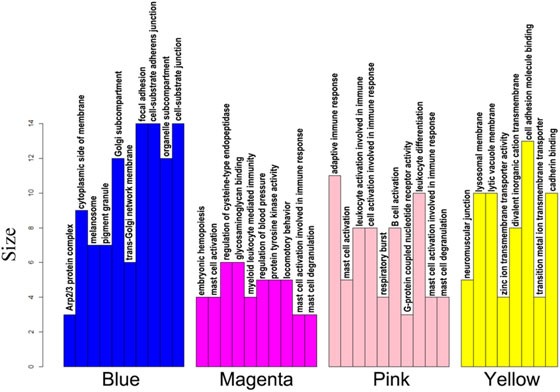
针对筛选到的四个重要模块:magenta、pink、yellow、 blue,进行GO富集分析,观察模块内基因的主要功能(下图),结果发现:magenta主要涉及肥大细胞活化与肥大细胞脱颗粒作用;pink模块主要进行适应性免疫应答;blue模块的基因主要功能是细胞的亚结构成分;yellow模块参与膜信号转导。这些功能都明显和过敏性哮喘有关。
蛋白质互作
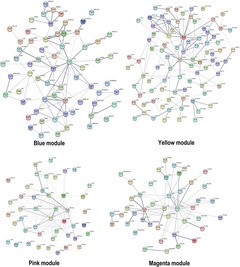
为显示四个模块内基因对应的蛋白质是否存在互作网络,利用STRING数据库进行了蛋白质互作网络分析,结果见下图。同时基于基因在模块内连通性的大小确定了6个hub基因:KIT、 KITLG、GATA2、 CD44、 PTPRC和CFTR,这些hub基因正好在蛋白质互作网络中显示。
Hub gene ROC曲线分析
针对6个hub基因,分析其在上呼吸道和下呼吸道样本中表达差异是否一致,结果发现(见下图)这些基因在上呼吸道样品存在明显的差异表达,而在下呼吸道中仅KIT、GATA2、 CD44三者存在明显差异。
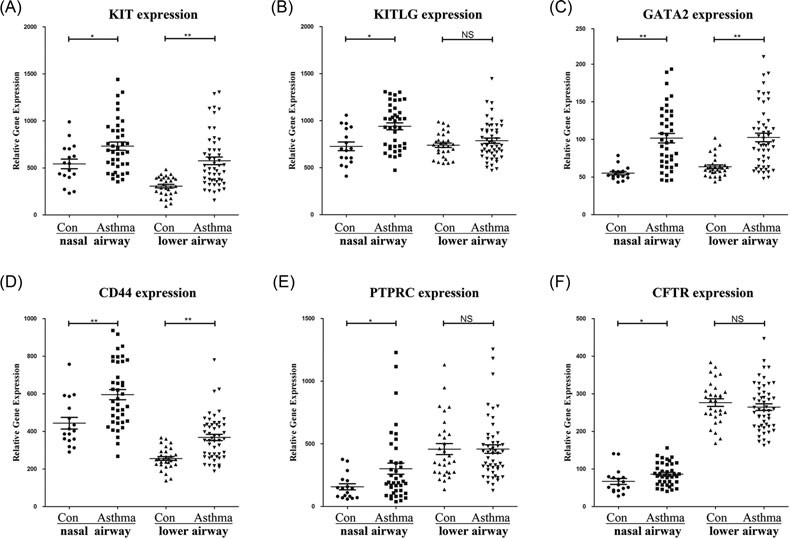
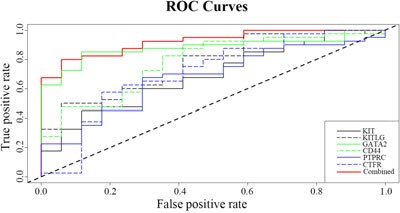
为进一步分析6个hub基因是否能作为biomarker进行疾病诊断,进行了ROC曲线分析(见下图),结果表明:针对KIT,cut-off 0.619时AUC为0.691(P=0.023),而combined结果显示cut-off 0.477时,AUC已达到了0.924(P=5.07e-07)。
总结
基于过敏性哮喘相关的基因表达数据,按照基本的WGCNA分析思路,挖掘出和过敏性哮喘严重程度具有明显关联的模块,并鉴定出了这些模块内的hub gene。这几个模块的功能富集结果表明,这些模块内的基因明显涉及到了过敏性哮喘的相关功能,并且存在比较明显的功能区分,而这些hub基因的主要功能也存在差异。结合ROC曲线分析也发现,这些hub基因作为潜在的biomarker,对后期的研究和疾病诊断具有重要价值。
参考文献:
Liu Z, Li M, Fang X, et al. Identification of surrogate prognostic biomarkers for allergic asthma in nasal epithelial brushing samples by WGCNA[J]. Journal of cellular biochemistry, 2018.
相关课程:
TCGA-基因差异表达分析、 TCGA-生存分析 、 GEO芯片数据挖掘、GEO芯片数据标准化、WGCNA-加权基因共表达网络分析
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、perl入门到精通、perl语言高级、R语言入门、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、GEO芯片数据标准化、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读
- 发表于 2018-12-07 16:24
- 阅读 ( 8267 )
- 分类:文献解读
你可能感兴趣的文章
- 用AI写代码批量整理GEO数据库中的单细胞数据 365 浏览
- 单细胞转录组数据挖掘流程记录-头颈癌(HNSCC)(GSE181919) 878 浏览
- R语言中转换成dummy dummy-variables 虚拟变量 1822 浏览
- 细胞焦亡、铁死亡及铜死亡等基因集 3883 浏览
- 记录一个数据下载:GSE12417 1384 浏览
- 生信挖掘GEO+TCGA 文献速览 1899 浏览
相关问题
- 大家好啊,新手一枚,请多关照哈 0 回答
- wgCNA一个模块中找hub基因 我把cytoScapeinput-edges文件输入cytoscape中 用cytohubba计算 但是每次出来的结果都不一样 试了很多遍也不行 求解答 谢谢! 0 回答
- 我得到转录组分析的wgcna数据需通过cytoscape筛选关键模块的hub基因,边的权重应该设定多少来筛好 1 回答
- WGCNA中过滤低表达量(0.5)时datExpr0=datExpr0[1:n,datExpr0[n+1,] > meanFPKM]不成功,且变量从上步30769变成了0. ? 2 回答
- 跑WGCNA,用的独享服务器,报错 1 回答
- 我想咨询一下wgcna问题 0 回答
