perl输出基因的位置信息按照基因所在染色体,和位置信息排序
perl输出基因的位置信息按照基因所在染色体,和位置信息排序
我们在整理基因组的gff文件,想输出基因的位置信息,以及基因所对应的多个转录本信息,需要对基因按照染色体排序,这里使用到了perl里面hash按照值来排序,而且还用了两个值基因型排序。示例代码如下,以下代码可以从gff文件当中提取所有基因的位置信息以及对应的多个转录本信息:
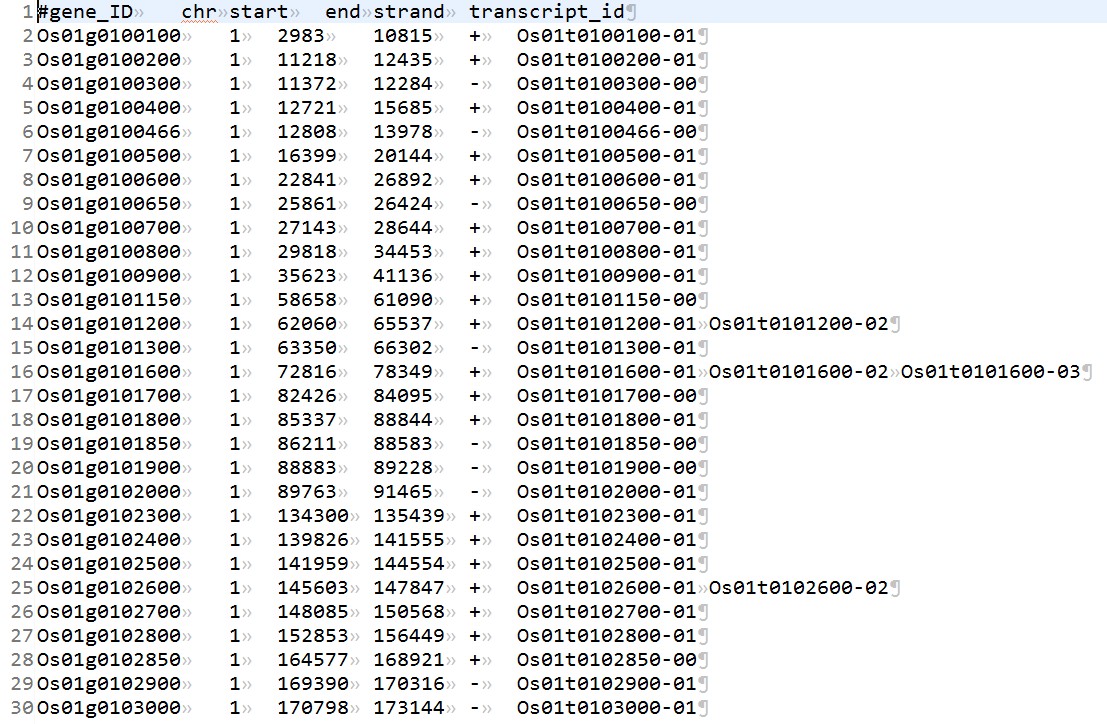
perl代码如下:
#!/usr/bin/perl -w
use strict;
use Cwd qw(abs_path getcwd);
use Getopt::Long;
use Data::Dumper;
die "perl $0 <gff> <outfile>" unless(@ARGV==2);
my$gff=$ARGV[0];
my%gene=();
my%gene_region=();
my%mRNA2Gene=();
my%Gene2mRNA=();
open IN,"$gff" or die "$!";
open OUT ,">$ARGV[1]" or die "$!";
print OUT "#gene_ID\tchr\tstart\tend\tstrand\ttranscript_id\n";
while(<IN>){
chomp;
next if (/^#/);
my@tmp=split(/\t/);
if($tmp[2] =~/^gene/){
my($id)=($tmp[8]=~/ID=([^;]+)/);
$gene{$id}=1;
$gene_region{$id}=[$tmp[0],$tmp[3],$tmp[4],$tmp[6]];
#print "gene:$id\n";
#my$gene_chr->{$id}=$tmp[0];
}
if($tmp[2] =~/mRNA|transcript/i){
my($id)=($tmp[8]=~/ID=([^;]+)/);
my($pid)=($tmp[8]=~/Parent=([^;]+)/);
if(exists $gene{$pid}){
push @{$Gene2mRNA{$pid}},$id;
}
#print "mRNA:$id\n";
}
}
close(IN);
#多层排序,先按染色体排序,再按基因位置排序
for my$id(sort {$gene_region{$a}->[0] cmp $gene_region{$b}->[0]
or $gene_region{$a}->[1] <=> $gene_region{$b}->[1] } keys %gene_region){
print OUT "$id\t".join("\t",(@{$gene_region{$id}},sort @{$Gene2mRNA{$id}}) )."\n";
}
close(OUT);
更多生物信息perl语言学习视频课程:《perl入门》《perl高级编程》
- 发表于 2018-12-13 22:26
- 阅读 ( 6171 )
- 分类:perl
你可能感兴趣的文章
- GFF或GTF格式转bed 1490 浏览
- gbff 格式注释文件转换成gff3注释文件格式 10380 浏览
- 解决基因组当中蛋白质序列ID和gff中ID不一致的问题 5116 浏览
- 根据GFF文件提取基因组中基因的蛋白,CDS,CDNA序列 3155 浏览
- GFF文件格式不标准,第三列只有GENE处理方法 2907 浏览
- GFF文件格式不标准,第三列只有mRNA处理方法 8655 浏览
相关问题
- 搜集的gff3文件;第一列不同,有的是chr,有的是id;这个影响吗? 2 回答
- 老师,我在做饭基因家族分析,我搜集的基因组的gff3文件,第一列有的是chr有的是基因id,这个会影响后续结果嘛? 2 回答
- 老师,为什么我的cds和pep文件导不出来呀?导出来报错,并且文件内容无。最后一张照片时我退出重进后的历史命令 2 回答
- 基因家族分析2.0,做基因结构分析时报错,完全按照提供指令做的 1 回答
- 老师,我这个是桑树的fasta和gff文件,不是从官网下载的。假如各个网站都找不到我想要的物种的基因组文件,只有手头这个文件,我要怎么做才能和教程的代码连接上呀?或者说碰到这种目标物种查不到的情况可以怎么做呢? 1 回答
- 老师,下面是报错、染色体信息、gff文件中的染色体和命令行 2 回答
0 条评论
请先 登录 后评论
