批量修改进化树中的名称及添加分组名称
通常进化树中的枝名称一般为物种名或基因名或样品名等(如下图所示),但是,有时候我们做完进化树之后发现先前命名的进化树的枝的名字不是很好,我们想整体修改一下。如果从头修改需要重新分析那就太麻烦了。这里我们可以通过iTOL网站提供的标签修改功能,批量的修改名字:
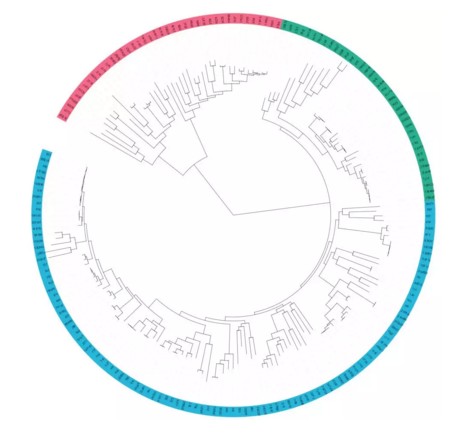
使用的工具是iTOL这款在线软件,可点这里 绘一棵超酷炫的系统发育树 了解iTOL的使用方法。基础的使用方法就不再讲述了,只对配置文件进行说明。
配置文件准备
首先来看一下所需要的文件的格式:
LABELS
SEPARATOR TAB
DATA
#NODE_ID,LABEL
4530 Oryza sativa
45157 Cyanidioschyzon merolae
237895 Cryptosporidium hominis
184922 Giardia lamblia
56636 Aeropyrum pernix
3702 Arabidopsis thaliana
33169 Eremothecium gossypii
222523 Bacillus cereus ATCC 10987
1423 Bacillus subtilis
216816 Bifidobacterium longum
格式说明:
注释文件中以"#"开头的行是注释行,只起说明作用,iTOL不会读取该行的内容。
LABELS:注释文件数据类型声明,这里声明数据集是对进化树标签进行修改。
SEPARATOR TAB:指定数据分隔符,即数据列与列之间使用Tab分隔。也可指定分隔符为空格,需将"TAB"改为"SPACE"。(建议使用Tab分隔,这样修改的标签中可以使用空格)
如果没有特殊设置,以上部分通常不需要改,直接拷贝使用即可。
DATA 之后的行就是所有标签修改的数据信息。数据分为两列,第一列为进化树枝的原来的名称,第二列是修改后的名称。
配置文件美化
按照以上格式准备好注释文件,准备好文件后直接将其拖到iTOL进化树展示所在的位置,就能自动修改进化树的标签了。
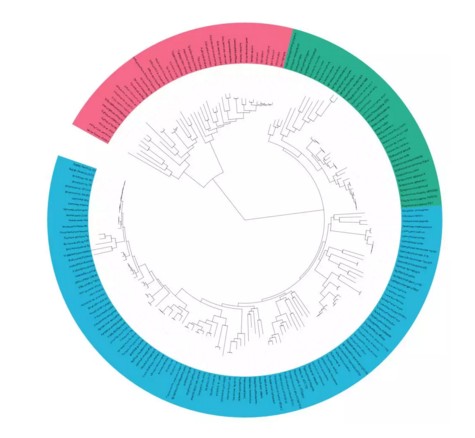
添加外部分组名称
除此之外,还可以在进化树中添加外部分组名称,同样可以展示样品的更多信息。
添加外部标签所需要的注释文件格式如下:
DATASET_TEXT
#SEPARATOR TAB
#SEPARATOR SPACE
SEPARATOR COMMA
DATASET_LABEL,example text dataset 2
COLOR,#ff00ff
MARGIN,0
LABEL_ROTATION,0
DATA
#the following fields are possible for each node:
#ID,label,position,color,style,size_factor,rotation
9606,Homo sapiens,-1,#ff0000,bold,1.5,45
4932,Eukaryota,-1,#000000,italic,2,90
160,Bacteria,-1,#0000ff,bold,4,90
2234,Archaea,-1,#0000ff,normal,3,-45
727,Something is here with a long label,-1,#ff0000,bold-italic,1,0
格式说明:
配置文件中以"#"开头的行是注释行,只起说明作用,iTOL不会读取该行的内容。
DATASET_TEXT:配置文件数据类型声明,这里声明数据集是对进化树添加文本信息。
SEPARATOR COMMA:指定数据分隔符,即数据列与列之间使用逗号分隔。
COLOR,#ff00ff:指定数据集初始颜色,可在后面进行更改。
MARGIN 0:指定外部标签与进化树之间的距离,可为负数,会导致与进化树重叠。
LABEL_ROTATION,0指定外部标签的初始角度。
如果没有特殊设置,以上部分通常不需要改,直接拷贝使用即可。
DATA 之后的行就是所有外部分组的数据信息。数据分为七列,第一列为进化树枝节点ID,第二列是外部分组名称,第三列是名称位置:"-1"代表位于外部,第四列是分组名称颜色,第五列是字体设置,第六列是字体大小,最后一列是名称的角度。
同样的,按照以上格式准备好注释文件后,直接将其拖到iTOL进化树展示所在的位置,就能自动在进化树中添加外部分组名称了。如下所示:
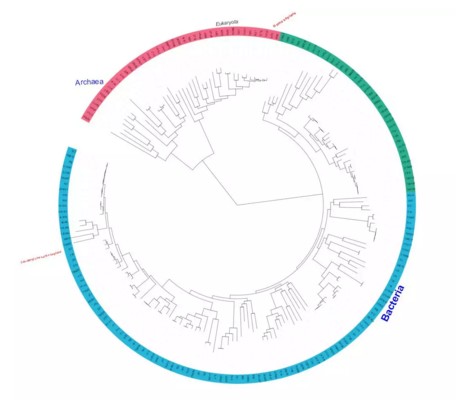
以上操作介绍完了,怎么样?是不是很简单?有兴趣的小伙伴自己动手试试吧!
最后祝您科研愉快!
此外,我们在网易云课堂上有各种教学视频,有兴趣可以了解一下:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘
6. 更多学习内容:linux、perl、R语言画图,更多免费课程请点击以下链接:
- 发表于 2018-12-29 15:18
- 阅读 ( 18855 )
- 分类:软件工具
你可能感兴趣的文章
- 给进化树标定化石时间 2301 浏览
- 快速生成iTOL配置文件美化你的进化树 2485 浏览
- itol添加圆环分组和节点标签时位置调整 4974 浏览
- 如何用R语言绘制进化树+热图 10449 浏览
- 进化树构建的方法及原理详解 43440 浏览
- 如何利用iTOL绘制漂亮的进化树 17203 浏览
