rainbow函数取色
R语言中如何依据rainbow取色?
rainbow()可以获取彩虹色板
rainbow(n, s = 1, v = 1, start = 0, end = max(1, n - 1)/n, alpha = 1)
例如,基于彩虹色渐变获取10个颜色,依次由red、yellow、green、cyan、green、blue 、magenta等顺序细化而成:
> rainbow(10)
[1] "#FF0000FF" "#FF9900FF" "#CCFF00FF" "#33FF00FF" "#00FF66FF" "#00FFFFFF" "#0066FFFF"
[8] "#3300FFFF" "#CC00FFFF" "#FF0099FF"
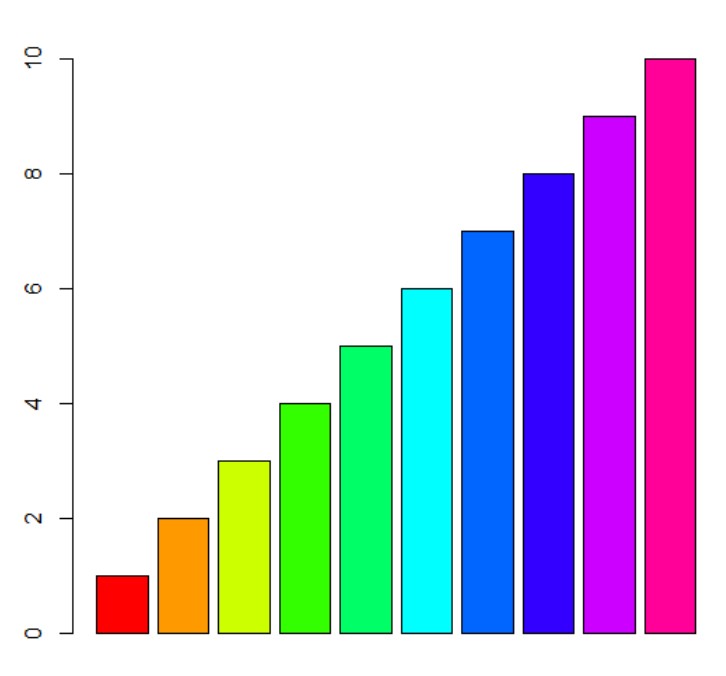
对应的颜色显示如下(绘制十个递增的柱子并填充对应颜色):
> barplot(1:10,col=rainbow(10))
如何获取彩虹色带的一部分,而不是从红色到结束位置?可以基于参数start和 end进行设置:
With rainbow, the parameters start and end can be used to specify particular subranges of hues. The following values can be used when generating such a subrange: red = 0, yellow = 1/6, green = 2/6, cyan = 3/6, blue = 4/6 and magenta = 5/6.
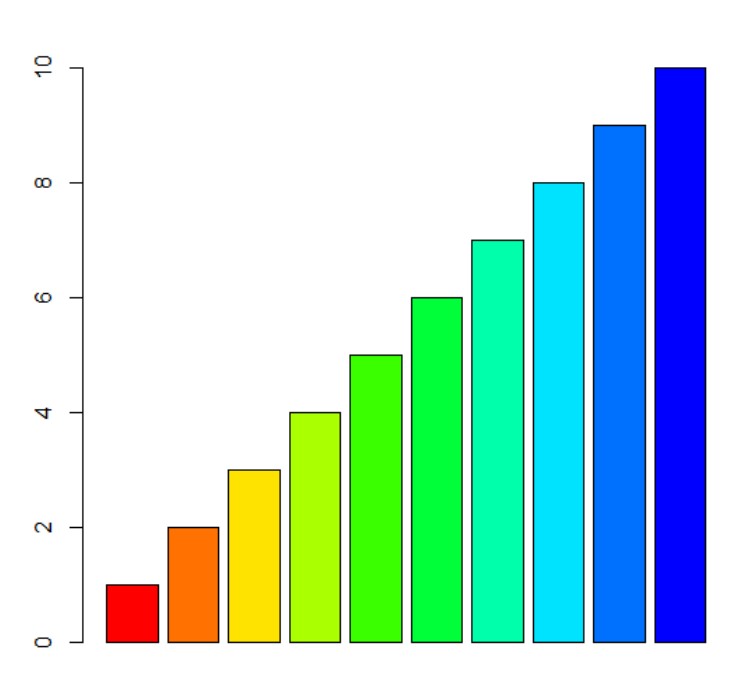
譬如取色由红色到蓝色:
> rainbow(10,start = 0,end=4/6)
[1] "#FF0000FF" "#FF7100FF" "#FFE300FF" "#AAFF00FF" "#39FF00FF" "#00FF39FF" "#00FFAAFF"
[8] "#00E3FFFF" "#0071FFFF" "#0000FFFF"
> barplot(1:10,col=rainbow(10,start=0,end=4/6))
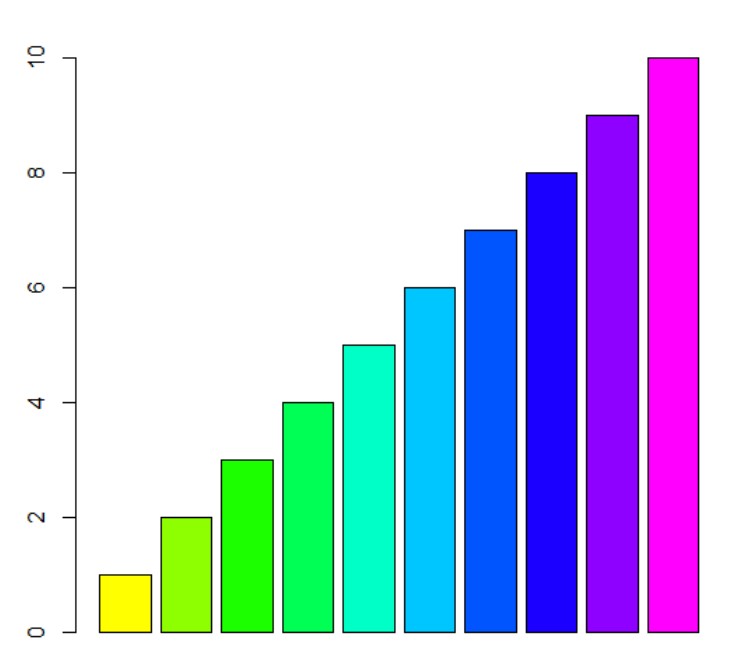
 又或者取中间颜色,start=1/6,end=5/6
又或者取中间颜色,start=1/6,end=5/6
> barplot(1:10,col=rainbow(10,start=1/6,end=5/6))
推荐课程:R语言画图
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、perl入门到精通、perl语言高级、R语言入门、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、GEO芯片数据标准化、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读
- 发表于 2019-01-03 17:43
- 阅读 ( 12718 )
- 分类:R
你可能感兴趣的文章
- r绘图0不在原点的问题 2496 浏览
- 生物视频教程:生物统计和绘图必备的R语言技能 7108 浏览
- colorRamPalette函数生成调色板 4959 浏览
- Vennerable包venn返回结果绘图 4580 浏览
- rgb函数选择颜色(并控制深浅) 9707 浏览
相关问题
- XP-EHH 0 回答
- 老师您好,我想问一下下面这个图是怎么画出来的呢 1 回答
- 聚类热图(heatmap)如何绘制? 1 回答
- 韦恩图(Venn)如何绘制? 1 回答
