如何批量分析蛋白序列domain-SMART数据库
SMART(http://smart.embl.de/)是我们常用来进行domain搜索和蛋白质注释的数据库,它集成了很多蛋白结构预测和功能分析的工具,比如可以预测蛋白的一些二级结构:跨膜区(Transmembrane segments)、复合螺旋区(coiled coil regions)、信号肽(Signal peptides)、蛋白结构域(PFAM domains)等 。
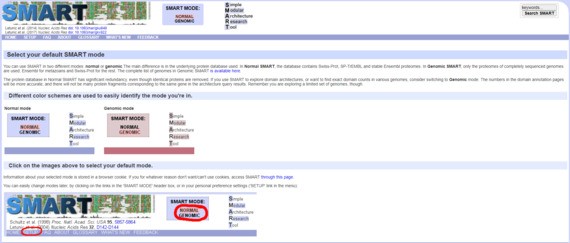
进入SMART主页,就会看到SMART有两种不同的模式:normal 和genomic,两种模式主要是用的数据库不一样:1. Normal SMART 用的数据库 Swiss-Prot、 SP-TrEMBL 和Stable Ensembl proteomes;2. Genomic SMART 用的是全基因组序列 ,两种模式可以点击下图红色剪头所指normal /genomic进行切换。
下面具体介绍下通过Uniprot/Ensembl ID 或者序列在SMART数据库中单序列/批量序列检索操作方法。
单序列domian搜索
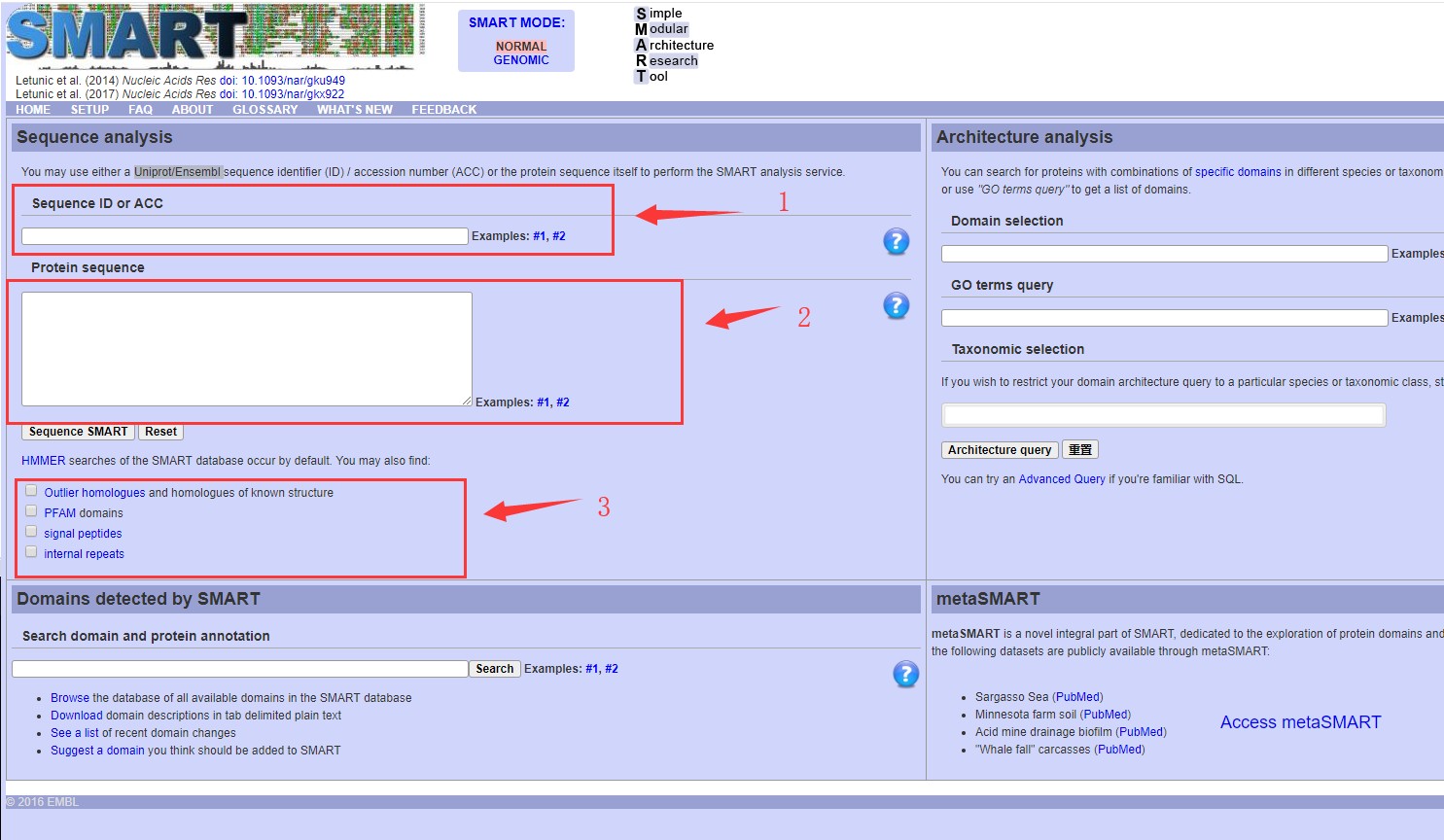
直接通过网站链接进入主页(如下图),输入需要鉴定的序列ID,包括Uniprot/Ensembl ID等(下图1处),或者直接输入序列(下图2处),并进行参数设置(下图3处)。
譬如小编直接利用拟南芥AT1G66550.1蛋白序列提交之后,获得如下图的分析结果。
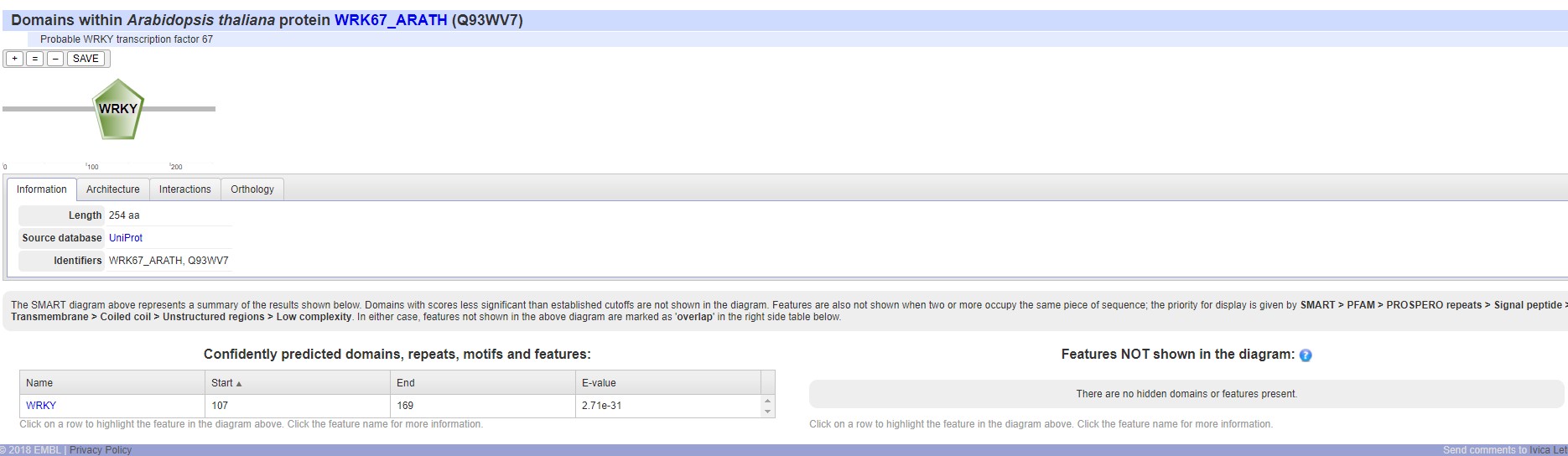
批量搜索
SMART批量搜索的设置隐藏的有点深,而这是科研工作的我们最需要的!批量搜索可以通过主页帮助(help)的位置进入(见下图),或者直接利用链接:http://smart.embl.de/smart/batch.pl。
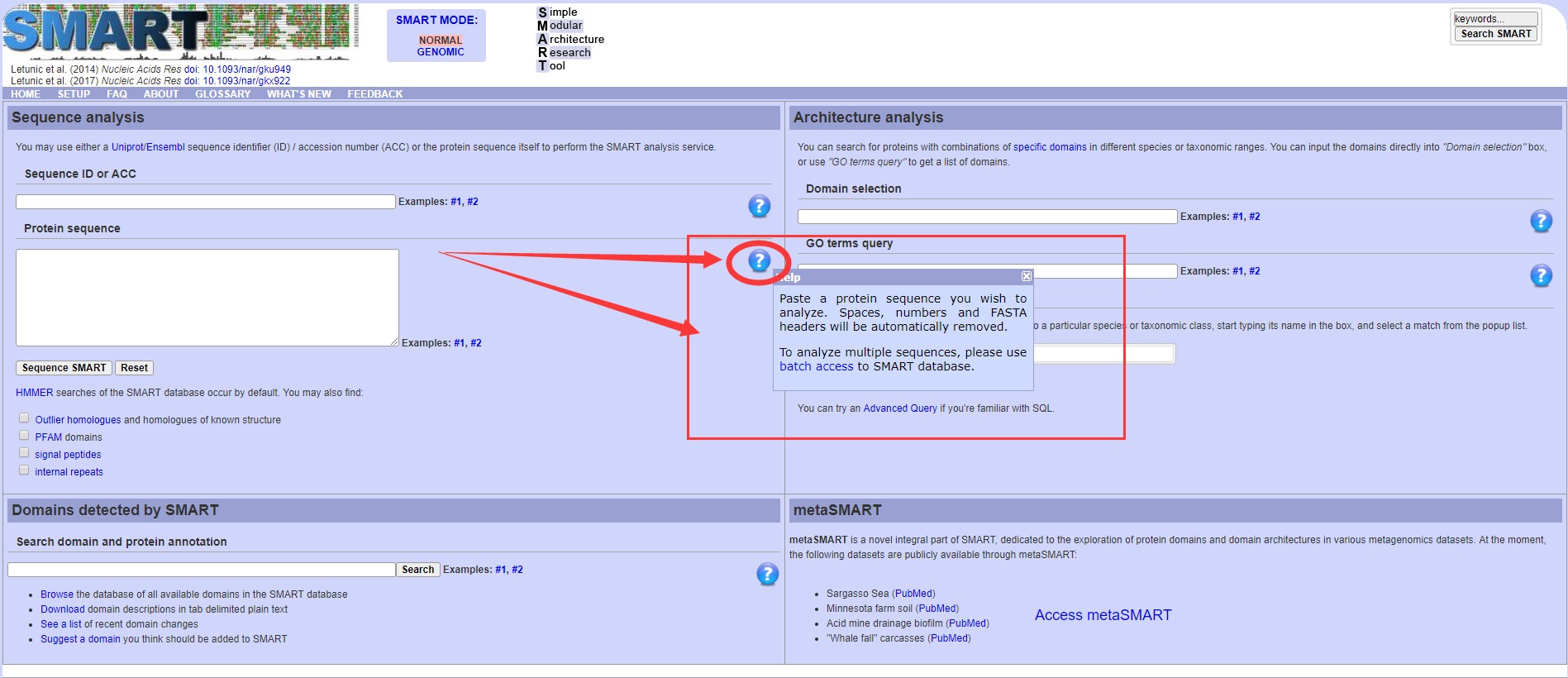
选择batch access进入批量分析页面(如下图),可以通过复制粘贴相关ID/序列 或者直接上传对应的文件,之后设置参数提交进行分析即可(参数可以选择仅输出分析结果文本格式)。
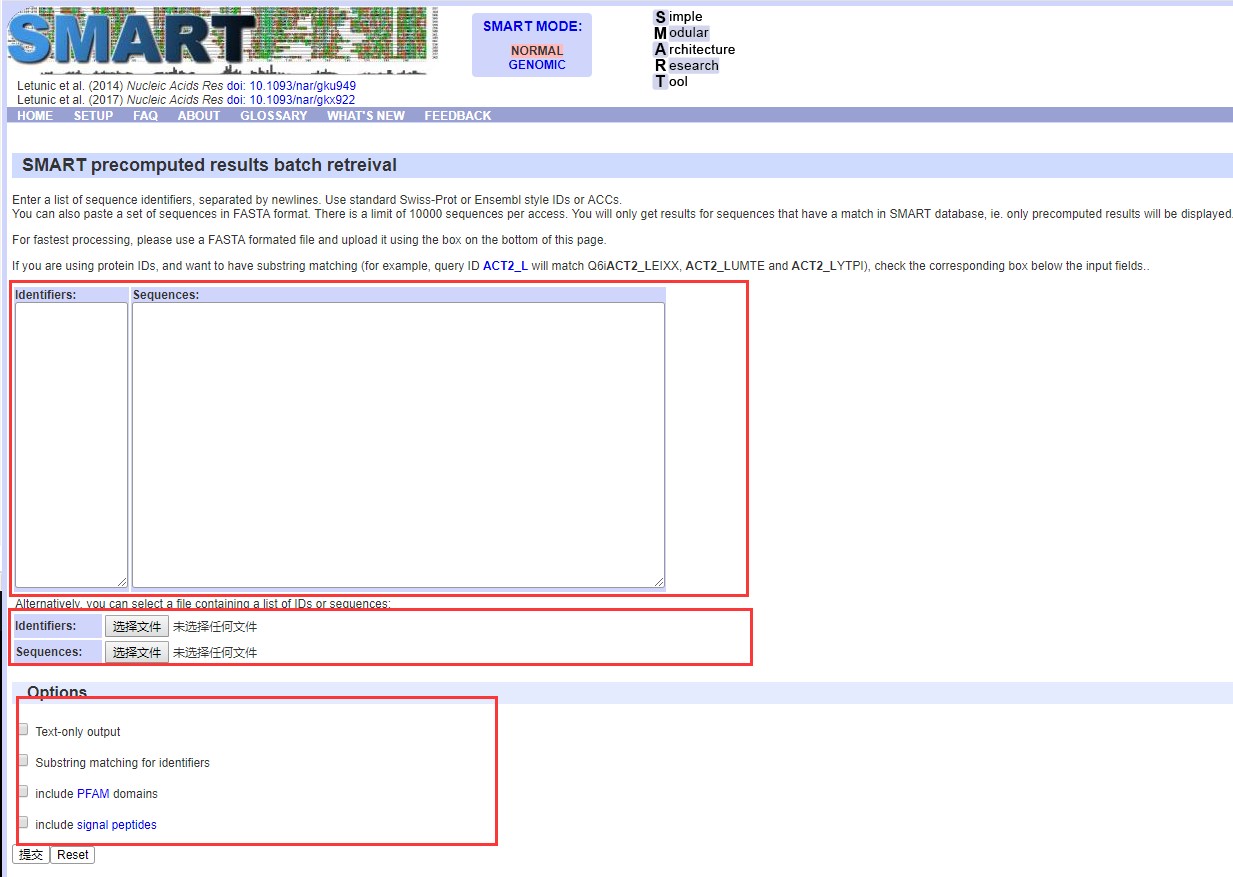
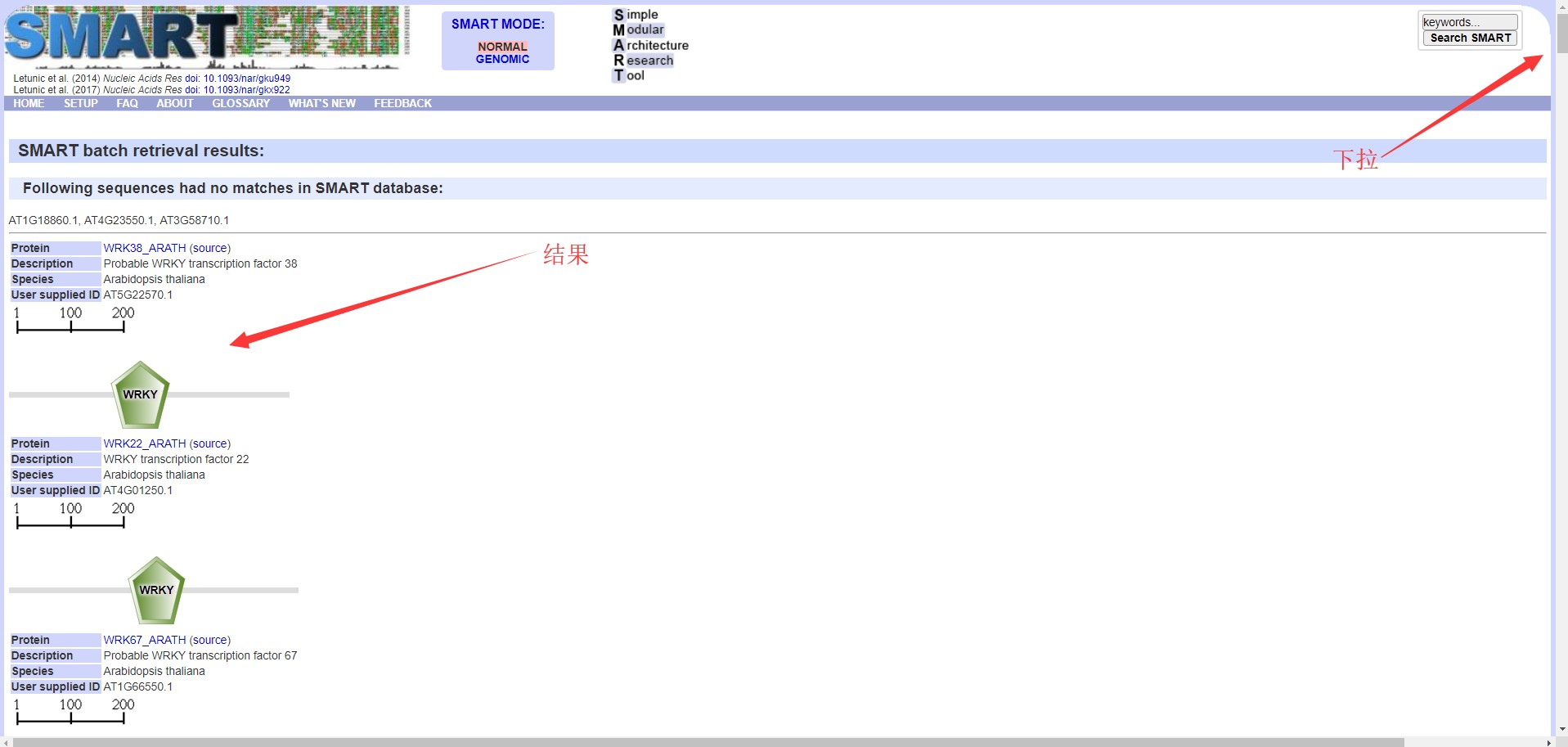
譬如小编把拟南芥的一部分蛋白序列提交之后结果如下图,移动滚动条可以对所有分析结果进行浏览。
推荐课程:
基因家族分析实操课程、基因家族文献思路解读
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、perl入门到精通、perl语言高级、R语言入门、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、GEO芯片数据标准化、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读
- 发表于 2019-01-18 10:38
- 阅读 ( 42930 )
- 分类:基因家族分析
你可能感兴趣的文章
- 鉴定domain同时分析蛋白互作 7437 浏览
- 一个预测基因保守结构域网站 15337 浏览