微生物多样性alpha分析看不明白?看这里!
在微生物多样性分析的报告中主要包括五个部分:Alpha多样性分析、Beta多样性分析、物种组成分析、进化关系分析、差异分析,其中Alpha多样性分析是生态学中生物多样性的一个重要的组成部分,也是比较基础的一部分。
Alpha多样性是指一个特定区域或生态系统内的多样性,是反映丰富度和均匀度的综合指标。Alpha多样性主要与两个因素有关:一是种类数目,即丰富度;二是多样性,群落中个体分配上的均匀性。群落丰富度(Community richness)的指数主要包括Chao1指数和ACE指数。群落多样性(Community diversity)的指数,包括Shannon指数和Simpson指数。另外,还有测序深度指数Observed spieces 代表OTUs的直观数量统计, Good’s coverage 指计算加入丰度为1 的OTUs数目,加入低丰度影响。
Alpha多样性各指数的意义
Chao1:是用chao1 算法估计群落中含OTU 数目的指数,chao1 在生态学中常用来估计物种总数,由Chao (1984) 最早提出。Chao1值越大代表物种总数越多。Schao1=Sobs+n1(n1-1)/2(n2+1),其中Schao1为估计的OTU数,Sobs为观测到的OTU数,n1为只有一条序列的OTU数目,n2为只有两条序列的OTU数目。Chao1指数越大,表明群落的丰富度越高。
Ace:是用来估计群落中含有OTU 数目的指数,同样由Chao提出(Chao and Yang, 1993),是生态学中估计物种总数的常用指数之一。默认将序列量10以下的OTU都计算在内,从而估计群落中实际存在的物种数。ACE指数越大,表明群落的丰富度越高。
Shannon:(Shannon, 1948a, b)综合考虑了群落的丰富度和均匀度。Shannon指数值越高,表明群落的多样性越高。
Simpson:用来估算样品中微生物的多样性指数之一,由Edward Hugh Simpson ( 1949) 提出,在生态学中常用来定量的描述一个区域的生物多样性。Simpson 指数值越大,说明群落多样性越低。辛普森多样性指数=1-随机取样的两个个体属于不同种的概率。
alpha多样性指数具体描述如下: 更多指数介绍:http://scikit-bio.org/docs/latest/generated/skbio.diversity.alpha.html
计算菌群丰度(Community richness)的指数有:
Chao - the Chao1 estimator (http://scikit-bio.org/docs/latest/generated/generated/skbio.diversity.alpha.chao1.html#skbio.diversity.alpha.chao1);
ACE - the ACE estimator (http://scikit-bio.org/docs/latest/generated/generated/skbio.diversity.alpha.ace.html#skbio.diversity.alpha.ace);
计算菌群多样性(Community diversity)的指数有:
Shannon - the Shannon index (http://scikit-bio.org/docs/latest/generated/generated/skbio.diversity.alpha.shannon.html#skbio.diversity.alpha.shannon);
Simpson - the Simpson index (http://scikit-bio.org/docs/latest/generated/generated/skbio.diversity.alpha.simpson.html#skbio.diversity.alpha.simpson);
测序深度指数有:
Coverage - the Good’s coverage (http://scikit-bio.org/docs/latest/generated/generated/skbio.diversity.alpha.goods_coverage.html#skbio.diversity.alpha.goods_coverage)
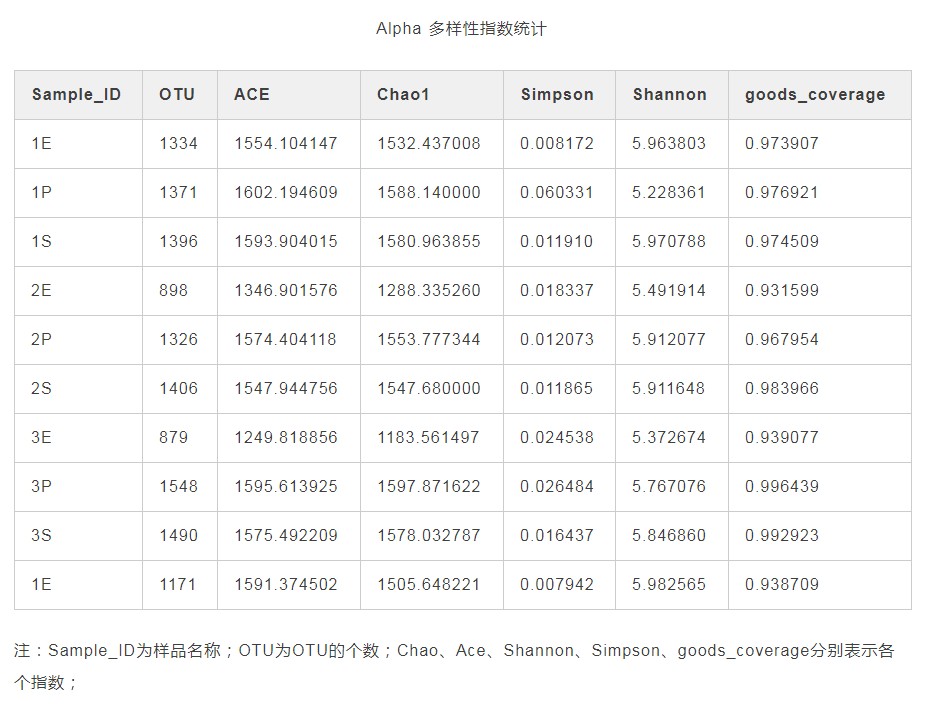 alpha多样性与丰度展示稀释曲线
alpha多样性与丰度展示稀释曲线
微生物多样性分析中需要验证测序数据量是否足以反映样品中的物种多样性,稀释曲线(丰富度曲线)可以用来检验这一指标,并间接反映样品中物种的丰富程度。具体方法为:利用已测得16S rDNA序列中已知的各种OTU的相对比例,来计算抽取n个(n小于测得reads序列总数)reads时出现OTU数量的期望值,然后根据一组n值(一般为一组小于总序列数的等差数列)与其相对应的OTU数量的期望值做出曲线来。当曲线趋于平缓或者达到平台期时也就可以认为测序深度已经基本覆盖到样品中所有的物种;反之,则表示样品中物种多样性较高,还存在较多未被测序检测到的物种。
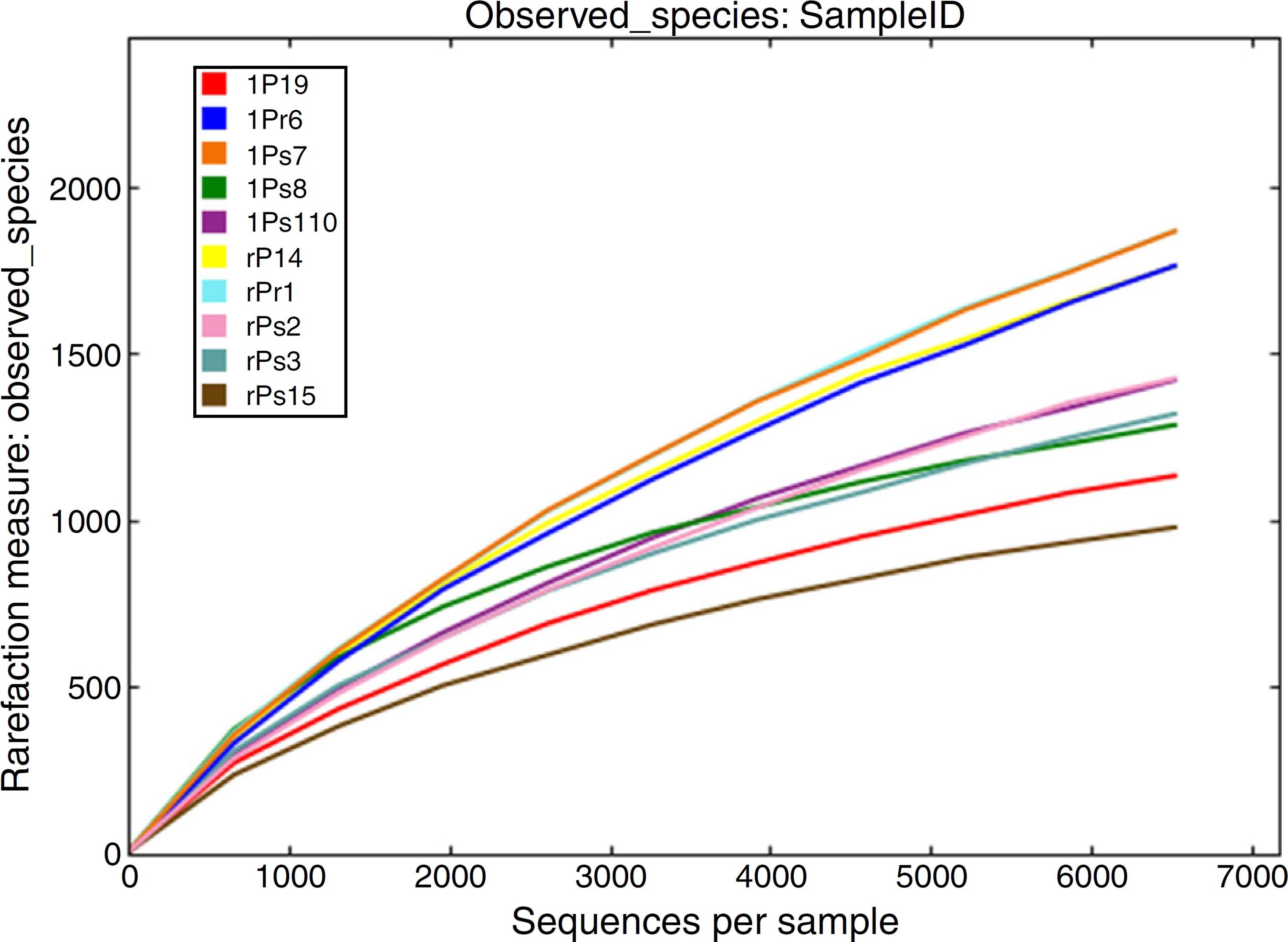 注:横坐标代表随机抽取的序列数量;纵坐标代表观测到的OTU数量。样本曲线的延伸终点的横坐标位置为该样本的测序数量,如果曲线趋于平坦表明测序已趋于饱和,增加测序数据无法再找到更多的OTU;反之表明不饱和,增加数据量可以发现更多OTU。Shannon-Winner曲线
注:横坐标代表随机抽取的序列数量;纵坐标代表观测到的OTU数量。样本曲线的延伸终点的横坐标位置为该样本的测序数量,如果曲线趋于平坦表明测序已趋于饱和,增加测序数据无法再找到更多的OTU;反之表明不饱和,增加数据量可以发现更多OTU。Shannon-Winner曲线
Shannon-Wiener 曲线,是利用shannon指数来进行绘制的,反映样品中微生物多样性的指数,利用各样品的测序量在不同测序深度时的微生物多样性指数构建曲线,以此反映各样本在不同测序数量时的微生物多样性。 当曲线趋向平坦时,说明测序数据量足够大,可以反映样品中绝大多数的微生物物种信息。样本曲线的延伸终点的横坐标位置为该样本的测序数量,如果曲线趋于平坦表明测序已趋于饱和,增加测序数据无法再找到更多的OTU;反之表明不饱和,增加数据量可以发现更多OTU。其中曲线的最高点也就是该样本的Shannon指数,指数越高表明样品的物种多样性越高。
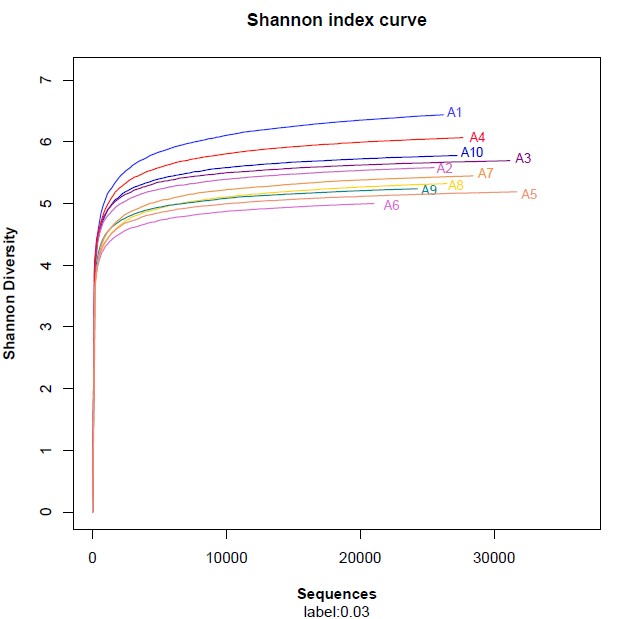 注:与上图一样,横坐标代表随机抽取的序列数量;纵坐标代表的是反映物种多样性的Shannon指数。Rank-Abundance曲线
注:与上图一样,横坐标代表随机抽取的序列数量;纵坐标代表的是反映物种多样性的Shannon指数。Rank-Abundance曲线
Rank-Abundance曲线用于同时解释样品多样性的两个方面,即样品所含物种的丰富程度和均匀程度。物种的丰富程度由曲线在横轴上的长度来反映,曲线越宽,表示物种的组成越丰富;物种组成的均匀程度由曲线的形状来反映,曲线越平坦,表示物种组成的均匀程度越高。
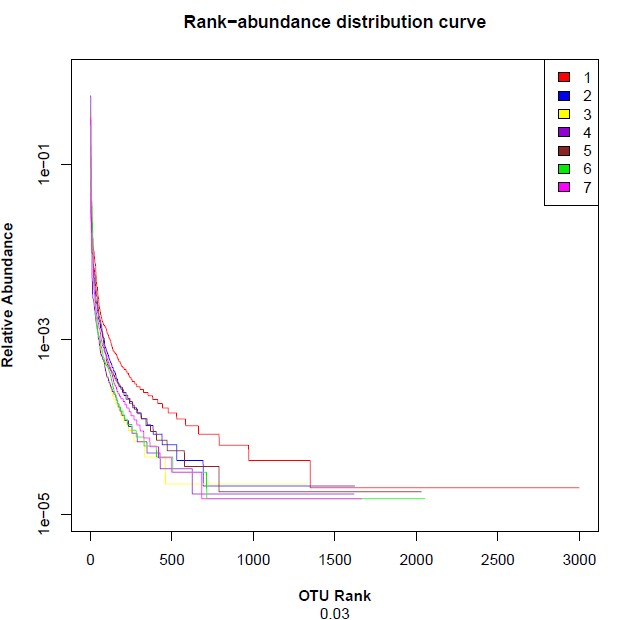 注:横坐标代表物种排序的数量;纵坐标代表观测到的相对丰度。样本曲线的延伸终点的横坐标位置为该样本的物种数量,如果曲线越平滑下降表明样本的物种多样性越高,而曲线快速陡然下降表明样本中的优势菌群所占比例很高,多样性较低。
注:横坐标代表物种排序的数量;纵坐标代表观测到的相对丰度。样本曲线的延伸终点的横坐标位置为该样本的物种数量,如果曲线越平滑下降表明样本的物种多样性越高,而曲线快速陡然下降表明样本中的优势菌群所占比例很高,多样性较低。
这部分内容就讲到这里,后期我们会介绍微生物多样性beta多样性分析,研究微生物的同学请保持关注哦。
课程推荐:微生物扩增子分析课程实操 微生物16S/ITS/18S分析原理及结果解读
更多可观看《微生物多样性分析原理视频课程》
参考文献
[1] Shannon, C.E. (1948a). A mathematical theory of communication. The Bell System Technical Journal 27, 379-423.
[2] Shannon, C.E. (1948b). A mathematical theory of communication. The Bell System Technical Journal 27, 623-656.
[3] Simpson, E.H. (1949). Measurement of Diversity. Nature 163, 688.
[4] Chao, A., and Yang, M.C.K. (1993). Stopping rules and estimation for recapture debugging with unequal failure rates. Biometrika 80, 193-201.
[5] Chao, A. (1984). Nonparametric Estimation of the Number of Classes in a Population. Scandinavian Journal of Statistics 11, 265-270.
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、biolinux搭建生物信息分析环境、linux命令处理生物大数据、perl入门到精通、perl语言高级、R语言画图、R语言快速入门与提高
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、 GEO芯片数据不同平台标准化 、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代fastq测序数据解读、
9.更多课程可点击:组学大讲堂视频课程
- 发表于 2018-04-20 19:31
- 阅读 ( 76365 )
- 分类:宏基因组
