从TCGA数据库中下载指定类型样本的数据
从TCGA数据库中下载指定类型样本的数据
在前面的文章中,我们介绍了如何从TCGA中下载数据,但是那种方法下载的数据中包括了癌症相关各种类型的样本,而有时候我们只想分析其中的一种样本的数据,那该如何下载指定类型的数据呢?
其实可以用TCGAbiolinks 软件包中的GDCquery 函数,指定sample.type 去筛选样本。
比如:需要下载实体瘤组织的样本,那么指定sample.type 为 “Primary solid Tumor”即可:
query <- GDCquery(project = "TCGA-LGG",
legacy = TRUE,
data.category = "Gene expression",
data.type = "Gene expression quantification",
platform = "Illumina HiSeq",
file.type = "results",
experimental.strategy = "RNA-Seq",
sample.type = "Primary solid Tumor")
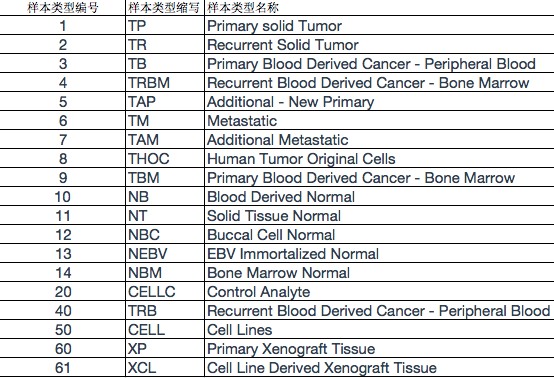
TCGA数据库中主要有如下几种类型的样本:
如果您想学习TCGA数据挖掘,请学习的我TCGA系列课程:
- 发表于 2019-02-15 17:28
- 阅读 ( 4824 )
- 分类:TCGA
你可能感兴趣的文章
0 条评论
请先 登录 后评论
