R语言中实现T检验及可视化
T检验,亦称student t检验(Student's t test),主要用于样本含量较小(例如n < 30),总体标准差σ未知的正态分布。T检验是用t分布理论来推论差异发生的概率,从而比较两个平均数的差异是否显著。在R语言中T检验用的方法为:t.test(),如果数据不符合正态分布,也就是数据当中有较大的离群值时,可选用非参数秩和检验法,如Wilcoxon test,R语言中对应的方法为:wilcox.test()。关于检验方法的选择可参考:差异统计检验如何选择
单样品T检验
例:某鱼塘水的含氧量多年平均值为4.5mg/L,现在该鱼塘设10点采集水样,问该次抽样的水中含氧量与多年平均值是否有显著差异。
#数据
s<-c(4.33,4.62,3.89,4.14,4.78,4.64,4.52,4.55,4.48,4.26)
shapiro.test(s) #如果P>0.05 符合正态分布
t.test(s,mu=4.5) #T检验, 如果 P>0.05 相等
非配对两样本T检验
例:为了了解某一降血压药物的效果,将28名高血压病患者随机等分到实验组和对照组,实验组采用新降压药物,对照组则用标准药物治疗,测得治疗前后舒张压的差值如下。问新药和标准药的疗效是否不同?
high<-c(134,146,106,119,124,161,107,83,113,129,97,123)
low<-c(70,118,101,85,107,132,94)
x<-c(high,low)
group<-c(rep("high",12),rep("low",7))
#正态性检验,wilcox.test()
shapiro.test(high) #如果P>0.05 符合正态分布
shapiro.test(low) #如果P>0.05 符合正态分布
#方差齐性检验:如果P>0.05 方差齐
bartlett.test(x~group)
#方法二:car包中leveneTest 检验,spss统计软件默认的检验方法
leveneTest(x~group)
#T检验, 如果 P<0.05 存在差异
t.test(high,low,paired=F,var.equal=T) #如果方差不齐,可更改:var.equal=F,
#或者:
t.test(x~group,paired=F,var.equal=T)
配对两样本T检验
例:为了解DSCT冠状动脉造影和超声心动图检查两种方法测定心脏病患者左室舒张末容积的差别,某医院收集心脏病患者12例,同时分别用两种检测方法测得其大小如下,问两种检测方法的检测结果是否不同?
ds<-c(82.5,85.2,87.6,89.9,89.4,90.1,87.8,87.0,88.5,92.4)
cs<-c(91.7,94.2,93.3,97.0,96.4,91.5,97.2,96.2,98.5,95.8)
#方差齐性检验,car包中leveneTest
leveneTest(ds,cs)
#作差,正态性检验
#差值正态性检验,差值符合正态分布(P>0.05)
d<-ds-cs
shapiro.test(d)
#配对T检验
t.test(ds,cs,paired=T,alternative="two.sided",conf.level=0.95)
统计检验及绘图-ggpubr
ggpubr包既可以做检验,有可以对统计结果进行整理绘图,输出结果比t检验更加友好。
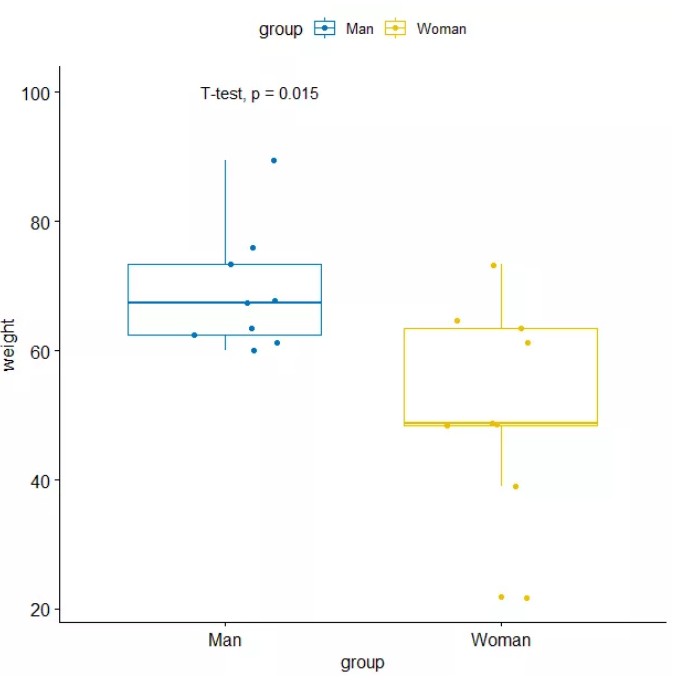
例:男女之间的体重是否存在差异,分别随机选取9个男女测量体重,数据:
women_weight <- c(38.9, 61.2, 73.3, 21.8, 63.4, 64.6, 48.4, 48.8, 48.5)
men_weight <- c(67.8, 60, 63.4, 76, 89.4, 73.3, 67.3, 61.3, 62.4)
mydata <- data.frame(
group = rep(c("Woman", "Man"), each = 9),
weight = c(women_weight, men_weight)
)
#统计检验
com1 <- compare_means( weight~ group , data = mydata, method = "t.test")
#结果P=0.015,小于0.05,具有显著差异:
#.y. group1 group2 p p.adj p.format p.signif method
# weight Man Woman 0.0154 0.015 0.015 * T-test
绘图显示
install.packages("ggpubr")
library(ggpubr)
p <- ggboxplot(mydata, x="group", y = "weight", color = "group", palette = "jco", add = "jitter", short.panel.labs = FALSE)
# 添加p值
p + stat_compare_means(method = "t.test",label.y=100)
# 显示p值但不显示方法
p + stat_compare_means(aes(label = ..p.format..),method = "t.test",label.x = 1.5)
# 只显示显著性水平
p + stat_compare_means(aes(label = ..p.signif..),method = "t.test",label.x = 1.5)
结果图如下:
推荐学习R语言获得更多生物信息分析技能:R语言画图、R语言快速入门与提高
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、biolinux搭建生物信息分析环境、linux命令处理生物大数据、perl入门到精通、perl语言高级、R语言画图、R语言快速入门与提高
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、 GEO芯片数据不同平台标准化 、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
- 发表于 2019-03-08 10:51
- 阅读 ( 16709 )
- 分类:R
你可能感兴趣的文章
- corrplot包绘制相关性图,简单又好看! 92 浏览
- 已知集合交集数量绘制韦恩图 382 浏览
- 绘制Upset 图 1672 浏览
- R公式中如何加入变量 755 浏览
- R语言基础入门—数据结构(2) 774 浏览
- R语言基础入门—数据结构(1) 723 浏览
