转录组测序分析水稻旱作的重要基因
随着气候的改变、水资源短缺以及人口的增长,进行作物品种改良,使其适应环境变化,对可持续农业具有重要的作用。水稻是重要的粮食作物,它包括了传统的水作水稻(水稻)和旱作水稻(陆稻)等。二代测序数据如何分析水稻旱作适应的分子机制呢?下面就给大家讲解一篇相关文献,帮助大家了解!
材料方法
材料:水稻(Oryza sativa L.) 两个品种,CR Dhan 202 (AAC,陆稻),BPT 5204 (NAC,水稻)。
处理:各品种分别进行旱作(土壤湿润,透气排水)和水作(水淹高度2.5 cm)培养,温室条件(28/20 °C day/night)。
取样:萌发后(60-70 days)进入圆锥花序期,每种处理分别采取嫩芽和根组织,各三个生物学重复。
测序:生物学重复混样提取RNA进行测序。
测序样品对应:BPT 5204 Aerobic Shoot,BAS;CR Dhan 202 Aerobic Shoot,CAS;BPT 5204 Anaerobic Shoot,BFS;CR Dhan 202 Anaerobic Shoot,CFS;BPT 5204 Anaerobic Root, BFR;BPT 5204 Aerobic Root ,BAR;CR Dhan 202 Aerobic Root, CAR;CR Dhan 202 Anaerobic Root ,CFR。
测序数据
八个测序文库的测序数据见下表,完成数据预处理并将clean reads和参考基因组比对,共1.38 billion的clean reasd比对上参考基因组,之后计算转录本的表达量FPKM,以便进一步分析。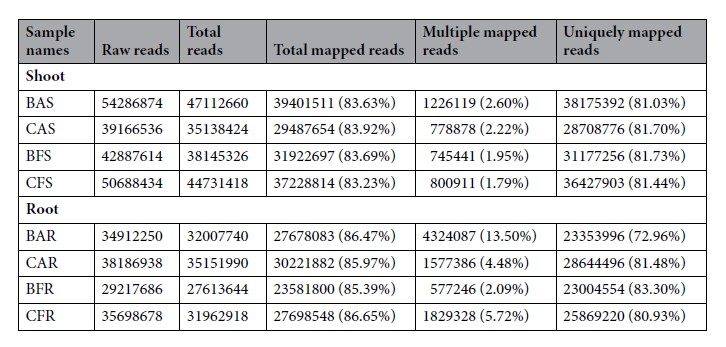
差异表达分析
对ACC 和NAC不同的处理方式的样本间进行差异分析,共四组差异比较(BAS vs. CAS、BAR vs. CAR、BFS vs. CFS、BFR vs. CFR),差异分析的阈值为FDR<0.005 & |log2FC|≥1.5。
结果发现:旱作条件下,根组织的差异转录本数量(DETs,BAR vs. CAR)高于水作条件下DETs(BFR vs. CFR),嫩芽组织相反,旱作条件下的DETs(BAS vs. CAS)低于水作条件下的DETs (BFS vs. CFS),具体见下图,实验基于根部差异分析共筛选出了237个DETs,而基于嫩芽组织共获得了188个DETs。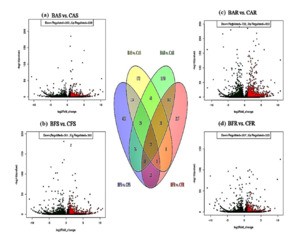
功能注释与富集
针对DETs进行了功能注释和富集分析,部分GO注释结果见下图,富集分析发现,涉及缺水响应 (GO: 0009414), 冷 (GO: 0009409) 以及胁迫相应 (GO: 0006950),核酸结合转录因子 (GO: 0001071) 以及氧化还原酶活性 (GO: 0016491),细胞外区域(GO:0005576)和胞质膜结合囊泡(GO:0016023)出现了明显富集。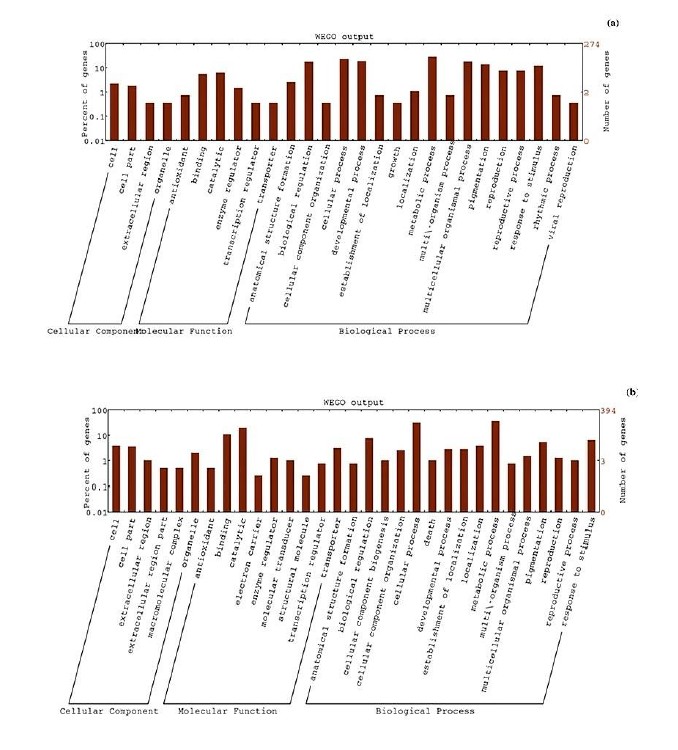
KEGG富集分析显示,旱作条件下,根部组织的DETs明显富集于代谢以及二级代谢合成等相关途径,嫩芽组织则主要涉及脂肪酸水解、乙醛酸盐和二羧酸盐代谢等。
此外,利用MapMan对这些DETs进行代谢通路分析,重点针对旱作条件下的DETs在代谢和二级代谢合成进行了分析,部分结果见下图。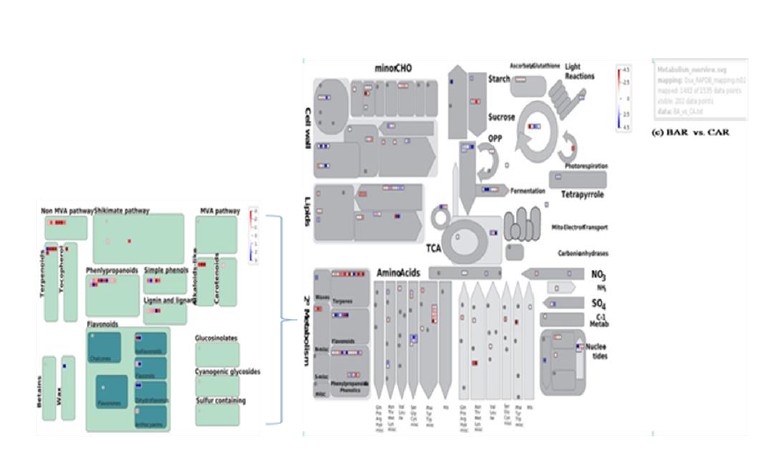
转录因子、转运体、根性状相关基因
针对DETs分析其是否属于转录因子(TFs)、转运体、根性状相关基因等。DETs转录因子分析结果显示:嫩芽组织,9个TFs(DETs)仅旱作条件下出现表达(较之水作条件),其中7个TFs在AAC中较之NAC显著性上调(BAS vs. CAS),这其中的5个(MADS4, MADS5, MADS6, MADS7 and putative WRKY transcription factor 6)仅在AAC中。根组织进行了类似分析,也有5个TFs( MADS15, MADS26, Dehydration-responsive element-binding (DREB) family protein, DREB1F and DREB1C)仅在ACC中(见下图)。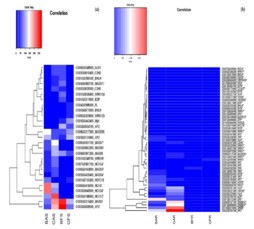
而转运体的分析方法类似转录因子,重点分析了离子转运体,结果见下图。此外还分析的根性状相关的基因,并基于以上的分析筛选的基因进行后期的实时荧光定量PCR(qRT-PCR)实验。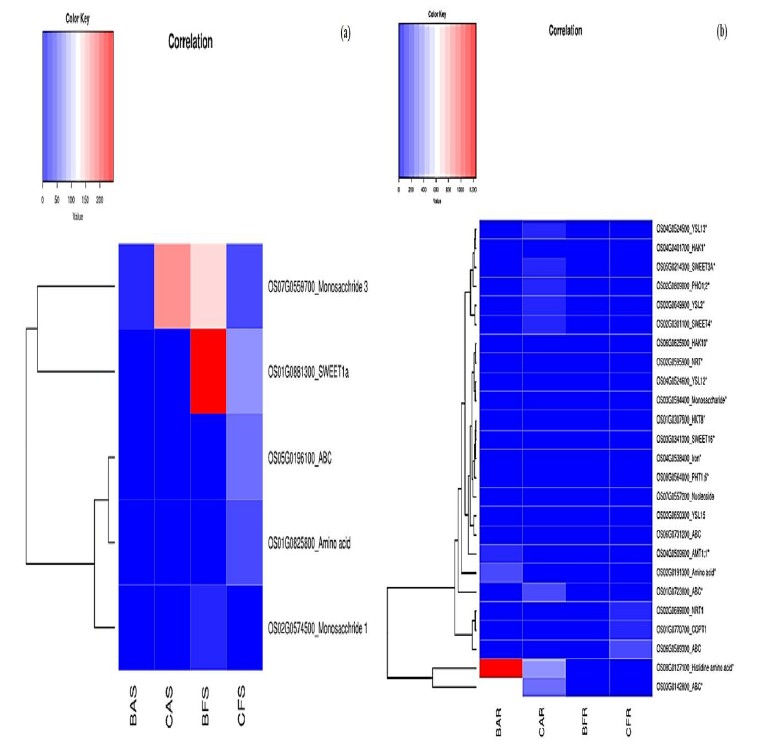
可变剪切
基于可变剪切对于基因表达调控的影响,实验重点分析了旱作条件对可变剪切是否产生影响,分别分析实验样本中的五种可变剪切事件,结果显示,旱作条件下,无论是根组织还是嫩芽组织中的可变剪切事件都明显高于水作条件。
其他
实验还涉及了其他的分析,譬如,为鉴定是否存在基因属于已报导缺水条件相关QTLs等,选择ACC中上调DETs于QTARO数据库进行in-silico mapping,结果发现MADS4基因比对上了两个根性状相关的QTLs,还涉及其他的一些基因和相关的QTLs等等。
此外,为验证测序和分析结果,基于此前的分析,针对重要的基因进行了qRT-PCR,并与测序计算的表达了量进行了拟合分析,结果见下图(a:BAS vs. CAS ,b:BAR vs. CAR):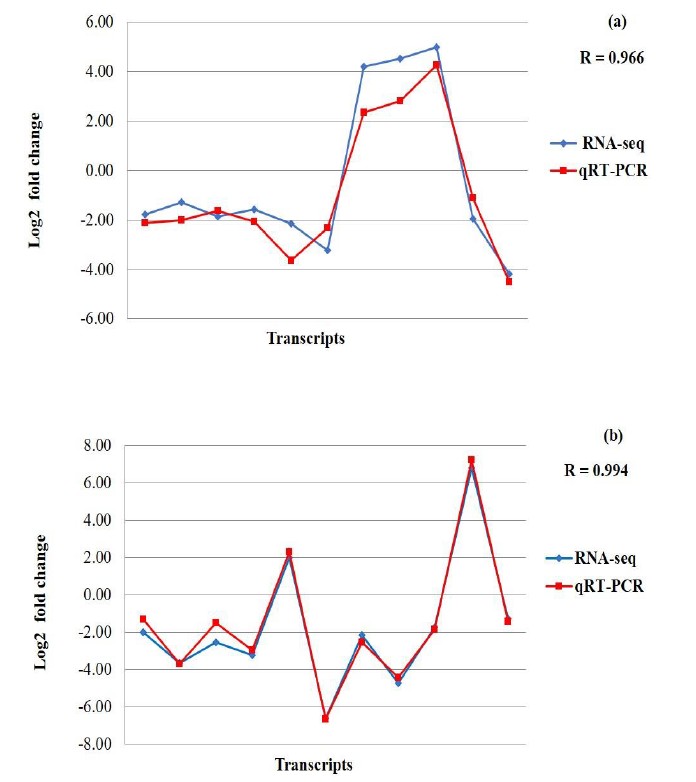
结论
作者针对陆稻和水稻品种进行不同栽培处理,分析不同品种间的差异转录本,并针对这些差异的转录本挖掘和根系性状、缺水相关的基因,重点探索了这些基因可能涉及到的功能,从而阐述水稻旱作的分子机制。
参考文献:
Amol S. Phule, Kalyani M. Barbadikar, Sheshu Madhav Maganti, P. Seguttuvel, D. Subrahmanyam, M. B. B. Prasad Babu & Polumetla A. Kumar RNA-seq reveals the involvement of key genes for aerobic adaptation in rice. Scientific Reports volume 9, Article number: 5235 (2019)
推荐:
二代测序转录组数据自主分,转录组标准分析后的数据挖掘、转录组文献解读
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、perl入门到精通、perl语言高级、R语言入门、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、GEO芯片数据标准化、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读
- 发表于 2019-05-16 09:38
- 阅读 ( 4993 )
- 分类:转录组
你可能感兴趣的文章
- Rna-seq 去除批次效应ComBat-seq 2463 浏览
- mapman非模式植物基因注释时遇到的问题 3142 浏览
- Mapman对表达数据进行venn绘制 4446 浏览
- 投稿时-SCI期刊文章中图片大小如何设置 59729 浏览
- Mapman对表达数据进行聚类分析 6696 浏览
相关问题
- RNA-seq课程中的初始数据来源于哪篇文章 0 回答
- mapman注册账号下载mapping文件,验证码图片加载不出来 0 回答
- mapman安装找不到路径,确定java配置到环境中。 0 回答
- mapman非模式植物基因注释时遇到的问题 1 回答
- 转录组差异基因分析若干问题 0 回答
- 老师您好,我在hisat2建立索引时报错,向您求助 1 回答
