鉴定domain同时分析蛋白互作
SMART(http://smart.embl.de/)是我们常用来进行domain搜索和蛋白质注释的数据库,它集成了很多蛋白结构预测和功能分析的工具,比如可以预测蛋白的一些二级结构:跨膜区(Transmembrane segments)、复合螺旋区(coiled coil regions)、信号肽(Signal peptides)、蛋白结构域(PFAM domains)等。而此前小编刚好给大家介绍过如何进行单序列或者批量domain鉴定(点击了解一下)。其实通过单序列domian的预测也可以获取该蛋白和其他蛋白的互作关系,进而提取出其对应的蛋白质互作网络。
蛋白互作关系获取
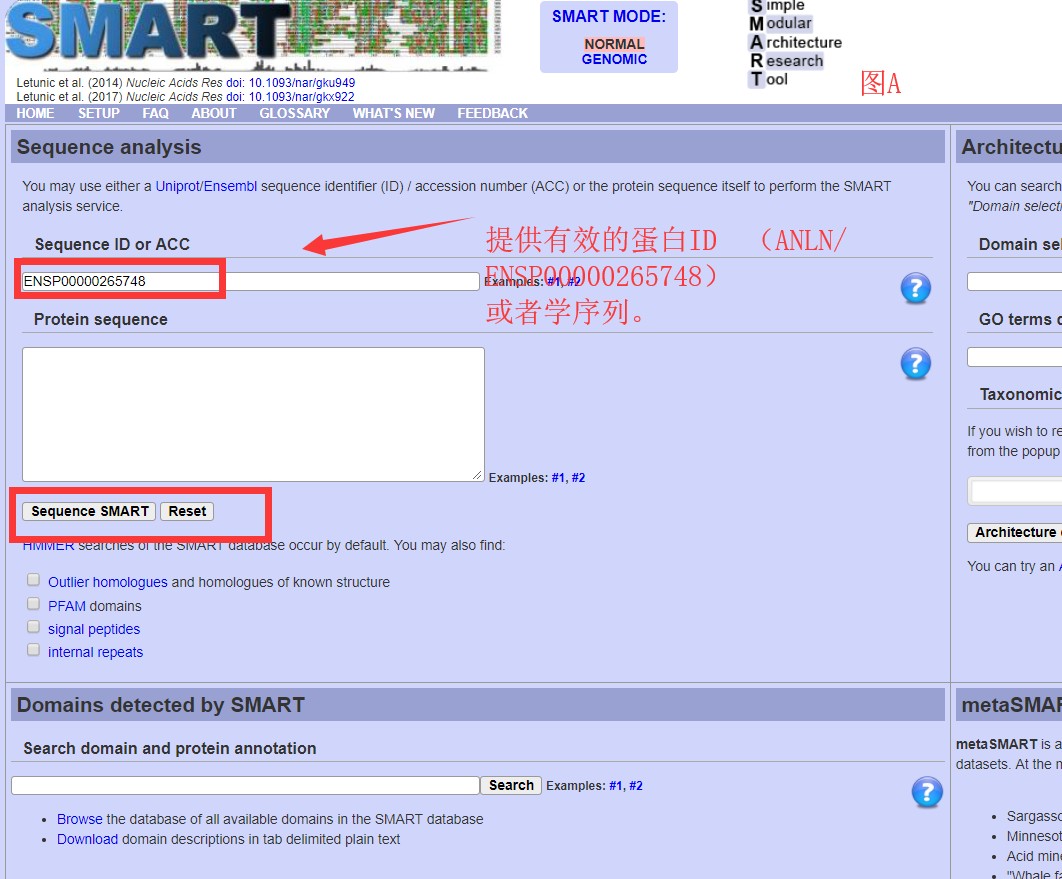
SMART数据库进行单序列domain鉴定过程,可以提交蛋白的相关ID或者氨基酸序列(见下图A),而提交之后跳转页面显示出对应的结构域信息,同时还展示了互作关系Interactions。其蛋白互作信息是基于STRING数据库获取,通过输入蛋白,能够判断哪些基因编码的蛋白与该蛋白存在紧密联系。
譬如:以人的ANLN基因编码的蛋白ENSP00000265748(ensembl 蛋白ID)为例输入,获得的互作关系结果见下图B。
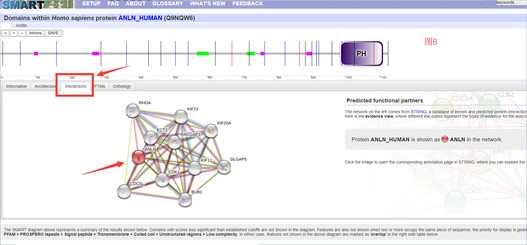
ANLN是一个与癌症相关的基因(可戳此了解),输入对应的ID,在domian分析界面选择Interactions将直接显示STRING获取的蛋白互作结果。基于此,单击此区域,可以直接跳转到STRING数据库(见下图),进而了解到完整的互作信息和相关参数设置,并可以将对应的互作网络进行下载。
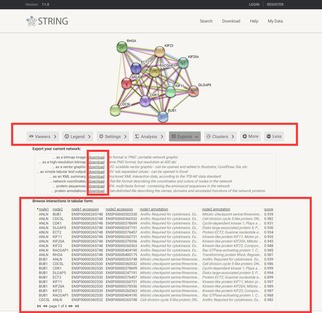
是不是发现你在进行蛋白质结构预测的同时也能分析它的互作关系了!
推荐:
二代测序转录组数据自主分,转录组标准分析后的数据挖掘、转录组文献解读
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、perl入门到精通、perl语言高级、R语言入门、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、GEO芯片数据标准化、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读
- 发表于 2019-06-13 17:57
- 阅读 ( 8492 )
- 分类:软件工具
你可能感兴趣的文章
- ppi_network.r 蛋白互作网络分析 3692 浏览
- STRING蛋白互作数据库在线分析基因互作网络(PPI) 30384 浏览
- 如何批量分析蛋白序列domain-SMART数据库 48406 浏览
- 一个预测基因保守结构域网站 16778 浏览
