技术贴-agriGO简单好用的GO富集网站来一个!
agriGO(http://bioinfo.cau.edu.cn/agriGO/index.php)是一个主要针对植物类以及部分动物的GO分析网站,此外,网站还涉及物种相关的数据可供用户下载。而用户选择的分析包括Singular Enrichment Analysis (SEA)、 PAGE (Parametric Analysis of Gene set Enrichment)、 BLAST4ID (Transfer IDs by BLAST) 、SEACOMPARE (Cross comparison of SEA)等。这里我们就来教大家使用它的主要功能GO富集分析SEA。
富集分析
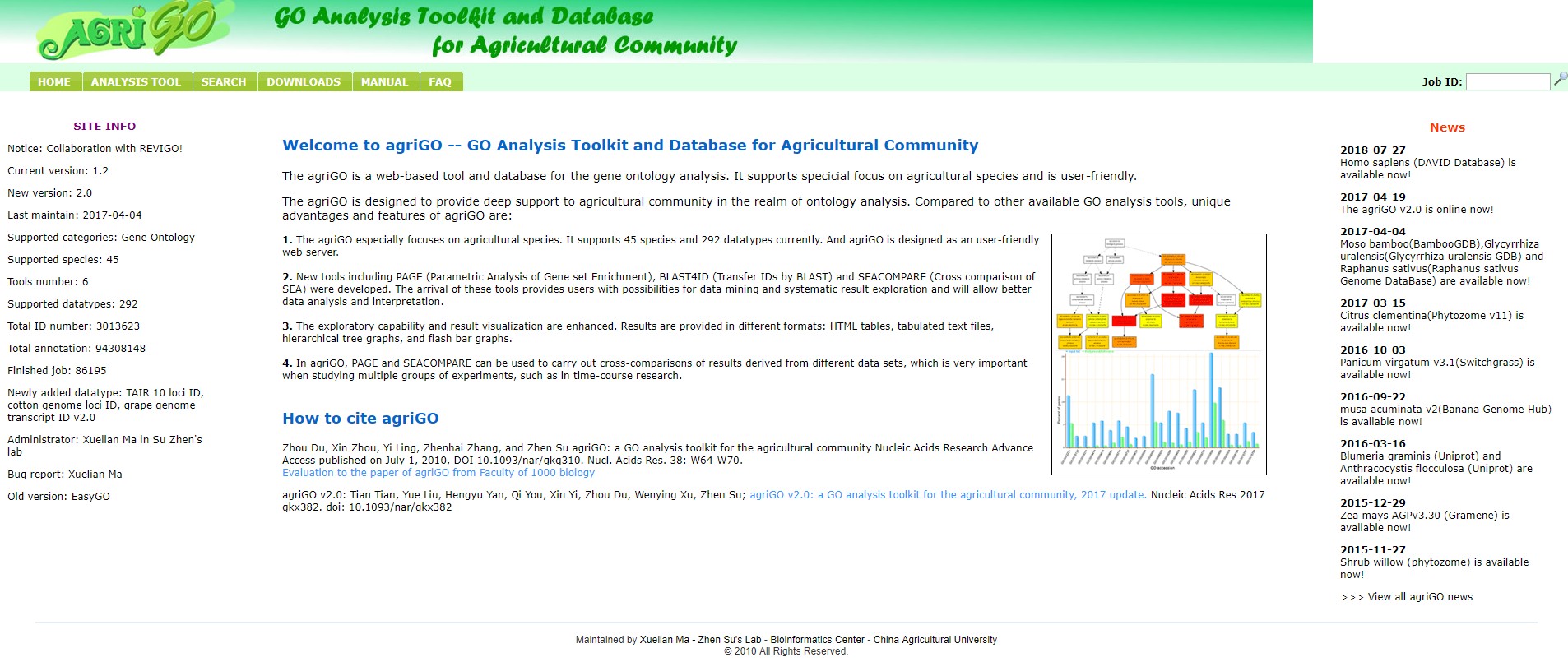
进行分析的操作非常简单友好,点击上方“Analysis Tool”进入分析界面,选择SEA,此时会根据选定的分析内容显示需要设置的分析选项,SEA的分析界面如下图所示。
各种分析具体概念解释和内容都将在页面的右侧显示,例如SEA分析就是传统的富集分析过程。而选定分析之后要确定分析的物种,在2.Select the species中进行物种设定,具体涵盖网站涉及的45个,同时在对应的输入框中输入正确的基因ID。不同的物种对应的ID类型有所差异,具体可接受的ID类型在网页右侧有明确的的介绍,譬如下图选择拟南芥之后输入的是TAIR locus ID。之后于3.Select reference中设定富集的backgrounds。
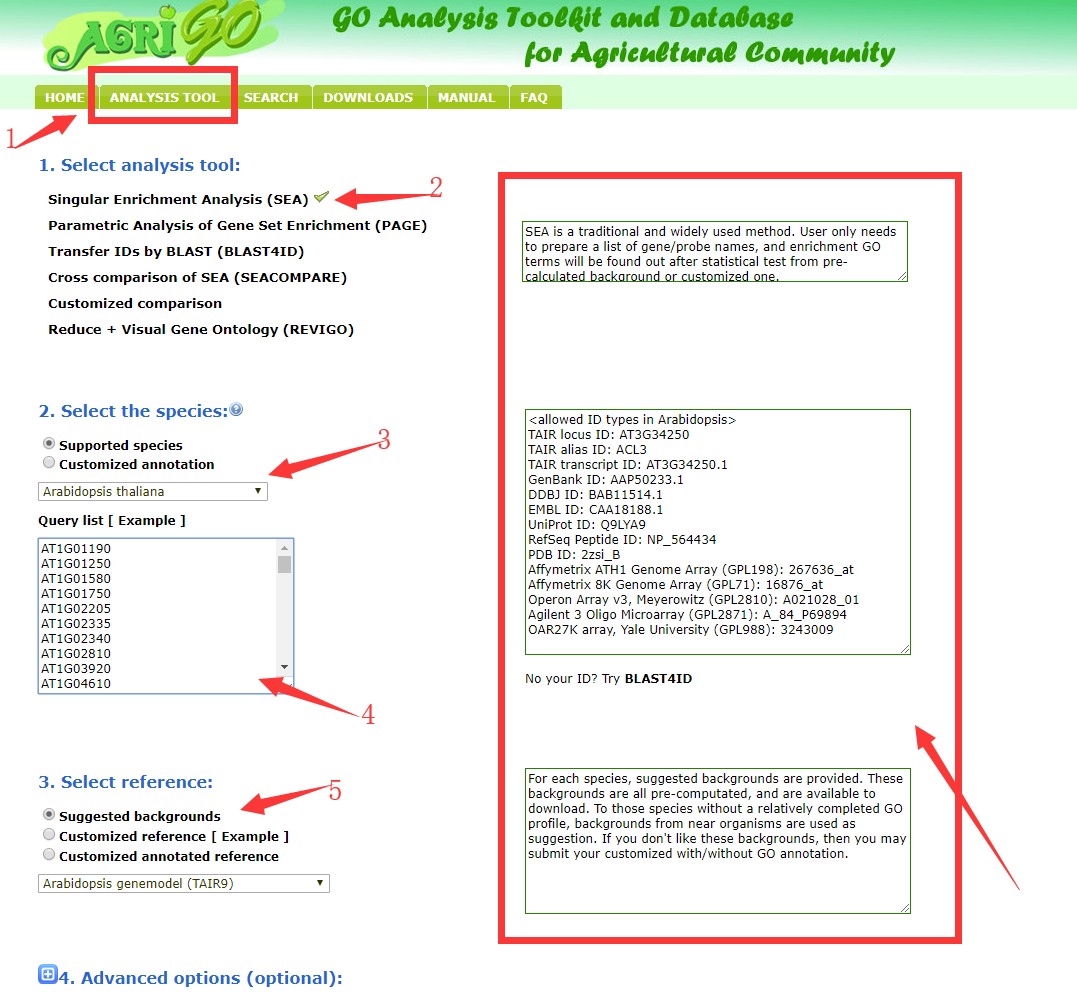
富集分析还涉及4.Advanced options(optional)可选设置,该设置并没有直接展示,鼠标左键单击可展示全部内容,主要涉及分析过程中的相关参数和统计方法、阈值的设定(见下调)。此处演示按照默认参数提交分析,点击Submit即可。
分析结果
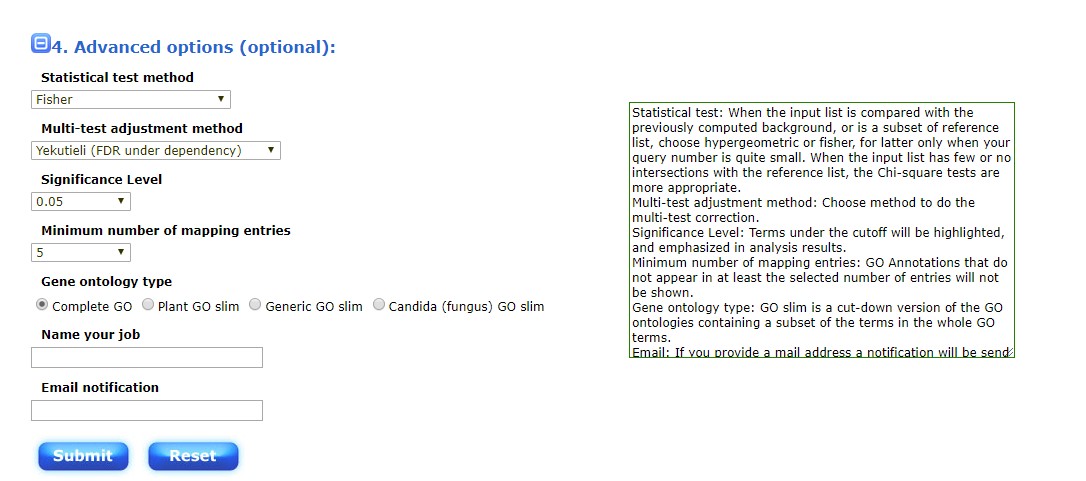
根据设定完成分析之后,跳转到结果页面(如下图所示),第一部分是对数据和分析的概括,之后是各部分详细分析结果,而每一部分都可选定项目进行绘图。
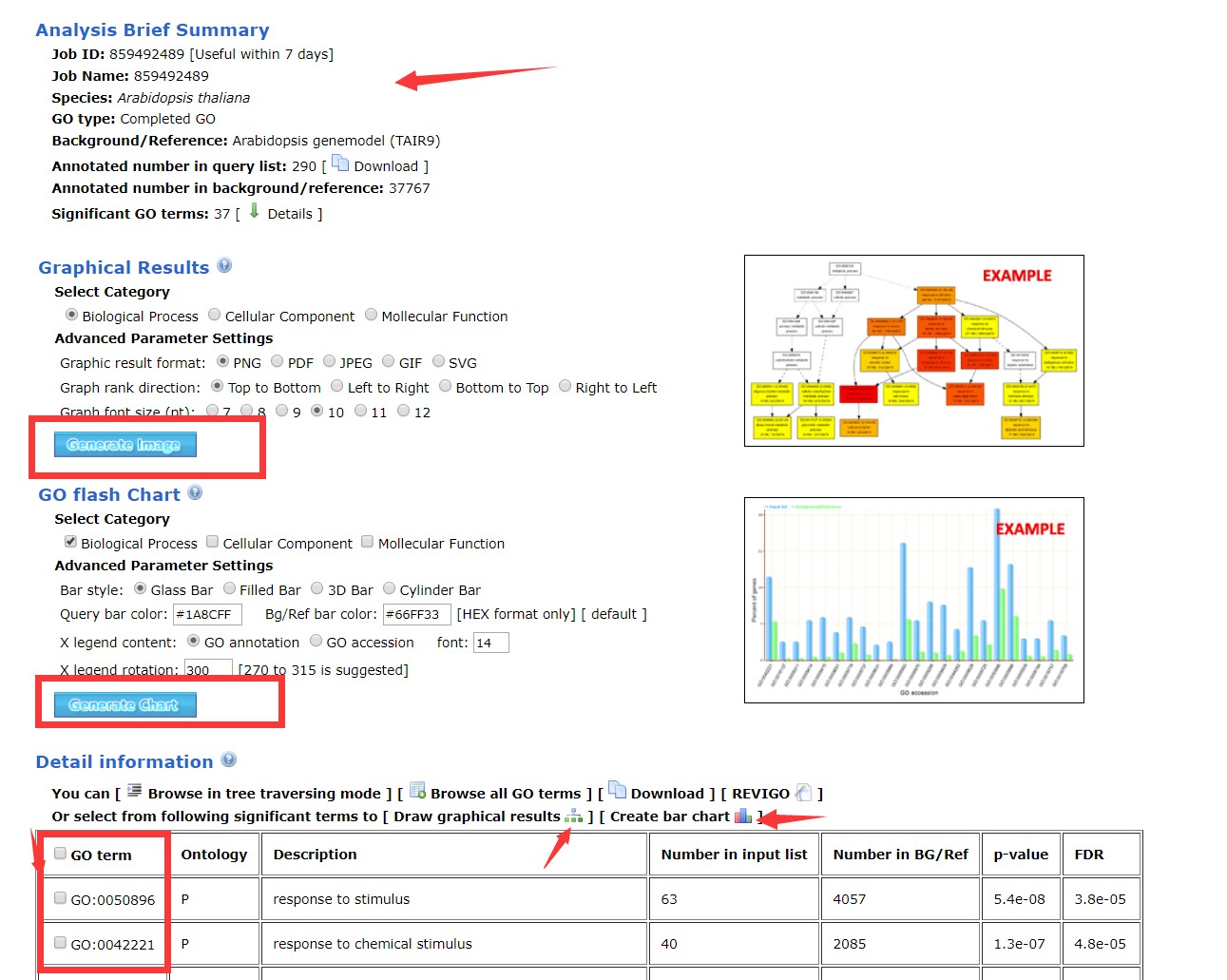
例如,针对第二部分,可以选择Mollecular Function,设定图片类型PDF、绘图方向、大小后点击Generate Image 即可进入图片界面。选择不同的图片类型,出现的界面可能不一致,有的会直接显示图片,而PDF界面会要求进行PDF下载,打开对应的PDF可以得到完整的GO富集的有向无环图(见下图)。
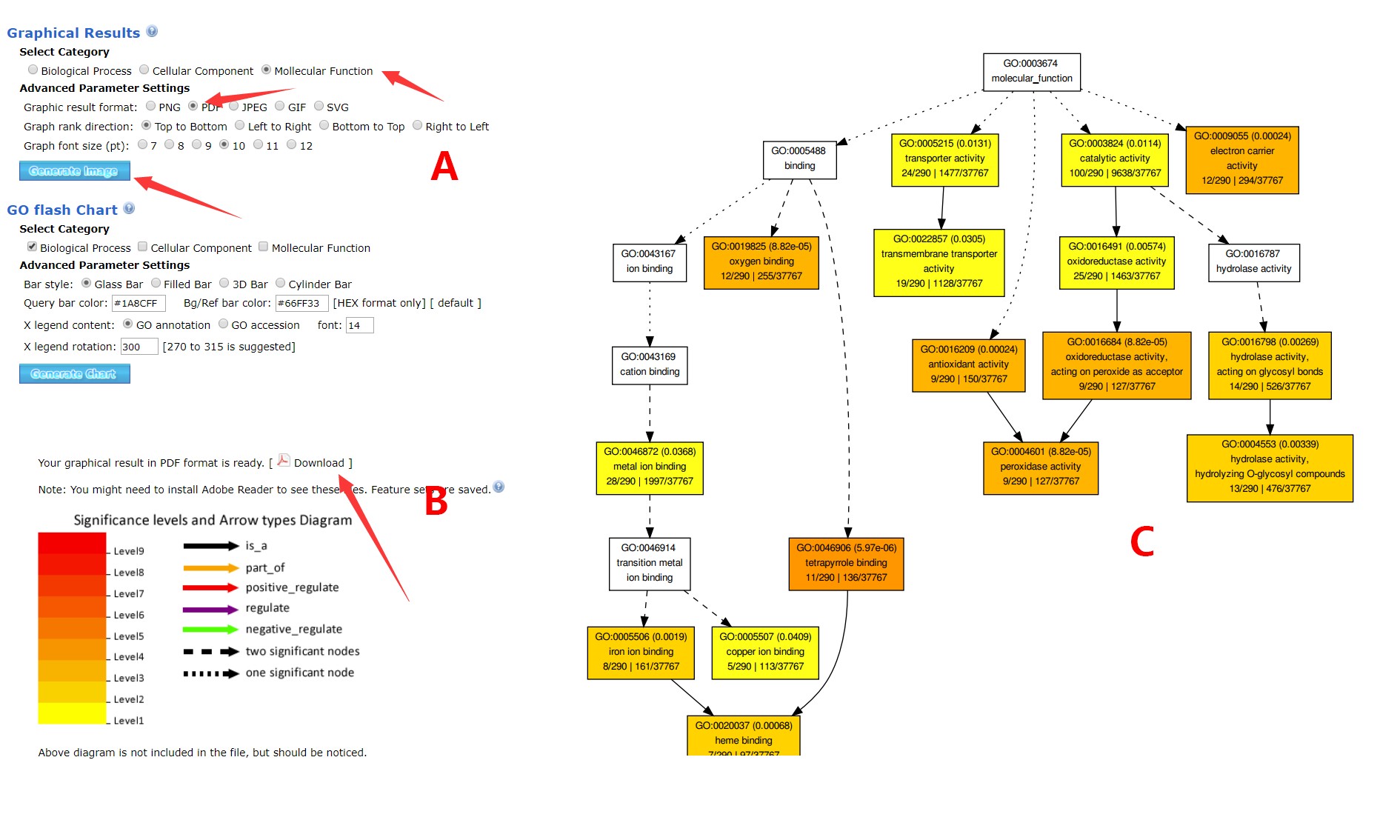
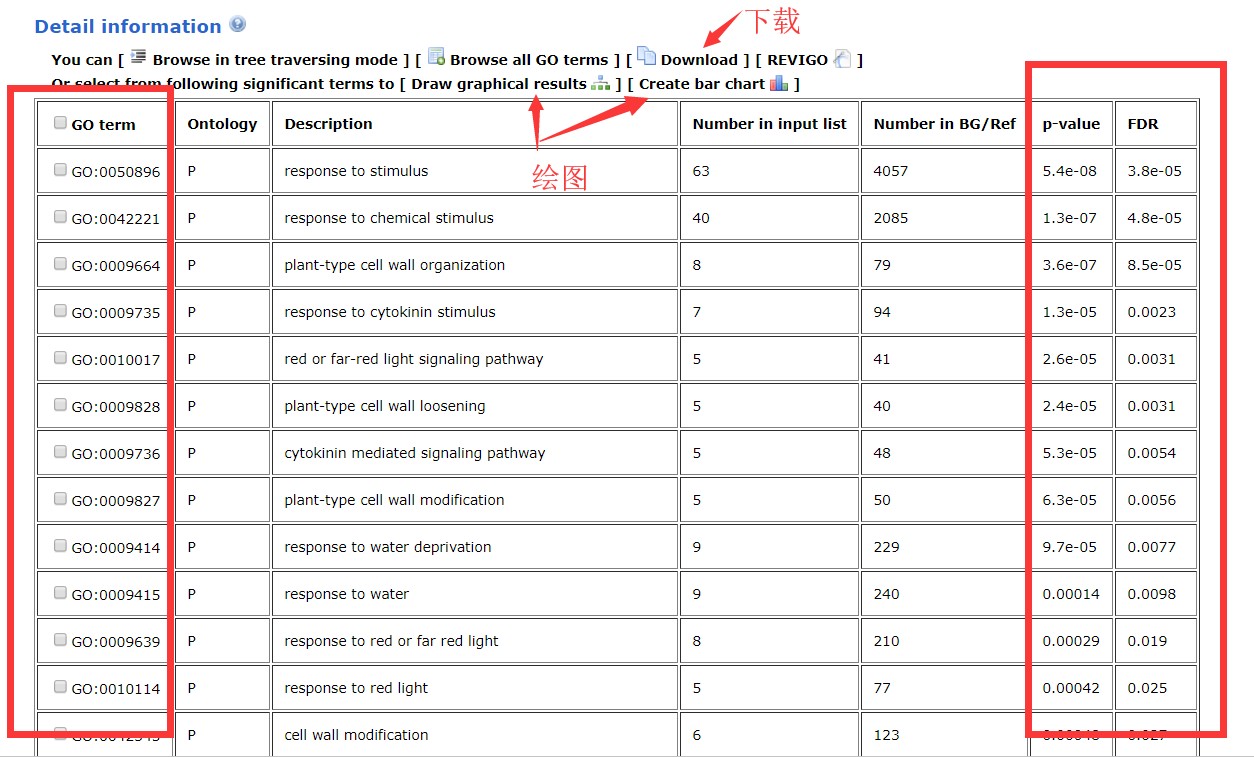
第三部分统计各类GO注释结果柱状图,操作和第二部分类似。至于第三部分的内容是关于富集分析的详细结果列表,基于此表格可以获得对应的GO term显著性大小,同时可以选中具体的几个GO term绘制图片并下载等等。
是不是确实简单又好用?赶快动手试试吧!
推荐:
二代测序转录组数据自主分,转录组标准分析后的数据挖掘、转录组文献解读
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、perl入门到精通、perl语言高级、R语言入门、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、GEO芯片数据标准化、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读
- 发表于 2019-06-28 14:47
- 阅读 ( 20815 )
- 分类:软件工具
你可能感兴趣的文章
- GO富集分析统计表中差异基因数量不一致 1255 浏览
- 一文教你使用Excel画GO富集分析图 15455 浏览
相关问题
- 我现在的数据格式,怎么进行GO和KEGG富集分析呢? 1 回答
- GEO数据挖掘中遇见的问题 1 回答
- GO层级整理 1 回答
