混池(BSA)定位必看的10个知识点!
作为经典的基因定位方法,BulkedSegregant Analysis(BSA,集群分离分析法)具有适用范围广,群体要求低,实验成本低的优势,能够有效对质量性状或数量性状的主效基因进行定位。
但很多初次接触的老师仍对BSA的取样、提取、测序等方面有不少疑问,在此我们总结了10个设计BSA时最常见的问题,希望对大家有所帮助。
Q1:取材时对亲本有什么要求?
A1:亲本要选用尽量纯的个体,可通过自交进行纯化,两个亲本要在目标性状上有显著差异,但其他性状尽量保持一致,以降低后期定位分析的干扰。
Q2:对子代群体有什么要求?
A2:理论上来说,只要目标性状不同的亲本杂交后代产生了性状分离都可以拿来做BSA,但是比较常用的群体有:F2、回交群体、重组自交系等。
如果是质量性状,子代的显隐性个体比值可能有1:1、3:1等情况;数量性状的话,子代性状应当符合正态分布为佳,如果严重偏离,需要考虑是否有隐性致死基因产生了作用。
Q3:没有亲本是否可以做BSA?
A3:可以,但是不建议。目前主流的定位算法有ED法和snp-index法,其中ED法可以在没有亲本的情况下进行,但这样定位效果肯定不如有双亲数据的实验。建议重新杂交构建群体,保存好亲本DNA,以备后用。
Q4:子代取多少样品才够呢?
A4:子代的取样应当符合以下原则:定位质量性状的,应该取尽可能多的隐性个体,最低不能低于20个,一般在30-50之间,然后取对应数量的显性个体;定位数量性状的,一般是选择最极端的各5%-10%性状的个体,还可以将子代的性状数据做个类似如下的直方图:
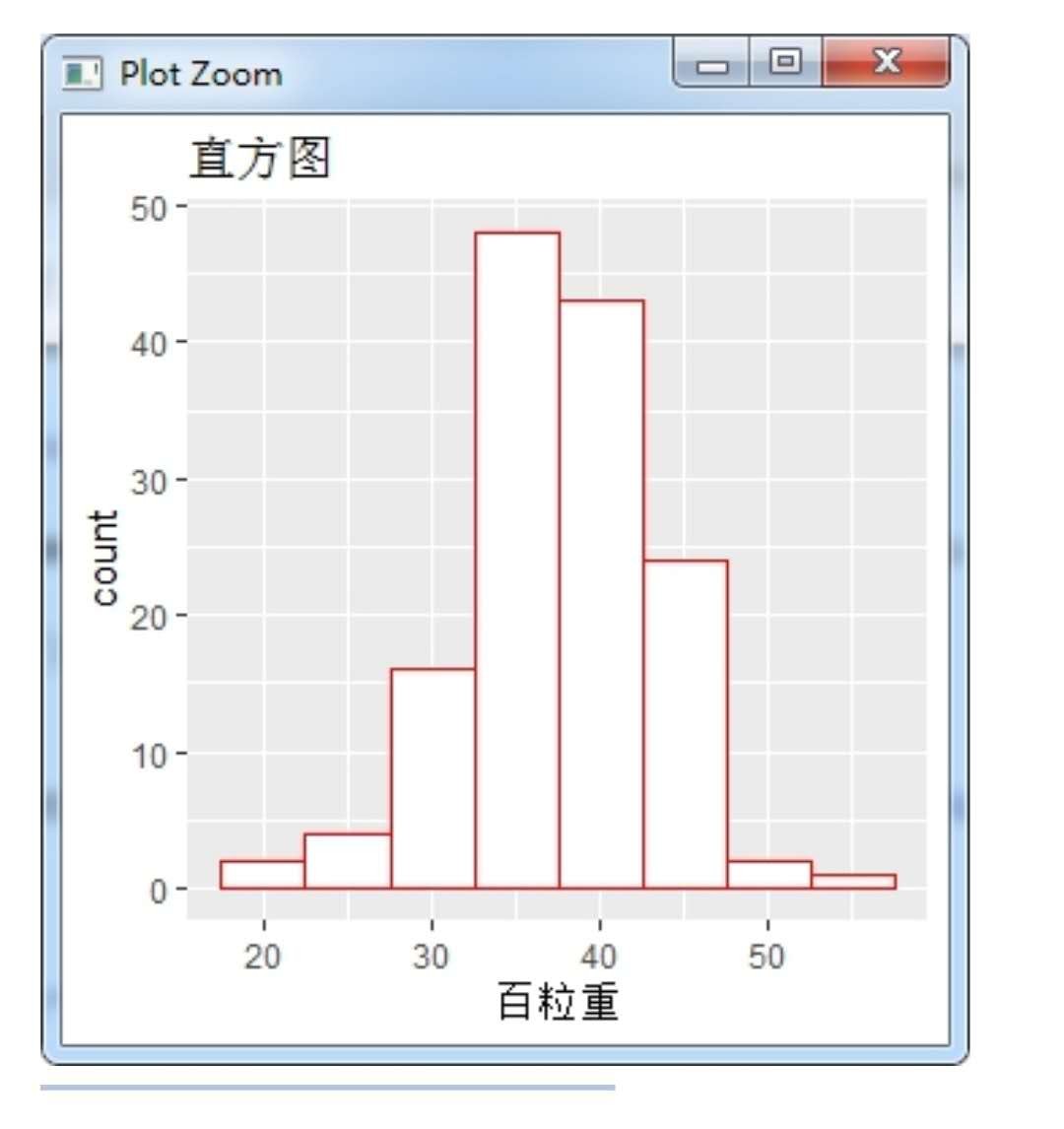
然后结合直方图的信息,共同来选择样品,如上图所示,最左侧的两列和最右侧的两列适合取样。
Q5:子代群体规模不够,应该优先取极端还是优先取数量?
A5:很多物种的子代群体很难做到200个以上,就会出现极端个体不足20的情况,这时我们建议宁可少取些样品,也不要把性状居中的样品选择进去,只会对后期分析产生干扰。当然每一个池只有十来个样品的实验,是否能够得到满意的结果,是存在一定风险的。
Q6:提取子代DNA时应当先混后提,还是先提后混?
A6:理想状态是将每个子代样品都单独提取DNA,然后根据DNA的浓度,等量均匀混合成一份DNA,这样各个子代的DNA信息量是等量的。但由于目前测序项目成交价较低,一般公司的做法是让客户从每个样品上取等量的组织混合,然后提取作为混池DNA样品。
等组织样混合提取效果不如单样品提取后混合,但是其影响完全是可以接受的。先混合再提取虽然会导致来自每个样品的DNA并不等量,但取样足够理想的话,这些样品在目标位点的基因型应该是一致的,主要影响的是背景噪音的组成,这样并不影响我们后期数据分析时找到目标位点。
Q7:做重测序对DNA的质量要求是怎么样的?
A7:虽然每个公司的具体要求略有不同,但重测序对DNA的质量要求并不太苛刻。需要您自己做的是跑一下琼脂糖胶,观察主带是否清晰,是否无蛋白污染,另外用分光光度计检测下浓度和总量(建议浓度不低于20ng/ml,总量不低于2微克)。
Q8:重测序时对亲本和子代的测序深度要求是多少?
A8:为保证SNP、InDel标记的准确性,测序应当保证一定的深度,亲本建议不低于20×,混池应当结合取样数量来定,平均每个样品不低于1×,比如子代是30+30,那么每个子代混池的测序深度就不能低于30×,经费允许的话,可以再适当加深。
Q9:定位效果不理想可能是什么原因导致的?
A9:导致定位不理想的因素很多,主要有以下几点:
① 亲本间遗传背景差异大,除了目标性状外,还有很多其他差异,这样对分析产生的干扰很大,难以定位;
② 性状统计复杂,目标性状可能是由多个简单性状构成,可以拆分性状,重新定位,另外数量性状本身也定位难度较高,有一定不可控性;
③ 测序数据有污染,可以通过抽取部分测序数据在nr库里做blast比对,检查比对结果;
④ 分析方法不适用,可以分别用ED法和snp-index法进行定位,比较定位结果。
Q10:定位区间有点大,怎么进一步往下做?
A10:为了进一步缩小定位区间,可以在BSA定位区间内找些SSR、SNP或者InDel标记,进行局部作图,可以有效的缩小定位区间。
今天的知识点就讲到这里,想学习生物信息学的老师、同学赶紧点击这里进入组学大讲堂网课首页吧!~

- 发表于 2019-07-04 12:40
- 阅读 ( 29357 )
- 分类:基因组学
你可能感兴趣的文章
- BSA分析中MutMap 分析:95% 99%阈值置信区间是如何计算的 5217 浏览
- BSA分析中QTL-seq 分析:95% 99%阈值置信区间是如何计算的 6211 浏览
- BSA分析之MutMap分析原理详解! 10157 浏览
- BSA分析算法中的ED算法和SNP-index有什么区别? 21502 浏览
- MutMap+和mutmap分析差异(BSA) 15844 浏览
相关问题
- 请问如何合并VCF文件中同一样本的生物学重复信息 1 回答
- 老师您好,我用mutmap的方法分析了f2群体,有三个峰,有个一个峰在99阈值之上,但是做基因分分型时 要么是突变体基因型要么是野生型基因型,而且区间前半段设计不出引物 0 回答
- 基因变异的VCF文件除了染色体部分外的其他变异数据如何过滤? 1 回答
- 老师,我想问一下BSA分析实操课程中qtlplot这个命令是一个脚本吗 qtlplot -v qtlseq.RS-blast.clean.vcf.gz -r HitomeboreWT --bulk1ID S-bulk --bulk2ID R-bulk --N-bulk1 20 --N-bulk2 20 -m 0.3 --out qtlseq_SR-blast 1 回答
- 老师好,请问课程bsa分析中,mutmap能同时输入两个子代混池吗? 1 回答
- BSA数据在哪里下载呢? 1 回答
