NCBI高通量测序数据SRA数据库下载新方法
SRA数据下载方法
NCBI下载SRA数据
从NCBI下载数据本来是一件很简单的事情,但是今天碰到几个坑:
1、paper里没有提供SRA数据号、也没有提供路径;
2、不知道文件在ftp的地址,不能直接用wget下载
所以通过在NCBI官网,直接在SRA搜索栏里输入paper的title关键词NIFTY BGI

搜索结果:
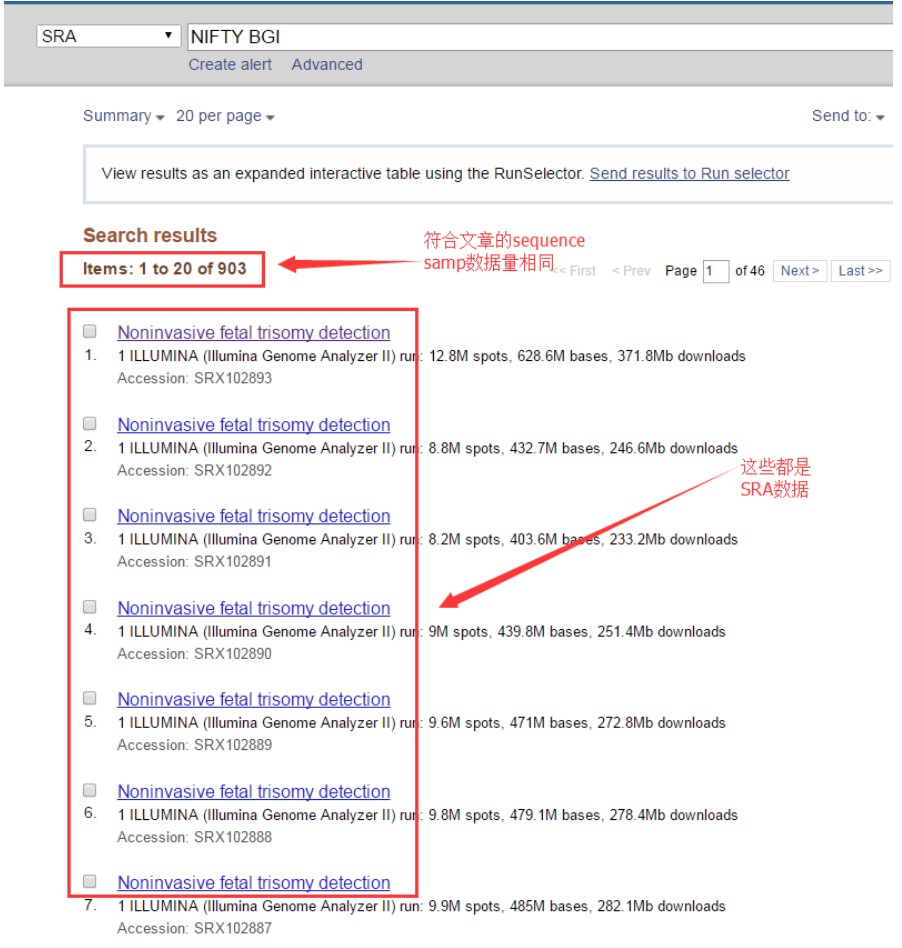
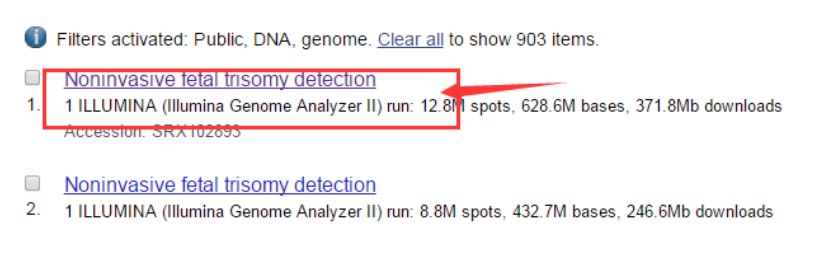
选一个文件点击进去
进去之后,再点击SRP
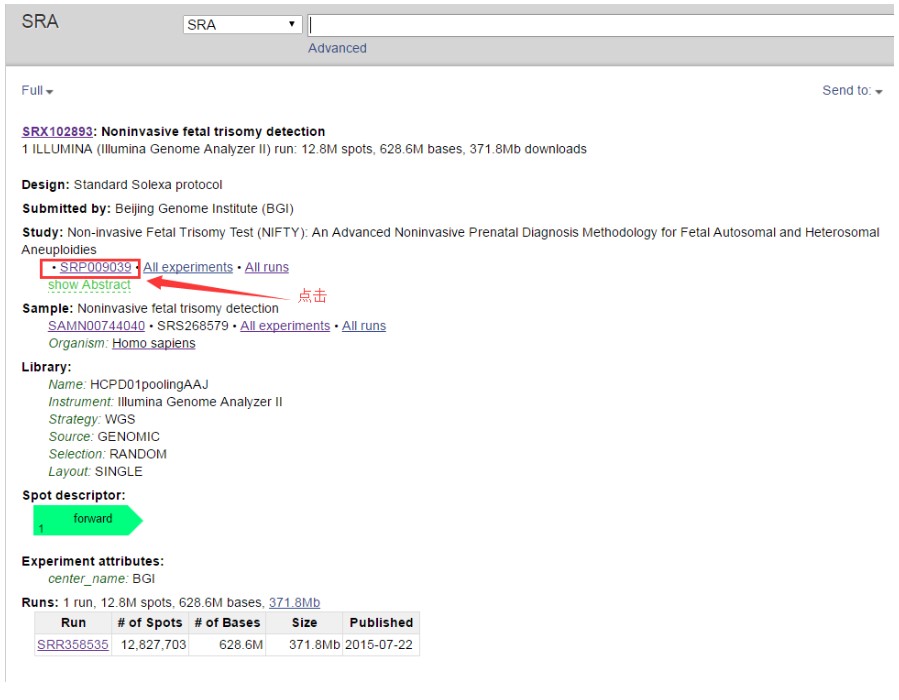
然后:
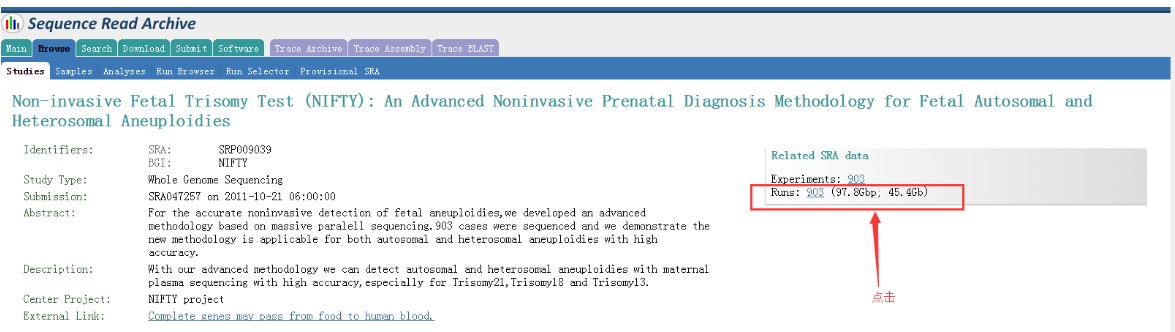
出现如下内容:
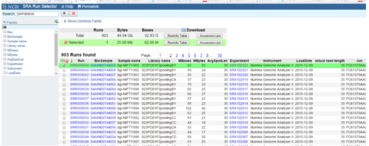
然后选择所有SRR文件:
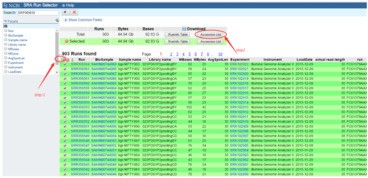
下载Accession list之后得到文件列表:
SRR354208 SRR357358 SRR357397 SRR357398 SRR357666 SRR357667 SRR357668 SRR357669 SRR357670 SRR357671 SRR357672 SRR357673 SRR357674 SRR357675 SRR357676 |
然后根据这个列表在linux下载:
[wuzengding@mn01 NIFTY_BGI_samp]$ cat /data1/Medicine/WZD/SRR_Acc_List.txt | while read line > do > echo $line > /home/wuzengding/biosoftware/sratoolkit.2.8.2-1-centos_linux64/bin/fastq-dump.2.8.2 ${line} > done |
注:另外一种更简单方法
在找到这个界面时
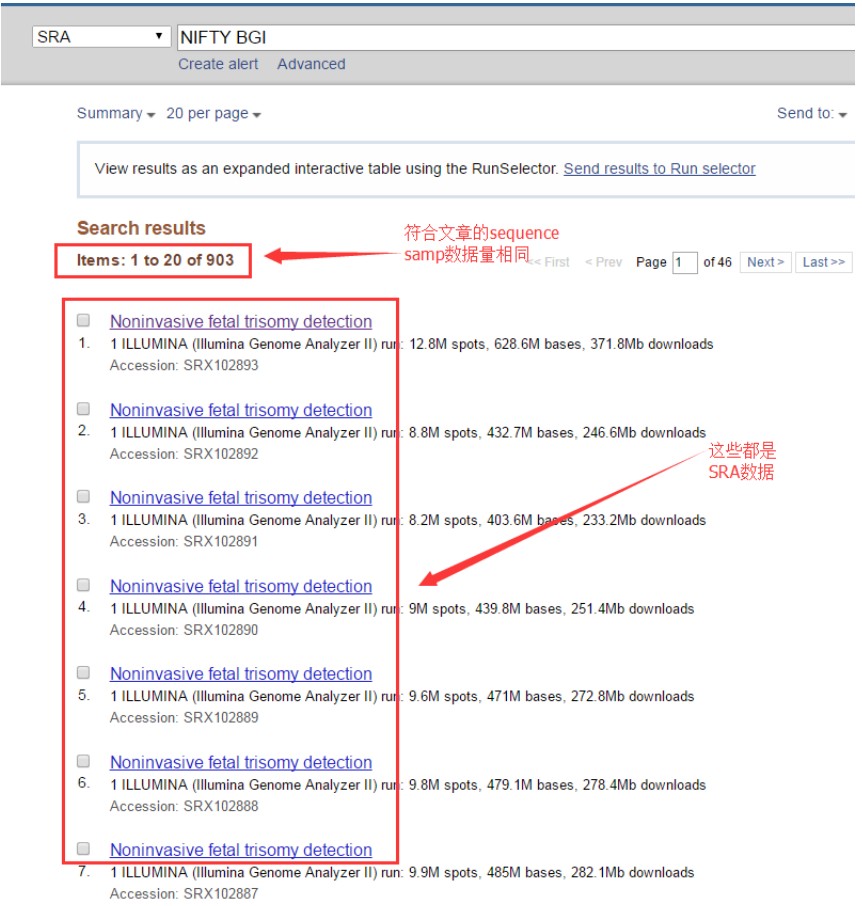
点击send to
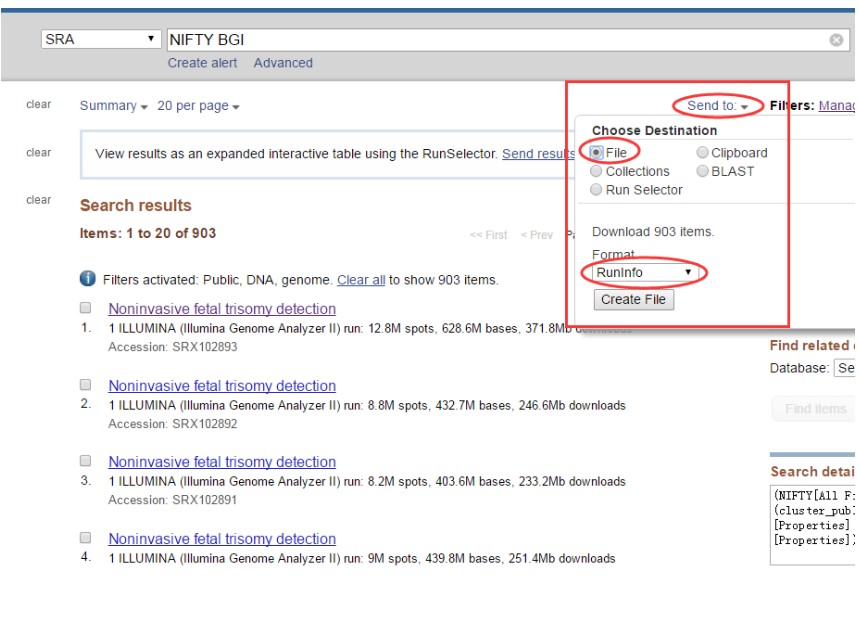
最后得到SraRunInfo.csv文件,文件内容是各个samp sequence的列表信息,包括FTP上的下载地址:
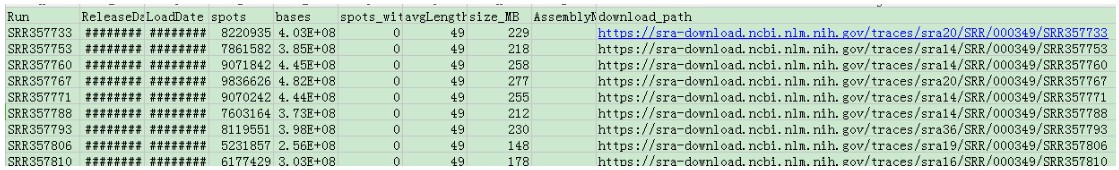
然后在linux中下载,
完毕!
此外,我们在网易云课堂上有各种教学视频,有兴趣可以了解一下:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘
6. 更多学习内容:linux、perl、R语言画图,更多免费课程请点击以下链接:
- 发表于 2019-07-12 11:39
- 阅读 ( 6302 )
- 分类:软件工具
你可能感兴趣的文章
- 探索基因大数据的利器——SRA Explorer 4252 浏览
- NCBI、ENA高通量测序数据下载-fastq数据 8869 浏览
- ascp 高速下载NCBI各种数据库中的数据(SRA NR NT 分类数据库,) 7013 浏览
- ENA数据库查询SRA数据 7251 浏览
- NCBI-SRA数据下载的3种方法 26146 浏览
- SRA工具sratoolkit把原始测序数据转为fastq格式 7895 浏览
相关问题
- SRA下载 1 回答
- SRA数据下载 2 回答
- 利用命令行进行sra数据下载有如下报错 1 回答
- sra文件转换为fastq文件 1 回答
- 怎么利用公开的转录组做基因家族的表达分析,还有GEO和SRA的区别是什么? 1 回答
- sratoolkit中prefetch下载一直报错是不是因为网的事情? 1 回答
0 条评论
请先 登录 后评论
